Leidi Herrera
María C. Ferreira
Leticia M. Rolón
Vanessa Ruiz
Nilsa González-Brítez
Artículo original
Diversidad genética de aedes aegypti en el eje transfronterizo Central-Alto Paraná en Paraguay
Genetic diversity of aedes aegypti in the central-upper Paraná Cross-Border axis in Paraguay
Sady C. Britez ![]() 1,2, Bióloga, máster en Ciencias Biomédicas
1,2, Bióloga, máster en Ciencias Biomédicas
Leidi Herrera ![]() 1,3, bióloga, PhD en Ciencias
1,3, bióloga, PhD en Ciencias
María C. Ferreira ![]() 1, bióloga, máster en Desarrollo Local Sostenible
1, bióloga, máster en Desarrollo Local Sostenible
Leticia M. Rolón ![]() 1,2, biotecnóloga
1,2, biotecnóloga
Vanessa Ruiz ![]() 1,2, biotecnóloga
1,2, biotecnóloga
Nilsa González-Brítez ![]() 1,2, bióloga, PhD en Parasitología
1,2, bióloga, PhD en Parasitología
1 Instituto de Investigaciones en Ciencias de la Salud, Universidad Nacional de Asunción, San Lorenzo, Paraguay.
2 Facultad de Ciencias Exactas y Naturales, Universidad Nacional de Asunción, San Lorenzo, Paraguay.
3 Instituto de Zoología y Ecología Tropical, Facultad de Ciencias, Universidad Central de
RESUMEN
Objetivos: Conocer la diversidad genética de Aedes aegypti en el corredor vial transfronterizo Central-Alto Paraná de Paraguay, con registros de casos de dengue. Materiales y métodos: Se seleccionaron veinte hembras adultas de la eclosión de huevos de Ae. aegypti procedentes de casas geolocalizadas en los departamentos de Alto Paraná, Caaguazú, Cordillera y Central, entre el 2018 y 2019. Se extrajo ADN del tejido de las hembras para amplificación aleatoria de sus patrones polimórficos mediante amplificación aleatoria del ADN polimórfico por PCR (RAPD-PCR), usando cebadores H3 y B03 a fin de conocer parámetros genéticos de diversidad poblacional. Las relaciones entre las poblaciones de mosquitos según la localidad fueron visualizadas mediante análisis no apareado de la media aritmética. Las áreas idóneas de distribución geográfica real y potencial de estas poblaciones de Ae. aegypti fueron analizadas mediante DIVA-GIS 7.3.0 y MAXENT. Resultados: Se identificaron 40 loci mediante perfiles RAPD-PCR, con diferenciación génica moderada (Gst = 0,12). El corredor transfronterizo presentó condiciones bioclimáticas para la presencia de poblaciones variantes de Ae. aegypti, siendo determinantes en la distribución la precipitación del trimestre más cálido y la temperatura media del trimestre más seco. Conclusiones: Se evidencia que existe diversidad genética moderada en las poblaciones de Ae. aegypti procedentes de zonas con registros de casos de dengue ubicadas en el corredor vial transfronterizo que une los departamentos Central y Alto Paraná de Paraguay. El estudio de variabilidad genética de Ae. aegypti es de gran utilidad para la vigilancia entomoepidemiológica y evaluación de posibles eventos de resistencia al control químico.
Palabras clave: Aedes; Polimorfismo Genético; Técnica del ADN Polimorfo Amplificado Aleatorio; Mosquitos Vectores; Vigilancia; Paraguay (fuente: DeCS BIREME).
ABSTRACT
Objective: To determine the genetic diversity of Aedes aegypti in the Central-Alto Paraná cross-border road corridor of Paraguay, an area that has reports of dengue cases.
Materials and methods: Twenty adult females were selected from hatching Ae. aegypti eggs from households geolocated in the departments of Alto Paraná, Caaguazú, Cordillera and Central, between 2018 and 2019. DNA was extracted from the tissue of females for amplifying their polymorphic patterns by random amplification of polymorphic DNA by PCR (RAPD-PCR), using primers H3 and B03 in order to identify genetic parameters of population diversity. The relationships between mosquito populations according to locality were observed by unpaired arithmetic mean analysis. We used DIVA-GIS 7.3.0 and MAXENT to analyze the suitable areas of actual and potential geographic distribution of these Ae. aegypti populations. Results: Forty loci were identified by RAPD-PCR profiling, with moderate gene differentiation (Gst = 0.12). The cross-border corridor presented bioclimatic conditions for the presence of variant populations of Ae. aegypti, with precipitation in the warmest quarter and mean temperature in the driest quarter being determinant in the distribution. Conclusions: There is evidence of moderate genetic diversity in Ae. aegypti populations from areas that have reported dengue cases in the cross-border road corridor linking the Central and Alto Paraná departments of Paraguay. The study of genetic variability of Ae. aegypti is very useful for entomo-epidemiological surveillance and evaluation of possible resistance to chemical control.
Keywords: Aedes; Genetic Polymorphism; Random Amplified Polymorphic DNA Technique; Mosquito Vectors; Surveillance; Paraguay (source: MeSH NLM).
INTRODUCCIÓN
Aedes aegypti (Linnaeus, 1762) (Diptera: Culicidae) es el vector transmisor de varias arbovirosis y es el responsable de la circulación de varios serotipos del virus dengue en simpatría con virus chikunguña y zika en las Américas (1). En la actualidad estas virosis, y en particular el dengue, han tenido gran impacto en la salud pública y en la pérdida de horas laborales. Esta pérdida se traduce en 70% de costos por hospitalización y costos por tratamiento y subsidios que pueden alcanzar hasta 80% del costo total (1). En el último trimestre de 2020, fueron registrados 60 925 casos de dengue en Paraguay, mayormente en el distrito capital (Asunción) y el departamento Central (2).
Las hembras de Ae. aegypti, que ejercen la hematofagia para la oviposición, presentan hábitos antropofílicoscrecientes, asociando sus altas densidades poblacionales a la presencia de depósitos de agua en recipientes artificiales o naturales como axilas de hojas, entrenudos de bambú y otras plantas fitotelmatas (acumulan agua en sus estructuras), que pueden estar presentes en el domicilio o el peridomicilio (3).
La variabilidad poblacional de Ae. aegypti es susceptible a cambios climáticos, movilidad de grupos humanos y selección por insecticidas, el conocimiento de estos factores contribuye a la comprensión de su dinámica poblacional (4-6).
Una alternativa para el estudio de análisis genético es el uso de la técnica molecular denominada amplificación aleatoria del ADN polimórfico por PCR o Random Amplified Polymorphic DNA (RAPD), de utilidad para la búsqueda de secuencias repetitivas aleatorias en regiones genómicas de alta variabilidad genética, dentro y entre poblaciones, con cantidad mínima de ADN y sin una secuencia diana previa (7). El estudio de poblaciones naturales de mosquitos permite determinar variaciones genéticas existentes en diferentes áreas geográficas y aporta información acerca de eventos de flujo génico en poblaciones separadas geográficamente, proporcionando datos para los programas de vigilancia y control en áreas de alta dispersión humana y elevado comercio a fin de implementar medidas específicas de vigilancia.
En el presente trabajo se propuso estudiar la diversidad genética de Ae. aegypti, en poblacionesde cuatro departamentos del eje transfronterizo constituido entre los departamentos de Central, Cordillera, Caaguazú y Alto Paraná, de Paraguay. Los vectores fueron capturados entre los años 2018 y 2019, en localidades con registros de casos de dengue.
|
MENSAJES CLAVE |
|
Motivación para realizar el estudio: conocer la biología del mosquito Ae. aegypti, vector de agentes de arbovirosis en países endémicos, tiene importancia si se estudia su diversidad genética con relación a áreas bioclimáticamente definidas. Principales hallazgos: los resultados mostraron diferenciación genética moderada y migración de genes que probablemente han mantenido a las poblaciones de mosquitos con similaridad genética. El eje transfronterizo Central-Alto Paraná mostró condiciones bioclimáticas favorables modeladas por DIVA-GIS y MAXENT para Ae. aegypti.. Implicancias: los corredores transfronterizos deben tener vigilancia entomoepidemiológica por ser vías de tránsito y de comercio, que favorecen la dispersión del vector en el Paraguay y países limítrofes. |
MATERIALES Y MÉTODOS
Área de estudio
Se llevó a cabo un estudio descriptivo con un muestreo por conveniencia. Se incluyeron ejemplares de Ae. aegypti de los departamentos Central (ciudad de San Lorenzo), Cordillera (ciudad de Piribebuy), Caaguazú (ciudad de Coronel Oviedo), Alto Paraná (ciudad de Hernandarias), todas son zonas colindantes y bajo la influencia del eje transfronterizo (ruta 2 y ruta 7) entre Gran Asunción (Central) y Ciudad del Este (Alto Paraná). Este corredor aproxima las áreas limítrofes de Argentina con Gran Asunción y llega hasta Brasil a través del Dpto. de Alto Paraná, que se caracteriza por ser de alta movilidad humana, transporte, migración y comercio, con algunos registros de brotes de dengue autóctono (8) (Tabla 1).
Tabla 1. Localización geoclimática, sociodemográfica y epidemiológica de las áreas del eje transfronterizo Central-Alto Paraná, Paraguay, para colecta de Aedes aegypti de áreas endémicas de dengue
|
Localidades |
Coordenadas geográficas |
Clima |
Población |
Actividad comercial |
Casos de dengue |
|
Hernandarias |
25°22′00″ S |
T= 23,5 °C |
79 735 |
Industria y servicios |
10 |
|
Coronel Oviedo |
25°26′53″S |
T=22,9 °C |
121 626
|
Ganadería y agricultura |
34 |
|
Piribebuy |
25°28′45″S
|
T=22 °C; |
28 179 |
Turismo y artesanía |
117 |
|
San Lorenzo |
25°20′35″S |
T=22,9 °C |
258 919 |
Comercio e industria |
1223 |
T: temperatura anual promedio. HR: humedad Relativa. VT: variación anual de la temperatura. pp: precipitación media/mes.
Fuente: el clima fue tomado de: https://es.weatherspark.com y los casos de dengue del Servicio Nacional de Erradicación del Paludismo (SENEPA) de Paraguay.
Material biológico
Los huevos de Ae. aegypti fueron colectados entre mayo del 2018 a mayo del 2019; a través de la colocación de dos a tres ovitrampas en áreas peridomiciliares de viviendas con características ambientales favorables para presencia del vector, tales como áreas húmedas y sombreadas, vegetación circundante, etc. Una única colecta se realizó en 80 viviendas seleccionadas a partir de un punto escogido al azar en cada localidad, con el fin de obtener agrupaciones naturales de individuos dentro de cada población (9). Dicha colecta permitió obtener aproximadamente 1500 a 2000 huevos por localidad. Las ovitrampas fueron retiradas a los siete días posteriores a la instalación, y transportadas al laboratorio, hasta la generación de mosquitos adultos, que fueron mantenidos por 24 h a 78% de humedad relativa, 28 °C de temperatura y alimentación con solución azucarada. La clasificación taxonómica de la especie se basó en su morfología y en claves taxonómicas (10,11).
Como control se incorporaron individuos de la cepa de referencia Rockefeller de Ae. aegypti (altamente endogámica y con más de 25 años de cría en laboratorio) mantenidos bajo las mismas condiciones.
Extracción del ADN
Se usaron veinte individuos hembras de cada población, colocándose cada uno en un tubo de microcentrífuga al cual se le adicionó 240 µL de resina Chelex® al 5% y 40 µL de solución salina NaCl 0,1M para su posterior maceración. El contenido permaneció en incubadora a 99 ⁰C por 10 min para luego ser centrifugado por 15 min. Se tomaron 200 µL del sobrenadante y se preservaron a -20 °C hasta su uso.
Amplificación por RAPD-PCR
La reacción de amplificación se formuló utilizando un volumen final de reacción de 20 µl, el cual contenía 0,2 mM de dNTPs, 1,2 mM de MgCl2, 1U de buffer 1X; 4,0 pmol de los cebadores de inserción aleatoria H3 (5´CATCCCCCTG´3) y B03 (5´CATCCCCCTG´3) Macrogene® (la reacción de PCR fue desarrollada con ocho oligonucleótidos comerciales: OP01, OP02, OP13, OP14, OP16, A2, H3, B03; se seleccionaron los cebadores con base a su mejor reproducibilidad y presencia de productos polimórficos), 1,0 U de enzima ADN Taq Polimerasa platinum termoestable (Taq DNA Polymerase, Invitrogen®) y 17,5 ng/µL de ADN molde. El procedimiento de amplificación se realizó utilizando el termociclador PTC-100 (MJ Research®, MA, USA) bajo las siguientes condiciones: desnaturalización inicial a 94 °C por 2 min, seguido de 45 ciclos con: desnaturalización a 93 °C por 1 min, hibridación a 35 °C por 1 min, extensión a 72 °C por 2 min y una extensión final a 72 °C por 5 min (6).
Los productos de amplificación fueron separados por electroforesis en gel de agarosa al 2% con buffer TAE 1X, corrida a 80 V por 1 h 45 min. El gel fue teñido con bromuro de etidio para visualización de las bandas e identificación de su tamaño, las cuales fueron contrastadas con marcadores moleculares de 100 pb. (DNA Ladder de Promega®) y 200 pb (Hyperladder I Bioline®). La fotodocumentación fue realizada a través de un transluminador UV, modelo Vilber E-box.
Análisis de datos
Los marcadores RAPD-PCR se analizaron bajo la premisa de que se segregaron siguiendo las proporciones mendelianas y que las frecuencias génicas obtenidas estuvieron en proporciones de Hardy-Weinberg (12). Las frecuencias alélicas fueron estimadas bajo el criterio de presencia de bandas de un alelo, asumiendo que cada banda es el producto de un alelo dominante en un locus homocigoto o heterocigoto, donde la ausencia de banda representa el genotipo homocigoto recesivo. La puntuación de las mismas generó como resultado los porcentajes de loci polimórficos (13).
Se determinaron los índices de diversidad genotípica (Shanon); la diversidad total de genes (Ht); la diversidad de genes de individuos en relación a su población especifica (Hs); el coeficiente de diferenciación génica (estimador Gst) como indicador de estructuración genética entre subpoblaciones y la tasa de migración efectiva o flujo de genes (Nm) (14), usando el programa de genética poblacional POPGENE (versión 1.3.2). Se construyó un dendrograma de relación genética intrapoblacional, utilizando el algoritmo de análisis de la media de grupos de pares no ponderados (UPGMA) (15).
La distribución geográfica real y potencial de las áreas idóneas de incidencia de variantes RAPD-PCR de Ae. aegypti fue analizada mediante software DIVA-GIS 7.3.0 y MAXENT. Se usaron los datos de presencia de Ae. aegypti geolocalizados mediante un dispositivo GPS-Garmin® serie GPSMAP 64sx y cargados al software Excel. Los datos fueron leídos en DIVA-GIS 7.3.0, para generación de mapa ráster de distribución real de las poblaciones en estudio, en función de las 19 variables bioclimáticas de Global Climate Data–WorldClim(http://www.wordclim.org), en formato ráster, con interpolación de datos tales como, rangos de temperatura diurna y anuales, isotermalidad, temperaturas promedio del trimestre más frio/caliente, más seco/húmedo del año, precipitación anual, precipitación del mes más húmedo/seco, precipitación del trimestre más seco/frío o más húmedo/seco compatibles con la geografía del Paraguay (resolución de 1 km2 en el Ecuador), predictores para 50 años (1950-2000). El software MAXENT permitió asociar los puntos georreferenciados (formato ASCII) y las variables bioclimáticas descritas (formato ASCII) en corridas de 10 modelos y 1000 iteraciones (16). Esta información se contrastó con datos bioecológicos asociados a temperatura y humedad para Ae. aegypti.
Consideraciones éticas
El trabajo fue aprobado por el Comité de Ética del Instituto de Investigaciones de la Universidad Nacional de Asunción, cuyo código de aprobación fue P36/2015. La colocación de trampas se realizó con previo consentimiento de los propietarios, las muestras de insectos utilizadas fueron las mínimas necesarias para lograr resultados óptimos y fueron manipuladas bajo criterios de bioseguridad. Se respetaron los derechos de autores y las fuentes consultadas.
RESULTADOS
La colecta permitió obtener 1500 a 2000 huevos por localidad, de los cuales se obtuvieron los adultos que proporcionaron el material genético. La técnica de RAPD reveló la amplificación de 40 bandas (entre 300 y 1700 pb), con los cebadores H3 y B03, de las cuales cuatro correspondieron a fragmentos conservados de entre 700 a 1200 pb para el cebador H3 y de 450 a 1200 pb, para el cebador B03, presentes en todos los individuos de las poblaciones en estudio (Figura 1).
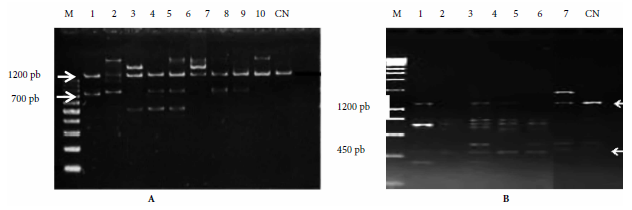
Figura 1. Patrones de amplificación de bandas por amplificación aleatoria del ADN polimórfico por PCR (RAPD-PCR) para el estudio de variabilidad en poblaciones naturales de Aedes aegypti del eje transfronterizo Central-Alto Paraná en Paraguay.
A) Perfiles de bandas amplificadas con el cebador H3. Las flechas blancas indican los perfiles de bandas comunes correspondientes a 700 y 1200 pb aproximadamente. (Carriles: 1-2 Hernandarias, 3-4 Caaguazú, 5-6 Piribebuy, 7-8 San Lorenzo, 9-10, Rockefeller [cepa patrón]), CN: control negativo, M: marcador de peso molecular/100 pb DNA Ladder de Promega®.
B) Perfiles de bandas amplificadas con el cebador B03. Las flechas blancas indican los perfiles de bandas comunes correspondientes a 450 y 1200 pb aproximadamente. (Carriles: 1-2 Piribebuy, 3-4 Hernandarias, 5-7 Caaguazú), CN: control negativo, M: marcador de peso molecular/200pb Hyperladder I Bioline®.
El análisis de la matriz de presencia y ausencia de bandas permitió estimar los valores medios de Ht (diversidad total de genes) y Hs (diversidad de genes relativa a la población), con los que se atribuyó mayor diversidad genética a la encontrada entre las poblaciones (interpoblacional) y no dentro de las poblaciones. El valor medio obtenido para el índice Gst=0,1231 sugirió diferenciación genética moderada (18) y el valor medio de Nm infirió migración efectiva de genes (Tabla 2), en concordancia al promedio encontrado de 15 loci polimórficos en todas las poblaciones estudiadas. Las poblaciones de Hernandarias y San Lorenzo presentaron el mismo porcentaje de polimorfismo intraespecífico (58,3%), en tanto las poblaciones de Caaguazú y la cepa de referencia Rockefeller presentaron un porcentaje de polimorfismo intraespecífico de 62,5%. El mayor porcentaje lo presentó la población de mosquitos de Piribebuy (66,6%) (Tabla 3).
Tabla 2. Diversidad genética (Nei, 1973) intrapoblaciones de Aedes aegypti de cuatro departamentos del eje transfronterizo Central-Alto Paraná, analizadas por RAPD-PCR (amplificación aleatoria del ADN polimórfico por PCR) con independencia de la región de estudio
|
índices |
Cebador H3 |
Cebador B03 |
Valores medio de los índices |
|
I |
0,3998 |
0,5173 |
0,4586 |
|
Ht |
0,2457 |
0,3344 |
0,2900 |
|
Hs |
0,2193 |
0,2879 |
0,2536 |
|
Gst |
0,1073 |
0,1389 |
0,1231 |
|
Nm |
4,1608 (%) |
3,0990 (%) |
3,6299 (%) |
I: índice de Shanon. Ht: diversidad total de genes. Hs: diversidad de genes relativa a la población. Gst: estimador de estructura génica poblacional. Nm: flujo de genes.
Los resultados provienen de la matriz interpoblacional anidada.
Tabla 3. Diversidad genética de Nei (1973) entre poblaciones de Aedes aegypti de cuatro departamentos del eje transfronterizo Central-Alto Paraná analizadas por RAPD-PCR (amplificación aleatoria del ADN polimórfico por PCR con independencia del marcador genético
|
Estimadores de diversidad genética Nei (1973) |
||||
|
Localidades |
I (Shanon) |
h (Nei) |
Loci polimórficos (%) |
Numero de loci polimórficos |
|
Hernandarias |
0,3135 |
0,2067 |
58,33 |
14 |
|
Caaguazú |
0,3559 |
0,2400 |
62,50 |
15 |
|
Piribebuy |
0,4055 |
0,2800 |
66,67 |
16 |
|
San Lorenzo |
0,3494 |
0,2400 |
58,33 |
14 |
I: índice de Shanon. h: diversidad genética de Nei, 1973.
Los resultados provienen de una matriz interpoblacional todo contra todo sin discriminar marcador molecular utilizado.
Las relaciones entre las poblaciones de mosquitos según la localidad de procedencia incluyendo a la cepa patrón Rockefeller, revelaron la existencia de dos grupos (clúster), con independencia de las ecorregiones; un grupo entre las poblaciones de Ae. aegypti provenientes de Hernandarias, Piribebuy y otro con las poblaciones de San Lorenzo, Caaguazú y Cepa de referencia Rockefeller. En el punto terminal del clúster, las poblaciones de Caaguazú se segregaron del resto de las poblaciones de otras regiones del eje y las poblaciones de San Lorenzo se segregaron con las de la cepa de referencia (Figura 2).
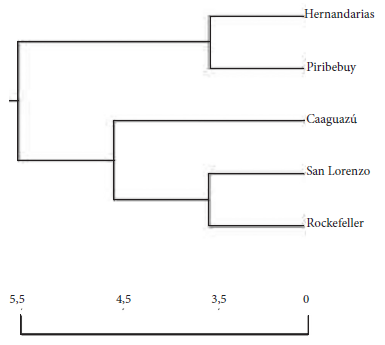
Figura 2. Dendrograma basado en distancia genética de Nei usando el método de análisis no apareado de la media aritmética (UPGMA), Nei, 1978, construido mediante análisis de frecuencias de bandas por amplificación aleatoria del ADN polimórfico por PCR (RAPD-PCR), para las poblaciones de Ae. aegypti procedentes de cuatro localidades de la Región Oriental del Paraguay y la cepa Rockefeller de referencia (software Pop Gene).
El modelo de distribución espacial real y potencial de Ae. aegypti en función de 19 variables bioclimáticas (DIVA-GIS y MAXENT) confirmó la existencia de áreas idóneas para el establecimiento del vector en un 70% de las regiones del eje transfronterizo. El mapa muestra el corredor entre las ecorregiones del Chaco Húmedo (San Lorenzo-Central y Piribebuy- Cordillera) y el Bosque Atlántico (Coronel Oviedo- Caaguazú y Hernandarias-Alto Paraná). La escala de grises (0 a 1), indica la probabilidad de presencia de la especie (Figura 3).
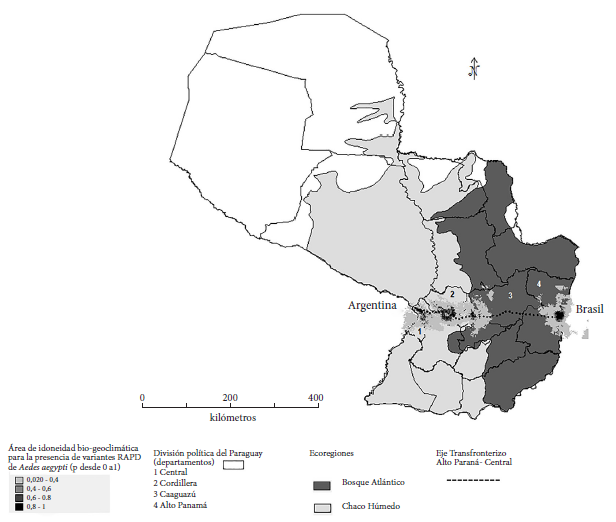
Figura 3. Áreas de idoneidad para la distribución espacial real y potencial de poblaciones de Ae. aegypti en el eje transfronterizo Central-Alto Paraná de Paraguay en función de variables bioclimáticas (DIVA-GIS y MAXENT; umbral de corte del percentil 10). En color gris claro; la ecorregión del Chaco Húmedo (Central y Cordillera) y en gris oscuro; el Bosque Atlántico (Caaguazú y Alto Paraná).
El modelo MAXENT reveló que las variables bioclimáticas idóneas para la presencia de Ae. aegypti, se ponderaron en forma contributiva en 33,3% para la precipitación del trimestre más cálido (media de 184 mm/mes), 13,2% para la precipitación del mes más húmedo (media de 75 mm/mes), y de 13% para la temperatura promedio del trimestre más seco (media de 26°C). Los datos fueron tomados del Servicio de Información Meteorológica OGIMET (https://www.ogimet.com).
La estacionalidad de la temperatura y la isotermalidad se sucedieron en importancia (7,4% aproximadamente), para la presencia de la especie. Es decir que, en las zonas de muestreo, la estacionalidad de la temperatura resultó variante en función de las ecorregiones, siendo 6 °C por debajo de la máxima temperatura para el Chaco Húmedo y entre 4 y 5 °C por debajo de la máxima temperatura para el Bosque Atlántico (OGIMET). Por otro lado, el modelo DIVA-GIS reveló la presencia de puntos geográficos polares o «zonas calientes de presencia» (color negro) en los extremos del eje transfronterizo, siendo estos Asunción (próximo a la frontera con Argentina) y Alto Paraná (próximo a la frontera con Brasil) (Figura 3).
DISCUSIÓN
Los resultados mostraron que las poblaciones naturales de Ae. aegypti colectadas en el eje transfronterizo Central-Alto Paraná en Paraguay, presentaron índices de diversidad genética moderada, lo que parece indicar que, a lo largo del eje transfronterizo, la población estaría estabilizándose genéticamente con un flujo de genes mayor a 1 y ponderación media del efecto de migraciones entre localidades.
La diversidad genética está condicionada entre otros factores por los efectos de cambios ambientales naturales o por la inclusión humana sobre la dinámica poblacional de organismos (19) y permite evaluar las condiciones de los vectores de enfermedades, especies invasoras o la existencia de variedades mejoradas y modificadas genéticamente, así como el flujo génico intra e interpoblacional de los agentes que tienen efectos en salud pública (20).
Estos resultados son similares a los índices de diversidad obtenidos por RAPD-PCR en seis estados de Brasil para Ae. aegypti , en el cual con 27 loci polimórficos el índice Ht fue 0,390 (21) y con 60 loci polimórficos, el índice Ht fue 0,388 (22) , resultados análogos fueron referidos en México con 60 loci polimórficos, donde el índice Ht=0,34 (12) y en Puerto Rico con 57 loci polimórficos este índice fue Ht= 0,35 (23). Estos valores revelaron índices de heterogeneidad moderada entre las poblaciones estudiadas, que acorde a los criterios de clasificación (18). Las poblaciones de estudio mostraron diferenciación moderada (Gst=0,107 para el cebador H3, y Gst=0,138 para el cebador B03) no dependiente de la región geográfica. Las distancias cortas de vuelo del insecto e índices Gst muy reducidos sugieren una dispersión pasiva o forzada entre localidades colindantes, favorecida por el transporte terrestre y modulada por la actividad comercial humana, así como por el transporte accidental de huevos en depósitos artificiales; en tal sentido algunos investigadores demostraron que existe una correlación significativa entre el flujo de genes en Ae. aegypti y el transporte pasivo humano (24). Este tipo de migración adicionalmente explicaría la reducida variación entre poblaciones distantes, las cuales probablemente no han tenido suficiente tiempo evolutivo para divergir, y su similitud resulta de la inercia de la frecuencia de los genes a lo largo del tiempo (21,25,26).
En cuanto al clúster de segregación de las poblaciones, construido en base a distancia genética de Nei, se observaron dos grupos de similaridad, cada uno con poblaciones de ambas ecorregiones; congruentes con la diferenciación genética moderada, siendo la población de Caaguazú, la más divergente de las demás poblaciones. La similitud de las poblaciones de mosquitos Ae. aegypti de San Lorenzo con la cepa de referencia Rockefeller, la cual es altamente endogámica y adaptada al laboratorio, asemeja la selección artificial de laboratorio (cepa Rockefeller) con la franca domiciliación de Ae. aegypti silvestre en las regiones altamente antropizadas (San Lorenzo), en tal sentido, en ambas condiciones se da un crecimiento óptimo, disponibilidad y frecuencia de ingesta sanguínea, confiriendo «docilidad» al comportamiento en espacio-tiempo que intervienen en los procesos de selección (27).
El modelo de distribución espacial real y potencial de Ae. aegypti en función de 19 variables bioclimáticas (DIVA-GIS y MAXENT) confirmó la existencia de áreas idóneas para el establecimiento del vector en un 70% de las regiones del eje transfronterizo (Argentina-Paraguay-Brasil), siendo las variables determinantes en orden de importancia, la precipitación del trimestre más cálido, la precipitación del mes más húmedo, temperatura promedio del trimestre más seco, estacionalidad de la temperatura y la isotermalidad.
Las áreas periurbanas cercanas a Coronel Oviedo (Caaguazú), parecieran no mostrar idoneidad para el establecimiento del vector, probablemente influenciadas por las grandes distancias entre áreas urbanizadas y rutas de acceso, la baja densidad poblacional y la alta movilidad de grupos familiares amerindios presentes en la zona, lo cual dificultaría la instalación perenne del vector, aun cuando las condiciones climáticas favorezcan su presencia. En otros países se ha reportado la distribución de Ae. aegypti en comunidades indígenas, en las cuales hay zonas endémicas de transmisión del virus y emergencias sanitarias de infección por dengue, sin embargo en Paraguay, son muy escasos los registros de dengue en poblaciones originarias (28).
Como limitación de este estudio se debe mencionar que el marcador RAPD-PCR requiere una rigurosa estandarización para reducir la variación provocada por los componentes de la reacción, y su condición de dominancia implica estimaciones de diversidad genética de manera indirecta. Sin embargo, su metodología sencilla, rápida y menos costosa permite detectar polimorfismos sin secuenciar o caracterizar el genoma de interés analizando varios loci a la vez.
El presente trabajo es el primero que aborda un estudio genético con poblaciones naturales de Ae. aegypti en Paraguay, que permitió aproximar lo que ocurriría en un eje vial de gran importancia comercial (conexión terrestre de Asunción, capital del Paraguay y la frontera con Brasil, Alto Paraná), en donde surge el establecimiento de Ae. aegypti, cuya dinámica debe ser vigilada, por ser estas regiones de alta actividad antrópica con áreas turísticas y comerciales que establecen puntos de entrada y salida entre países, favoreciendo así la diseminación del agente transmisor, en función de la aparición de casos y en cuanto a la selección de subpoblaciones resistentes al control químico.
Este estudio evidenció que existe diversidad genética moderada en las poblaciones de Ae. aegypti procedentes de zonas con registros de dengue ubicadas en el corredor vial transfronterizo que unen los departamentos Central y Alto Paraná de Paraguay. La diversidad interpoblacional fue mayor que la diversidad intrapoblacional, lo que nos lleva a suponer que existen migraciones frecuentes pero moderadas del vector, condicionados por factores bioclimáticos idóneos que contribuyen con dicha diferenciación. Como recomendación se considera necesario incluir otros marcadores, tales como los genes mitocondriales que permitirían continuar con la asignación de haplotipos a través de pruebas de referencia como la secuenciación del genoma del Ae. aegypti en Paraguay, con el objetivo de conocer mejor su biodiversidad y/o rutas de migración local que permitan estratificar áreas de riesgo para definir mejores estrategias de control vectorial y métodos que aseguren adecuada vigilancia entomológica.
Contribuciones de los autores: SB, NGB y LH concibieron la idea y el diseño del artículo. SB, MF, VR realizaron la colecta en campo y manutención de crías de mosquitos en laboratorio. SB, LR, NGB realizaron el diseño y consecución de las pruebas moleculares. LH aplicó el diseño de modelado de distribución y predicción de nichos. SB, NGB y LH realizaron el análisis de los datos y la discusión. NGB y LH revisaron el manuscrito. Todos los autores aprobaron la versión final.
Conflicto de intereses: durante la ejecución del trabajo o la redacción del manuscrito no han incidido intereses que sean diferentes a la investigación. Por tanto, los autores declaran que no existe ningún conflicto de interés para la publicación del presente manuscrito.
Financiamiento: este trabajo fue financiado por los proyectos PINV15-777 y PINV 15-704 del Programa PROCIENCIA del Consejo Nacional de Ciencia y Tecnología (CONACYT) de Paraguay.
Referencias bibliográficas
1. Laserna A, Barahona-Correa J, Baquero L, Castañeda-Cardona C, Rosselli D. Economic impact of dengue fever in Latin America and the Caribbean: a systematic review. Rev Panam Salud Pública. 2018;42:e111. doi: 10.26633/RPSP.2018.111.
2. Ministerio de Salud Pública. Boletín Epidemiológico. Semana 1-35 [Internet]. Asunción; 2020 [citado el 8 de noviembte del 2021]. p. 1–29. Disponible en: https://dgvs.mspbs.gov.py/files/boletines/SE35_2020_Boletin.pdf Asunción; 2020.
3. Navarro JC. Fauna de mosquitos (Diptera : Culicidae) del Parque Nacional Cerro El Copey y nuevos registros para La Isla de Margarita. Bol Entomol Venez. 1998; 13(2):187-94.
4. Leiva N, Cáceres O. Variabilidad genética de Aedes aegypti en algunas áreas del Perú usando Single Stranded Conformational Polymorphism (SSCP). Rev Peru Med Exp Salud Publica [Internet]. 2004;21(3):157–66.
5. Bracco JE, Capurro ML, Lourenço-de-Oliveira R, Sallum MAM. Genetic variability of Aedes aegypti in the Americas using a mitochondrial gene: Evidence of multiple introductions. Mem Inst Oswaldo Cruz. 2007;102(5):573-80.
6. Hiragi C, Simões K, Martins E, Queiroz P, Lima L, Monnerat R. Variabilidade Genética em Populações de Aedes aegypti (L.) (Diptera: Culicidae) Utilizando Marcadores de RAPD. Neotrop Entomol. 2009;38(4): 542-7. doi :10.1590/S1519-566X2009000400018.
7. von Eggeling F, Spielvogel H. Applications of random PCR. Cell Mol Biol (Noisy-le Grand,France).1995;41(5):653-70.
8. Ministerio de Salud Pública. Boletín Epidemiológico. Semana 52 [Internet]. Asunción 2018 [citado el 3 de diciembre del 2021]. p 1-37. Disponible en: https://dgvs.mspbs.gov.py/webdgvs/files/boletines/17_01_2018_11_49_39_Boletin-Epidemiologico_SE-52.pdf.
9. Santos S, Santos-Melo M. Field Evaluation of ovitraps consociated with grass infusión and Bacilus thuringiencis var isrraelensis to determine oviposition rates of Aedes aegypti. Dengue Bulettin. 2003;27:156–62.
10. Darsie RF. Keys to mosquitoes of Costa Rica (Diptera, Culicidae). Columbia: International Center for Disease Control, University of South Carolina; 1993. 58 p.
11. Rueda L. Pictorial keys for the identification of mosquitoes (Diptera: culicidae) associated with dengue virus transmission. Zootaxa. 2004;589(1):1-60. doi: 10.11646/zootaxa.589.1.1.
12. Gorrochotegui-Escalante N, De Lourdes Munoz M, Fernandez-Salas I, Beaty BJ, Black IV WC. Genetic isolation by distance among Aedes aegypti populations along the northeastern coast of Mexico. Am J Trop Med Hyg. 2000;62(2). 200-9. doi: 10.4269/ajtmh.2000.62.200.
13. Zambrano AY, Demey JR, Fuenmayor F, Segovia V, Gutiérrez Z. Diversidad Genética de una Colección de Yuca a través de Marcadores Moleculares RAPDs. Agronomía Trop. 2003 53(2):155–74.
14. Shannon C. The mathematical theory of Communications. B Bell Syst Tech J. 1948; 27(3): 379–423. doi: 10.1002/j.1538-7305.1948.tb01338.x.
15. Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics. 978;89(3): 583-90. doi: 10.1093/genetics/89.3.583.
16. Manel S, Ceri Williams H, Ormerod SJ. Evaluating presence-absence models in ecology: The need to account for prevalence. J Appl Ecol. 2001;38(5):921-31. doi: 10.1046/j.1365-2664.2001.00647.x.
17. Plasencia-Vázquez AH, Escalona-Segura G, Esparza-Olguín LG. Modelación de la distribución geográfica potencial de dos especies de psitácidos neotropicales utilizando variables climáticas y topográficas. Acta Zoologica Mex. 2014;30(3): 471-90. doi: 10.21829/azm.2014.30372.
18. Hart D, Clark AG. Principles of Populations Genetics. 3th ed., editor. Sunderland, USA: Edit Sinauer Associates Inc; 1997.
19. Piñero D, Cabrera-Toledo D, Canteros CE, Casas A, Castañeda Sortibran A, Castillo A. La diversidad genética como instrumento para la conservación y el aprovechamiento de la biodiversidad: estudios en especies mexicanas. En: Capital natural de México, vol I : Conocimiento actual de la biodiversidad . Mexico: CONABIO; 2008. p. 437–94.
20. Atencia MC, Pérez M de J, Caldera SM, Jaramillo MC, Bejarano EE. Genetic variability of Aedes aegypti in the department of Sucre, Colombia, by analysis of the nucleotide sequence of the mitochondrial ND4 gene. Biomedica. 2018;38(2):38:267-76 doi: 10.7705/biomedica.v38i0.3728.
21. Ayres CFJ, Melo-Santos MAV, Solé-Cava AM, Furtado AF. Genetic differentiation of Aedes aegypti (Diptera: Culicidae), the major dengue vector in Brazil. J Med Entomol. 2003;40(4) 40(4):430-5. doi: 10.1603/0022-2585-40.4.430.
22. dos Santos Paduan K, Araújo JP, Ribolla PEM. Genetic variability in geographical populations of Aedes aegypti (Diptera, Culicidae) in Brazil elucidated by molecular markers. Genet Mol Biol. 2006;29(2): 391-5. doi: 10.1590/S1415-47572006000200030.
23. Apostol BL, Black IV WC, Reiter P, Miller BR. Population genetics with RAPD-PCR markers: The breeding structure of Aedes aegypti in Puerto Rico. Heredity .1996;76(4) :325-34. doi: 10.1038/hdy.1996.50.
24. Huber K, Loan L Le, Chantha N, Failloux AB. Human transportation influences Aedes aegypti gene flow in Southeast Asia. Acta Trop. 2004;90(1):23-9. doi: 10.1016/j.actatropica.2003.09.012.25.
25. Scarpassa VM, Cardoza TB, Cardoso RP. Population genetics and phylogeography of Aedes aegypti (Diptera: Culicidae) from Brazil. Am J Trop Med Hyg. 2008;78(6):895-903. doi: 10.4269/ajtmh.2008.78.895.
26. Yañez P, Mamani E, Valle J, Garcia M, León W, Villaseca P, el tal. Genetic Variability of Aedes aegypti Determined by Mitochondrial Gene ND4 Analysis in Eleven Endemic Areas. Rev Peru Med Exp Salud Publica. 2013;30(3):246–50. doi: 10.17843/rpmesp.2013.302.199.
27. Lindqvist C. Domestication effects on foraging behaviour- consequences for adaptability of chickens. Linköping University. Sweden; 2008. 37p.
28. Lorenzetti M, Lavazza VH. Relaciones interétnicas y cuidados de salud en comunidades mbya guaraní de Puerto Iguazú (Misiones, Argentina). Jangwa Pana. 2021;20(1); 34–51. doi: 10.21676/16574923.3885.
Citar como: Britez SC, Herrera L, Ferreira MC, Rolón LM, Ruiz V, González-Brítez N. Diversidad genética de Aedes aegypti en el eje transfronterizo Central-Alto Paraná en Paraguay. Rev Peru Med Exp Salud Publica. 2022;39(2). doi: https://doi.org/10.17843/rpmesp.2022.392.10709.
Correspondencia: Nilsa González-Brítez; gbritez.nilsa@gmail.com
Recibido: 27/01/2022
Aprobado: 08/06/2022
En Línea: 08/07/2022