
Artículo Original
Detección de Leishmania spp. en base al gen que codifica la proteína HSP20
Detection of Leishmania spp. based on the gene encoding HSP20
Ana M. Montalvo1,a, Jorge Fraga1,a, Omaira Rodríguez2,b, Orestes Blanco1,c, Alejandro Llanos-Cuentas3,a, Ana L. García4,a, Braulio M. Valencia3,d, Carlos Muskus5,a, Gert Van der Auwera6,a, José M. Requena7,a
1
Departamento de Parasitología, Instituto de Medicina Tropical Pedro Kourí. La
Habana, Cuba.
2
Laboratorio de referencia e investigación en enfermedades tropicales de sanidad
militar. Bogotá, Colombia.
3
Instituto de Medicina Tropical Alexander von Humboldt, Universidad Peruana
Cayetano Heredia. Lima, Perú.
4
Universidad de San Simón. Cochabamba, Bolivia.
5
Programa de Estudio y Control de Enfermedades Tropicales, Universidad de
Antioquia.
Medellín, Colombia.
6
Biomedical Sciences Department.
Institute of Tropical Medicine of Antwerp.
Amberes, Bélgica.
7
Centro de Biología Molecular Severo Ochoa. Madrid, España.
a
PhD ; b bacterióloga; c Médico especialista de II grado en
Dermatología; d MSc
El artículo contiene parte de los resultados de la Tesis: "Evaluación de la utilidad del gen que codifica la proteína de choque térmico de 20 kDa (HSP-20) para la detección de Leishmania spp. mediante la técnica de PCR", presentada por Omaira Rodríguez Angarita para aspirar al título de máster en Parasitología. Instituto de Medicina Tropical Pedro Kourí. La Habana, 2012.
RESUMEN
Objetivos.Explorar una nueva diana para el diagnóstico molecular de Leishmania. Materiales y métodos. Se evaluó la utilidad del gen que codifica la proteína de choque térmico de 20kDa (hsp20) para la detección de Leishmania por medio de la reacción en cadena de la polimerasa (PCR).Se normalizó la PCR y se determinaron los parámetros analíticos, así como la validez y seguridad diagnóstica y la concordancia con la PCR-18S. Se evaluó la PCR-hsp20 con ADN obtenido de un grupo de muestras clínicas de distinta procedencia. Resultados. Los parámetros analíticos resultaron adecuados. La sensibilidad obtenida fue de 86% y la especificidad del 100%, la concordancia con el método de referencia resultó buena (κ = 0,731), lo que apoya su posible uso para el diagnóstico. La posibilidad de identificación posterior de la especie mediante secuenciación del producto amplificado le confiere una ventaja adicional. Conclusiones. Se demuestra la utilidad de este gen como una nueva diana para la detección del género Leishmania. Debido a su potencial, se recomienda mejorar la sensibilidad del procedimiento y realizar su evaluación en diversas regiones endémicas.
Palabras clave: Leishmania; Diagnóstico; Proteínas del choque térmico HSP20 (fuente: DeCS BIREME).
ABSTRACT
Objectives. Explore a new target for molecular diagnosis of Leishmania. Materials and methods. We evaluated the utility of the gene that encodes the heat shock protein 20-kDa (Hsp20) for detecting Leishmania by polymerase chain reaction (PCR). PCR was normalized and analytical parameters were determined, as well as the validity and diagnostic accuracy, and concordance with the PCR - 18S. PCR-Hsp20 with DNA was obtained from a group of clinical samples from different sources. Results. The analytical parameters were adequate. The sensitivity obtained was 86% and the specificity was 100%. The concordance with the reference method was good (κ = 0.731), which supports its potential use for diagnosis. The possibility of subsequent identification of the species by sequencing the amplified product gives an additional advantage. Conclusions. The usefulness of this gene as a new target for the detection of Leishmania was demonstrated. Because of its potential, it is recommended to improve the sensitivity of the method and to evaluate it in different endemic regions.
Key words: Leishmania; Diagnosis; HSP20 heat-shock proteins (source: MeSH, NLM).
INTRODUCCIÓN
La leishmaniosis es una enfermedad causada por pro-tozoos parásitos del género Leishmania que se transmiten por la picadura de insectos de los géneros Lutzomyia (Nuevo Mundo) y Phlebotomus (Viejo Mundo). Aproximadamente, 30 especies o subespecies de estos géneros se consideran vectores comprobados, al cumplir criterios establecidos por Killick-Kendrick (1), los cuales han sido recientemente enriquecidos con el fin de demostrar la importancia biomédica del vector en un foco específico, donde se muestre el efecto directo del control sobre la transmisión (2).
Esta parasitosis se presenta con diversas formas clínicas, que abarcan desde úlceras que pueden curar espontáneamente o tardar más de un año en sanar (leishmaniosis cutánea localizada), lesiones destructivas en las mucosas de la oronasofaringe, (leishmaniosis mucocutánea) hasta la enfermedad visceral, que afecta órganos como hígado, bazo, médula ósea, y compromete la vida si no se trata a tiempo, así como numerosas presentaciones atípicas que se notifican con frecuencia (3).
La variedad en la presentación clínica y las posibilidades reales de los laboratorios que enfrentan la detección del agente etiológico, determinan la forma de diagnosticar la enfermedad, que se puede realizar por la identificación microscópica del parásito en las muestras adecuadas, el cultivo del material, mediante métodos serológicos y mole-culares, los que difieren en sensibilidad y especificidad (4).
La reacción en cadena de la polimerasa (PCR) es una técnica molecular de comprobada eficacia, sensible y específica para la detección de ácido desoxirribonucleico (ADN) parasitario. Sin embargo, muchos de los protocolos reportados responden a las necesidades locales de países o áreas geográficas, sin que exista un consenso sobre la factibilidad del uso de unas u otras dianas genéticas para la amplificación. De otra parte, es necesario apuntar que numerosas áreas endémicas poseen laboratorios de referencia o investigación con facilidades para la aplicación de la PCR con variados fines, lo que facilitaría contar con esos resultados en diferentes situaciones.
Actualmente, la PCR se considera un acercamiento que no requiere recursos muy cuantiosos ni gastos considerables como la PCR en tiempo real (5).
Entre las dianas genéticas utilizadas para la detección de Leishmania se encuentran distintos fragmentos del gen que codifica para el ADN ribosomal (ADNr), que presenta entre 10 y 100 copias. La PCR-ADNr (PCR-18S) amplifica un fragmento de 115pb y los valores de sensibilidad reportados son adecuados: 92,1% (sangre); 92,9% (médula ósea) (6). En un modelo oligocromatográfico de visualización de la PCR, los valores notificados en muestras de leishmaniosis cutánea son de 93,2% y para leishmaniosis mucocutánea de 91,7% (7); mientras en Perú se reportó 92% de positividad para raspados y 74% para aspirados (8).
Hace pocos años se notificó por primera vez la proteína HSP20 en Leishmania amazonensis, que se mantiene conservada en varias especies del parásito (9). Durante su ciclo de vida el parásito se somete a un cambio drástico, desde la temperatura ambiente en el insecto vector hasta las altas temperaturas que encuentra en su hospedero mamífero, cuya respuesta está mediada por las proteínas de choque térmico (en inglés heat shock proteins, HSP) que constituyen un mecanismo homeostático que protege a las células del efecto destructivo del calor u otras condiciones de estrés ambiental (10). Por estas razones, se considera que las HSP deben desempeñar un papel fundamental en la interacción hospedero-parásito.
Se demostró que la proteína HSP20 recombinante es reconocida por anticuerpos presentes en el suero de personas con leishmaniosis visceral, y de forma más eficiente en el suero de perros con leishmaniosis canina, lo que demuestra su importancia biológica y su posible utilidad en el diagnóstico (9).
Teniendo en cuenta las características del gen que codifica la proteína HSP20, nos proponemos explorar su utilidad para la detección de especies del género Leishmania de diverso origen geográfico, utilizando la PCR, con la finalidad de contribuir a la identificación de un nuevo blanco genético de probable utilidad y posible aplicación a la epidemiología molecular en áreas endémicas de leishmaniosis.
MATERIALES Y MÉTODOS
CEPAS DE REFERENCIA
Se utilizó ADN obtenido de parásitos pertenecientes a 45 cepas de referencia de Leishmania (Tabla 1) que fueron donados por el Laboratorio de Parasitología Molecular del Instituto de Medicina Tropical de Amberes. Estos parásitos son representativos de 14 especies procedentes del Nuevo y Viejo Mundo.

DETERMINACIÓN DE LA CONCENTRACIÓN Y PUREZA DEL ADN PROCEDENTE DE CEPAS
Este procedimiento se realizó para determinar la concentración de ácidos nucleicos de las cepas de referencia que se utilizaron durante la optimización de la PCR-hsp20 y en los estudios de sensibilidad analítica. La concentración de ADN genómico se determinó mediante lectura de la absorbancia a 260 nm en un espectrofotómetro (Spectrophotomer JASGO V-630, Tokio, Japón) y teniendo en cuenta que una unidad de densidad óptica a 260 nm, corresponde con 50 µg de ADN de doble cadena, según la fórmula: concentración de ADN (µg/ µl) = (50 x dilución x D.O 260 nm) / 1 000. La lectura también se realizó a 280 nm, longitud de onda a la cual absorben las proteínas. Para determinar la pureza del material se utilizó la siguiente relación: D.O 260 nm / 280 nm, la cual debe estar en un rango entre 1.8- 2.0.
MUESTRAS CLÍNICAS Y ADN DE OTROS AGENTES INFECCIOSOS
Se evaluaron cuatro muestras clínicas de pacientes atendidos en el Instituto de Medicina Tropical Pedro Kourí (IPK), La Habana; ADN extraído de 94 muestras clínicas de personas con antecedentes clínicos y/o epidemiológicos de leishmaniosis que habían sido diagnosticadas utilizando diferentes métodos en los laboratorios de origen (Perú, Bolivia, Colombia) y ADN obtenido de 24 raspados del borde de lesiones cutáneas de pacientes cubanos con enfermedades cutáneas distintas de leishmaniosis (Tabla 2).

Se utilizaron, además, muestras de ADN de distintos agentes infecciosos para estudios de especificidad analítica, provenientes del Banco de ADN del Laboratorio de Biología Molecular del Departamento de Parasitología, IPK: Toxoplasma gondii; del Laboratorio de Parasitología Molecular del Instituto de Medicina Tropical de Amberes (IMT-A): Trypanosoma cruzi, T. rangeli, T.c.marikellei, T. brucei brucei, T. b. gambiense, T. brucei rhodesiense, T. congolense, T. vivax, T. equiperdum, T. evansi, T. theileri, Plasmodium falciparum, Schistosoma mansoni ; del Departamento de Bacteriología del IPK: Escherichia coli, Candida albicans, Candida parasilopsis, Cryptoccocus neoformans, Haemophilus influenzae, Streptococcus pneumoniae, Mycobacterium tuberculosis, Mycobacterium habana, Staphylococcus aureus, Staphylococcus epidermidis, Pseudomonas aeruginosa, Neisseria meningitidis y del Departamento de Virología del IPK: Herpes zoster, Citomegalovirus (CMV) y Herpes simplex.
EXTRACCIÓN DE ADN
El ADN de las muestras obtenidas en el IPK, se extrajo usando el kit de extracción QIAamp DNA mini kit (Qiagen, Hilden, Alemania) siguiendo las instrucciones del fabricante, se resuspendió en volúmenes entre 10-50 µl de agua, en dependencia del tamaño de la muestra inicial.
CRITERIOS PARA ESTABLECER LOS GRUPOS DE ESTUDIO: POSITIVO Y NEGATIVO
Todas las muestras de ADN que correspondían con casos sospechosos, y que fueron previamente diagnosticados en los laboratorios de origen utilizando diferentes métodos, se verificaron mediante la realización de la PCR-18S (rADN) siguiendo las condiciones reportadas por Deborggraeve et al. (7). Los cebadores fueron sintetizados por Sigma Genosys, Inglaterra y las secuencias (5’-3’) en cada caso son:
18S-L-positivo CGTAGTTGAACTGTGGGCTGTGC
18S-L-negativo ACTCCCGTGTTTCTTGTTTCTTTGAA
Como molde de la reacción se usaron 5µL del ADN obtenido de las muestras clínicas. Se incluyó un control negativo con agua en todos los casos y un control positivo que contuvo 100 fg de la cepa de L. amazonensis MHOM/BR/72/LTB0016.
El total de muestras clínicas que resultaron positivas se consideraron como grupo control positivo (casos con leishmaniosis) para realizar una evaluación preliminar de la PCR-hsp20. Todas las muestras cuyos resultados fueron negativos mediante la PCR-18S se consideraron como grupo control negativo (casos sin leishmaniosis). Estos grupos constituyeron la base para calcular la sensibilidad y especificidad diagnóstica de la nueva PCR en estudio.
NORMALIZACIÓN DE LA PCR-hsp20
Se utilizó la cepa de L. amazonensis (MHOM/BR/73/ M2269), disponible en nuestro laboratorio. Los cebadores y el programa de corrida, previamente reportado por Fraga etal. (11) amplifican un fragmento del gen hsp20 de 370pb. Las secuencias 5´- 3´de los cebadores son las siguientes:
hsp20F: RGRGACTCGCTCAKCAACAGCG hsp20R: CGTTGAAGSTGGCCTTGATTTTGCTG
El programa de ciclos consistió en: desnaturalización inicial a 95 °C por 5 min; seguido de 35 ciclos consistentes en: 94 °C por 40 s, 58 °C durante 1 min, y 72 °C durante 1 min; con un paso de extensión final de 8 min a 72 °C. Se evaluaron distintas concentraciones de las componentes de la mezcla de reacción: cebadores [0,2; 0,4; 0,6; 0,8; 1µM], enzima polimerasa [0,5; 1; 1,5; 2 U] y MgCl2 [1,5; 2; 2,5; 3; 3,5; 4,0 mM], cada uno de manera individual y manteniendo el resto de los parámetros constantes.
En todos los experimentos se utilizó un termociclador
MJ Research, MiniCycler (Waltham, MA, EUA). Cada resultado se verificó mediante electroforesis en gel de agarosa 2%, teñido con bromuro de etidio, en tampón de corrida TBE a 130V por 30 min.
SENSIBILIDAD Y ESPECIFICIDAD ANALÍTICA DE LA PCR-hsp20
Para estudiar la sensibilidad se realizaron diluciones se-riadas (1/10) en tampón TE desde 1 ng hasta 10 fg, uti-lizando ADN de cepas representativas de 11 especies pertenecientes a ambos subgéneros: L. (Leishmania) y L. (Viannia) (Tabla 1). En el análisis de la especificidad, se utilizó como molde 10 ng de ADN de los distintos microor-ganismos. Posteriormente, se realizó cada PCR siguiendo las condiciones previamente normalizadas, en el mismo volumen final. Se preparó un tubo control positivo con igual cantidad de ADN de la cepa de L. amazonensis MHOM/ BR/72/M2269 y un control negativo con agua bidestilada estéril. Los productos de amplificación se observaron en una corrida electroforética en las condiciones descritas.
EVALUACIÓN DE LA UTILIDAD DE LA PCR-hsp20
Se realizó la PCR-hsp20 en las condiciones previamente normalizadas utilizando como molde 100 fg del ADN obtenido de las cepas de referencia de Leishmania (Tabla 1) y se determinó en qué especies es posible obtener el amplicón de la talla esperada. A continuación, se evaluaron todas las muestras clínicas sospechosas de infección con Leishmania, así como las muestras de pacientes con otras enfermedades cutáneas, utilizando 5 µL del ADN en todos los casos.
CONSIDERACIONES ÉTICAS
En todos los casos, la obtención de la muestra estuvo precedida de la aprobación del paciente mediante la firma del consentimiento informado y de la aprobación de los comités de ética correspondientes de cada institución.
RESULTADOS
Se determinaron las concentraciones y cantidades óptimas para los componentes fundamentales de la mezcla de reacción, teniendo en cuenta que se obtuvieran bandas de amplificación nítidas a la menor concentración posible en cada evaluación. Las condiciones establecidas fueron: concentración de cebadores (0,8 µM de cada uno); MgCl2, (3 mM) y cantidad de la enzima Taq polimerasa (0,5 U).
El estudio de sensibilidad analítica de la PCR-hsp20 incluyó especies que representan los subgéneros L. (Leishmania ) y L. (Viannia), puesto que con anterioridad no se había empleado este blanco para la detección del parásito. En la mayoría de los casos se pudo detectar hasta 10 fg de ADN genómico del parásito. Solamente para L. tropica y L. major la detección fue menor (100 fg), lo que se muestra en la Figura 1.
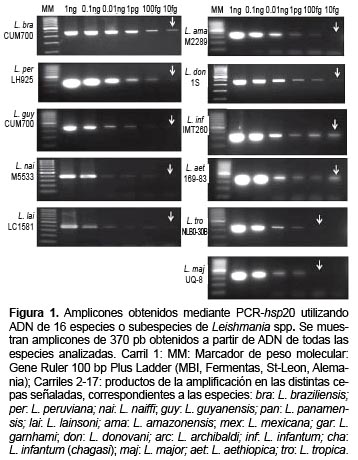
VALIDEZ Y SEGURIDAD DIAGNÓSTICA DE LA PCR-hsp20
La validez de la PCR-hsp20 se evaluó en base a la determinación de la sensibilidad y especificidad diagnóstica (12). La prueba de referencia fue la PCR-18S. A partir de este resultado, se agruparon las muestras de pacientes en dos grupos, como se describió. Para evaluar la seguridad, se determinaron los valores predictivos positivo (VPP) y negativo (VPN) (13). En el análisis se utilizó una tabla de contingencia de 2x2, donde se enfrentó el resultado de la PCR-hsp20 (en filas), con el resultado de la prueba de referencia (en columnas). Para determinar la validez y seguridad diagnóstica se utilizó el programa EPIDAT 3.1 (Dirección General de Salud Pública, Organización Panamericana de la Salud, Galicia, España).
DETERMINACIÓN DEL ÍNDICE DE CONCORDANCIA
Se determinó el índice de concordancia entre la prueba de referencia y la prueba en estudio usando el coeficiente de correlación Kappa de Cohen (14), utilizando el Programa Epidat 3.1. La interpretación se realizó de acuerdo a la escala siguiente: excelente (0,81-1,00), bueno (0,61-0,8), moderado (0,41-0,6), débil (0,21-0,4) y no significativo (0,0-0,2) (15).
La especificidad analítica fue adecuada. Sin embargo, se obtuvieron amplicones inespecíficos de diferentes tallas, comprendidas entre 500 y 1000 pb, para algunas especies del género Trypanosoma: (T.c. marinkelli; T.b. brucei; T.b. gambiense; T.b. rhodesiense; T. congolese; T. vivax y T. equiperdum; así como bandas entre 200 y 600 pb para CMV. Debido a que no se contó con un número elevado de cepas representativas de esas especies, no fue posible determinar si los amplicones son consistentes en cada uno de los casos. En el resto de los microorganismos evaluados no ocurrió amplificación.
En las condiciones establecidas, se obtuvo la banda de la talla esperada mediante PCR-hsp20, en las 14 especies de Leishmania analizadas (Figura 2). Este resultado se corroboró al evaluar la amplificación utilizando como molde ADN puro proveniente de las 45 cepas evaluadas, correspondientes a estas mismas especies.

En relación a las 98 muestras sospechosas evaluadas por el método de referencia, 96 resultaron positivas por PCR-18S y dos resultaron negativas. Las 24 muestras de pacientes con otras enfermedades cutáneas resultaron negativas a Leishmania usando este protocolo.
La validez de la PCR-hsp20 se determinó para el total de muestras evaluadas (Tabla 3). La sensibilidad diagnóstica fue de 86,5% y la especificidad de 100%. Los resultados de la amplificación obtenida para algunas de las muestras se pueden observar en la Figura 3. La seguridad diagnóstica se expresó mediante los valores predictivos positivo y negativo: VPP: 100% y VPN: 66,7%. El índice de concordancia entre el método de referencia y la PCR-hsp20 fue de ƙ=0,731, lo que se considera una buena concordancia.


DISCUSIÓN
El diagnóstico de la leishmaniosis continúa siendo objeto de atención por parte de la comunidad médica y científica, interesados en la disponibilidad de métodos sensibles y específicos que permitan la definición certera del caso clínico, de forma oportuna. Esto posibilita la indicación de un tratamiento eficaz, brindar al paciente un seguimiento adecuado y tomar las medidas de control necesarias, todo lo cual ha estimulado el desarrollo de técnicas basadas en la detección molecular.
Para ese propósito, la PCR ha demostrado ser la técnica más sensible y específica, su uso está restringido al tercer nivel de cuidados hospitalarios y laboratorios de investigación, lo que constituye una desventaja para la mayoría de las áreas endémicas. Sin embargo, en la actualidad muchos países en vías de desarrollo cuentan con facilidades para el estudio molecular de diversos agentes infecciosos (16) cuestión que podría ser tomada en cuenta en contextos similares, para abordar el diagnóstico molecular de algunas enfermedades desatendidas, entre ellas la leishmaniosis.
Recientemente, se ha apoyado la idea de utilizar el gen hsp70 para la detección y la identificación de la especie de Leishmania infectante en diferentes zonas geográficas (17,18). Sin embargo, la detección molecular del parásito a nivel de género continúa siendo muy importante para ofrecer un diagnóstico rápido, y apoyar estudios ecoepidemiológicos que involucren vectores y reservorios. Para esto se precisa de protocolos con buen desempeño y elevada sensibilidad y especificidad.
La sensibilidad analítica del método optimizado fue excelente para la mayoría de las especies representantes de los dos subgéneros: L. (Viannia) y L. (Leishmania), lo que permitiría detectar cantidades mínimas de un solo parásito, aunque para dos especies de este último:
L. tropica y L. major, fue menor. Esta diferencia en la sensibilidad pudiera ser una consecuencia de la calidad del ADN, de su degradación, o de la presencia de elementos inhibitorios que afectan la amplificación.
De otra parte, existen pocos estudios de secuenciación de este gen, por lo cual conocer la secuencia de un número mayor de cepas de esas especies ayudaría a arribar a resultados más concluyentes sobre las posibles zonas polimórficas que pudieran afectar los sitios de unión y en consecuencia, provocar un mal apareamiento entre los cebadores utilizados y las cadenas complementarias respectivas. Otra posible explicación pudiera ser la existencia de diferentes copias del gen, como ocurre con el gen que codifica la proteína de choque térmico HSP70 (19,20) aspecto que aún no se ha reportado para el gen hsp20, por lo cual la secuenciación completa del gen hsp20 debería constituir una prioridad en el futuro.
A pesar de que se obtuvo amplificación cuando se utilizó ADN de varias especies de trypanosomátidos, así como de CMV, en ninguno de los casos los amplicones obtenidos tuvieron la talla esperada, por lo cual se consideran amplificaciones inespecíficas, que no afectan la especificidad de la técnica. En contraste, otros protocolos han notificado la amplificación simultánea con ADN de organismos relacionados, como Trypanosoma cruzi (21).
La PCR-hsp20 normalizada amplificó un fragmento de 370pb del gen deseado en las 14 especies de interés a partir del ADN obtenido del total de cepas analizadas, con bandas intensas en todos los casos. Esto resulta muy favorable con vistas al posible uso diagnóstico del protocolo, por cuanto existe una gran variedad de especies de importancia médica en las distintas áreas geográficas, que podrían ser teóricamente detectadas, lo que no ocurre con propuestas previas dirigidas a la amplificación de dos o varias especies circulando en un país, o un área geográfica (22).
Con relación a la especificidad, a pesar de que se alcanzó un 100% en el análisis diagnóstico, sería necesario evaluar muestras negativas provenientes de áreas endémicas para arribar a resultados concluyentes, puesto que el grupo de muestras analizado provino, en gran mayoría, de pacientes con otras enfermedades que viven en área no endémica de leishmaniosis.
Por otra parte, consideramos que la sensibilidad diagnóstica obtenida para la PCR-hsp20 es adecuada para sugerir su uso como método de detección en los casos de leishmaniosis cutánea, a pesar de que comparar los niveles de sensibilidad es difícil debido a la diferencia en los tipos de muestras y criterios de inclusión tomados en cuenta por otros autores. Sin embargo, el valor obtenido, de 86,45%, se encuentra en el rango de sensibilidad reportada en zonas específicas de circulación usando otras dianas genéticas, como kDNA, de amplio uso y que presenta 10 mil copias en el parásito (23), con valores de 88,4% (LC); 86,4% (LMC) (24); 89,3 (LC) (25); 98,7% (LC) (26); SSUrRNA 84,6% (LC) (27) y PRSI 87,5% (LC) (28). Otros autores por el contrario, informan valores muy bajos, como 48,7% (29).
Como ventaja, el empleo de esta PCR, diseñada para la detección del género Leishmania, pudiera posibilitar la identificación posterior de la especie infectante a través de la secuenciación nucleotídica, pues se demostró recientemente que las diferencias en este mismo fragmento del gen hsp20 discriminan las especies de importancia médica del Nuevo y el Viejo Mundo, incluyendo entidades que no se logran distinguir por otros protocolos, (11). Esto pudiera ser de interés en laboratorios que disponen de facilidades para la secuenciación.
Recientemente se confirmó que es posible la detección de Leishmania en muestras tomadas de forma no invasiva, como el papel de filtro impregnado o el cepillado citológico de la lesión, lo que resulta más conveniente para el paciente (30,32). De igual forma, se demostró que un mismo protocolo de amplificación molecular, en este caso basado en el ADN del kinetoplasto, se desempeña de manera diferente en relación al tipo de muestra clínica (33). Sería interesante realizar evaluaciones posteriores de la PCR-hsp20 en diferentes escenarios, donde se evalúen diferentes tipos de muestras clínicas o formas de presentación de la enfermedad, para verificar las posibles ventajas y desventajas de este proceder.
La PCR-hsp20 permite detectar 14 especies de Leishmania presentes en todas las áreas geográficas (Nuevo Mundo y Viejo Mundo), lo que le confiere un valor adicional a otras propuestas existentes. Su adecuada sensibilidad, especificidad y buen desempeño, estimulan la realización de nuevas evaluaciones para profundizar en sus bondades diagnósticas u otros beneficios concernientes al paciente, lo que permitirá validar su aplicación y utilidad en variados contextos.
Agradecimientos: la investigación se benefició de recursos obtenidos mediante el proyecto FAIII de cooperación entre el IPK y el IMTA (Cooperación Belga para el Desarrollo). Se aprecia la contribución del Laboratorio de Parasitología Molecular del CBMSO, Universidad Autónoma de Madrid. Los autores expresan su agradecimiento al Prof. Jean Claude Dujardin (IMTA, Bélgica), por su constante apoyo en el desarrollo de investigaciones encaminadas al mejoramiento del diagnóstico molecular de la leishmaniosis.
Fuentes de financiamiento: esta investigación se benefició de recursos obtenidos del Proyecto FA3-III, Programa DGOS de Colaboración entre el Instituto de Medicina Tropical de Amberes y el IPK; y de la colaboración del Centro de Biología Molecular Severo Ochoa, Madrid, España.
Conflictos de interés: los autores declaran no tener conflictos de interés.
REFERENCIAS BIBLIOGRÁFICAS
1. Killick-Kendrick R. Phlebotomine vectors of the leishmaniosis: A review. Med Vet Entomol. 1990 Jan;4(1):1-24.
2. Ready PD. Biology of phlebotomine sand flies as vectors of disease agents. Annu Rev Entomol. 2013;58:227-50. doi: 10.1146/annurev-ento-120811-153557.
3. Goto H, Lauletta Lindoso JA. Cutaneous and mucocutaneous leishmaniasis. Infect Dis Clin North Am. 2012 Jun;26(2):293-307. doi: 10.1016/j.idc.2012.03.001.
4. Reithinger R, Dujardin JC. Molecular diagnosis of leishmaniosis: current status and future applications. J Clin Microbiol. 2007 Jan;45(1):21-5. Epub 2006 Nov 8.
5. Antinori S, Calattini S, Piolini R, Longhi E, Bestetti G, Cascio A, et al. Is real-time polymerase chain reaction (PCR) more useful than a conventional PCR for the clinical management of leishmaniosis? Am J Trop Med Hyg. 2009 Jul;81(1):46-51.
6. Deborggraeve S, Boelaert M, Rijal S, De Doncker S, Dujardin JC, Herdewijn P, Büscher P. Diagnostic accuracy of a new Leishmania PCR for clinical visceral leishmaniasis in Nepal and its role in diagnosis of disease. Trop Med Int Health. 2008 Nov;13(11):1378-83. doi: 10.1111/j.1365-3156.2008.02154.x.
7. Deborggraeve S, Laurent T, Espinosa D, Van der Auwera G, Mbuchi M, Wasunna M, et al. A simplified and standardized polymerase chain reaction format for the diagnosis of leishmaniasis. J Infect Dis. 2008 Nov 15;198(10):1565-72. doi: 10.1086/592509.
8. Espinosa D, Boggild AK, Deborggraeve S, Laurent T, Valencia C, Pacheco R, et al. Leishmania OligoC-TesT as a simple, rapid, and standardized tool for molecular diagnosis of cutaneous leishmaniosis in Peru. J Clin Microbiol. 2009 Aug;47(8):2560-3. doi: 10.1128/ JCM.00259-09.
9. Montalvo-Álvarez AM, Folgueira C, Carrión J, Monzote-Fidalgo L, Cañavate C, Requena JM. The Leishmania HSP20 is antigenic during natural infections, but, as DNA vaccine, it does not protect BALB/c mice against experimental L. amazonensis infection. J Biomed Biotechnol. 2008;2008:695432. doi: 10.1155/2008/695432.
10. Folgueira C, Cañavate C, Chicharro C, Requena JM. Genomic organization and expression of the HSP70 locus in New and Old World Leishmania species. Parasitology. 2007 Mar;134(Pt 3):369-77.
11. Fraga J, Montalvo AM, Van der Auwera G, Maes I, Dujardin JC, Requena JM. Evolution and species discrimination according to the Leishmania heat-shock protein 20 gene. Infect Genet Evol. 2013 Aug;18:229-37. doi: 10.1016/j.meegid.2013.05.020.
12. Altman DG, Bland JM. Diagnostic tests 1: sensitivity and specificity. BMJ. 1994 Jun 11;308(6943):1552.
13. Altman DG, Bland JM. Diagnostic tests 2: Predictive values. BMJ. 1994 Jul 9;309(6947):102.
14. Cohen J. A coefficient of agreement for nominal scales. Educ Psychol Meas. 1960;20:37-46.
15. Landis JR, Koch GG. The measurement of observer agreement for categorical data. Biometrics 1977 Mar;33(1):159-74.
16. Tak V, Mirdha BR, Yadav P, Vyas P, Makharia GK, Bhatnager S. Molecular characterisation of Giardia intestinalis assemblages from human isolates at a tertiary care centre of India. Indian J Med Microbiol. 2014 Jan-Mar;32(1):19-25. doi: 10.4103/0255-0857.124290.
17. Montalvo AM, Fraga J, Maes I, Dujardin JC, Van der Auwera G. Three new sensitive and specific heat-shock protein 70 PCRs for global Leishmania species identification. Eur J Clin Microbiol Infect Dis. 2012 Jul;31(7):1453-61. doi: 10.1007/ s10096-011-1463-z.
18. Fraga J, Veland N, Montalvo AM, Praet N, Boggild AK, Valencia BM, et al. Accurate and rapid species typing from cutaneous and mucocutaneous leishmaniasis lesions of the New World. Diagn Microbiol Infect Dis. 2012 Oct;74(2):142-50. doi: 10.1016/j. diagmicrobio.2012.06.010.
19. Van der Auwera G, Maes I, De Doncker S, Ravel C, Cnops L, Van Esbroeck M, et al. Heat-shock protein 70 gene sequencing for Leishmania species typing in European tropical infectious disease clinics. Euro Surveill. 2013 Jul 25;18(30):20543.
20. Zurita AI, Rodríguez J, Piñero JE, Pacheco R, Carmelo E, del Castllo A, et al. Cloning and characterization of the Leishmania (Viannia) braziliensis Hsp70 gene. Diagnostic use of the C-terminal fragment rLb70(513-663). J Parasitol. 2003; 89:372-8.
21. García L, Kindt A, Bermudez H, Llanos-Cuentas A, De Doncker S, Arévalo J, et al. Culture independent species typing of neotropical Leishmania for clinical validation of a PCR-based assay targeting heat shock protein 70 genes. J Clin Microbiol 2004 May;42(5):2294-7.
22. Gadisa E, Kuru T, Genet A, Engers H, Aseffa A, Gedamu L. Leishmania donovani complex (Kinetoplastida, Trypanosomatidae): comparison of deoxyribonucleic acid based techniques for typing of isolates from Ethiopia. Exp Parasitol. 2010 Oct;126(2):203-8. doi: 10.1016/j.exppara.2010.04.026.
23. Rodgers MR, Popper SJ, Wirth DF. Amplification of kinetoplast DNA as a tool in the detection and diagnosis of Leishmania. Exp Parasitol 1990 Oct;71(3):267-75.
24. Satow MM, Yamashiro-Kanashiro EH, Rocha MC, Oyafuso LK, Soler RC, Cotrim PC, et al. Applicability of kDNA-PCR for routine diagnosis of American tegumentary leishmaniasis in a tertiary reference hospital. Rev Inst Med Trop Sao Paulo. 2013 Nov-Dec;55(6):393-9. doi: 10.1590/ S0036-46652013000600004.
25. Ampuero J, Rios AP, Carranza-Tamayo CO, Romero GA. Genus-specific kinetoplast-DNA PCR and parasite culture for the diagnosis of localized cutaneous leishmaniosis : applications for clinical trials under field conditions in Brazil. Mem Inst Oswaldo Cruz. 2009 Nov;104(7):992-7.
26. Bensoussan E, Nasereddin A, Jonas F, Schnur LF, Jaffe CL. Comparison of PCR assays for diagnosis of cutaneous leishmaniosis. J Clin Microbiol. 2006;44(4):1435-9.
27. Lemrani M, Hamdi S, Laamrani A, Hassar M. PCR detection of Leishmania in skin biopsies. J Infect Dev Ctries. 2009 Sep 15;3(2):115-22.
28. Rotureau B, Ravel C, Couppié P, Pratlong F, Nacher M, Dedet JP, et al. Use of PCR-Restriction Fragment Length Polymorphism analysis to identify the main New World Leishmania species and analyze their taxonomic properties and polymorphism by application of the assay to clinical samples. J Clin Microbiol. 2006 Feb;44(2):459-67.
29. Martins L, Alexandrino A, Guimarães G. Detection of Leishmania braziliensis DNA in American tegumentary leishmaniasis patients. Rev Saude Publica. 2010;44:571-4.
30. Boggild AK, Valencia BM, Espinosa D, Veland N, Ramos AP, Arevalo J, et al. Detection and species identification of Leishmania DNA from filter paper lesion impressions for patients with American cutaneous leishmaniosis. Clin Inf Dis 2010; 50:e1-6.
31. Boggild AK, Ramos AP, Valencia BM, Veland N, Calderón F, Arévalo J, et al. Diagnostic performance of filter paper lesion impression PCR for secondarily infected ulcers and nonulcerative lesions caused by cutaneous leishmaniasis. J Clin Microbiol. 2011 Mar;49(3):1097-100. doi: 10.1128/ JCM.02457-10.
32. Valencia BM, Veland N, Alba M, Adaui V, Arévalo J, Low DE, et al. Non-invasive cytology brush PCR for the diagnosis and causative specis identification of American cutaneous leishmaniosis in Peru. PLoS One. 2012 Nov;7(11):e49738.
33. Neitzke-Abreu HC, Venazzi MS, Bernal MVZ, Reinhold-Castro KR, Vagetti F, Alves-Mota Camila, et al. Detection of DNA from Leishmania (Viannia): Accuracy of polymerase chain reaction for the diagnosis of cutaneous Leishmaniasis. PLoS ONE. 2013 Jul;8:e62473.
Correspondencia:
Ana Montalvo.
Dirección: Autopista Novia del Mediodía Km 6 y ½, La Lisa, La Habana,
Cuba.
Teléfono: 537 255 3601
Correo electrónico:
amontalvo@ipk.sld.cu
Recibido:
02-06-14
Aprobado: 20-08-14