Original Breve
Betalactamasas de espectro extendido tipo TEM y CTX-M en Klebsiella spp y Escherichia coli aisladas de superficies de ambientes hospitalarios
TEM and CTX-M extended-spectrum beta-lactamase in Klebsiella spp and Escherichia coli isolates from inanimate surfaces of hospital environments
Marco Rivera-Jacinto1,a, Claudia Rodríguez-Ulloa1,b, René Flores Clavo2,c, Luis Serquén López 2,d, Zhandra Arce Gil 3,e
1 Facultad de Ciencias de la Salud, Universidad Nacional de Cajamarca. Cajamarca, Perú.
2 Hospital Regional de Lambayeque. Lambayeque, Perú.
3 Universidad Católica Santo Toribio de Mogrovejo. Lambayeque, Perú.
a Biólogo-microbiólogo, doctor en Ciencias Biomédicas; b bióloga-microbióloga, doctora en Ciencias Biomédicas; c licenciada en Microbiología; d licenciado en Microbiología e licenciada en Microbiología, magíster en Ciencias.
RESUMEN
El objetivo del estudio fue determinar el genotipo BLEE de 15 cepas de enterobacterias resistentes a betalactámicos, aisladas de superficies inanimadas y caracterizadas fenotípicamente como productoras de betalactamasas de espectro extendido. Previa evaluación y tamizaje de las cepas bacterianas, se hizo un PCR para amplificar fragmentos de 1078 pb y 544 pb correspondientes a BLEE tipo TEM y CTX-M. Once cepas presentaron ambos fragmentos a la vez y tres presentaron solamente blaCTX-M. En conclusión, se demostró la presencia de genes BLEE en cultivos de origen ambiental, algunos de los cuales podrían pertenecer a más de un tipo; esta información podría servir de base para implementar medidas de prevención que eviten la trasmisión de bacterias multirresistentes desde superficies inanimadas a los pacientes, principalmente en áreas hospitalarias críticas.
Palabras clave: beta-Lactamasas; Infección hospitalaria; Klebsiella pneumoniae; Escherichia coli; Reacción en cadena de la polimerasa (fuente: DECS/Bireme)
ABSTRACT
The aim of the study was to determine the genotype of 15 ESBL strains of Enterobacteriaceae resistant to beta-lactams, isolated from inanimate surfaces and phenotypically characterized as producing extended-spectrum beta-lactamase. After evaluation and screening of the bacterial strains, a PCR was conducted to amplify fragments of 1078 bp and 544 bp corresponding to type TEM and CTX-M ESBL. Eleven strains presented both fragments at the time and only three had blaCTX-M. In conclusion, the presence of ESBL genes in cultures from the environment was demonstrated, some of which may belong to more than one type. This information could serve as a basis for implementing preventive measures to prevent the transmission of multiresistant bacteria from inanimate surfaces to patients, mainly in critical hospital areas.
Key words: beta-Lactamases; Cross infection; Klebsiella pneumoniae; Escherichia coli; Polymerase chain reaction (source: MeSH/NLM)
INTRODUCCIÓN
Las betalactamasas son enzimas bacterianas capaces de hidrolizar los antimicrobianos betalactámicos, inactivándolos y haciéndolos inefectivos. Las enzimas de espectro extendido (BLEE) están entre las de mayor relevancia clínica e incluyen tres tipos principales, TEM, SHV y CTX-M (1). Las TEM provienen de las clásicas TEM-1 y TEM-2 contenidas en plásmidos, mientras que las SHV tienen origen cromosómico (1,2). Las CTX-M, de historia evolutiva distinta, tienen actividad BLEE intrínseca movilizada desde su predecesora cromosómica en Kluyvera (2), y son las BLEE de mayor reporte mundial, predominando en agentes de infecciones nosocomiales (IN) como Escherichia coli y Klebsiella spp (2-4).
Aunque la fuente endógena es la más importante, el ambiente hospitalario es un reservorio a tener en cuenta (5,6) debido a que las superficies inanimadas pueden ser colonizadas por bacterias productoras de BLEE (7,8), y constituirse en fuente potencial de contaminación cruzada y colonización de las manos del personal de salud. El objetivo de este trabajo fue determinar el genotipo BLEE de 15 cepas de enterobacterias que fueron aisladas de superficies inanimadas, y caracterizadas fenotípicamente como productoras BLEE en un estudio previo en el Hospital Regional de Cajamarca (7), en el norte del Perú.
EL ESTUDIO
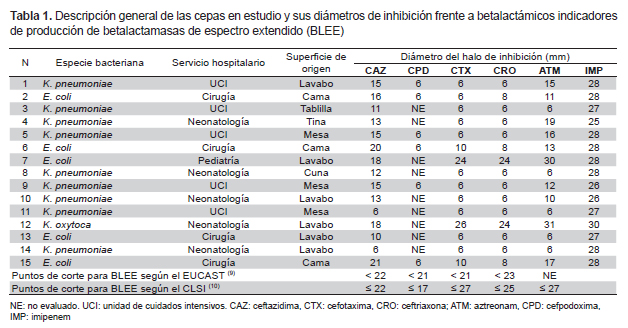
Se colectaron mediante hisopado 125 muestras desde lavatorios, mesas, camas y otras superficies en las salas de cirugía, pediatría, medicina, neonatología, maternidad y unidad de cuidados intensivos (UCI), durante el periodo de septiembre-2009 a junio-2010. De 20 cepas de E. coli, 19 de K. pneumoniae y 4 de K. oxytoca, 15 cepas (5 de E. coli, 9 de K. pneumoniae y 1 de K. oxytoca), caracterizadas como productoras BLEE mediante pruebas fenotípicas (sinergia de doble disco y disco combinado) en un estudio previo (7), fueron reactivadas y sometidas nuevamente a pruebas de cribado con betalactámicos indicadores empleando los puntos de corte propuestos por el European Committee on Antimicrobial Susceptibility Testing (EUCAST) (9) y el Clinical and Laboratory Standars Institute (CLSI) (10).
La extracción de ADN de cada cepa se hizo con el kit Pure Link Genomic DNA (Invitrogen), según el protocolo para muestras bacterianas. Los primers empleados fueron blaTEM F-5’-ATAAAATTCTTGAAGACGAAA-3’, R-5’-GACAGTTACCAATGCTTAATC-3’ y blaCTX-M F-5’-TTTGCGATGTGCAGTACCAGTAA-3’, R-5’- CGATATCGTTGGTGGTGCCAT-3’, para amplificar fragmentos de 1078 pb de TEM (11), y de 544 pb de CTX-M (12). La reacción fue ajustada al volumen final de 50 mL según el Fast Start Taq DNA Polymerase (Roche): 25,6 mL agua PCR; 5 mL de buffer 10x, 4 mL 25mM MgCl2, 1mL de mix de nucleótidos; 5 mL de cada primer; 0,4 mL de Taq DNA polimerasa y 4 mL del ADN extraído. La PCR se realizó en Verita Thermal Cycler (Applied Biosystems), bajo las condiciones descritas previamente (11,12).
La electroforesis se hizo a 100 V por 45-60 minutos en gel de agarosa 1,5% (Promega, USA) con bromuro de etidio (1,0 mg/mL); los productos se evidenciaron a través de un transiluminador Biorad Pharos FX Plus®, editado con el programa Quantityone®. El tamaño del ADN amplificado se verificó usando un marcador de peso molecular de 1500 pb.
HALLAZGOS
Todas las cepas evaluadas con ceftazidima (30μg) y cefpodoxima (10μg) presentaron diámetros de inhibición menores a los señalados como puntos de corte para el tamizaje de BLEE por el CLSI y el EUCAST. Las cepas 7 (E. coli) y 12 (K. oxytoca) tuvieron diámetros para cefotaxima (30μg) y ceftriaxona (30μg) mayores al corte según el EUCAST pero menores al del CLSI; estas mismas cepas tuvieron halos mayores a los 30 mm para aztreonam (30 ug). Con imipenem (10 μg), todas las cepas mostraron halos mayores al corte según el EUCAST para carbapenemasas (Tabla 1).
Siete cepas de K. pneumoniae y 4 cepas de E. coli presentaron ambos genes (blaTEM y blaCTX-M), 2 cepas de K. pneumoniae y la única de K. oxytoca presentaron solo blaCTX-M, y una cepa de E. coli aislada de pediatría no presentó los genes evaluados (Figura 1 y 2).
DISCUSIÓN
Diversos estudios reportan que K. pneumoniae es hallada con más frecuencia que E. coli en el ambiente hospitalario, incluyendo superficies inanimadas (5,6,8), lo cual coincide con este reporte (Tabla 1). Esto se debe a la mayor capacidad de Klebsiella spp. para sobrevivir sobre las superficies y es fundamentalmente importante porque a partir de ellas es posible la colonización de los pacientes, en mayor o menor frecuencia, dependiendo de la especie bacteriana y de sus diferencias biológicas intrínsecas (5,6,8). La alta transmisibilidad que presentan las bacterias productoras de BLEE también ha sido demostrada en estos estudios, y explica ese dialelo en el que las superficies inanimadas son contaminadas por los pacientes infectados o por el personal de salud colonizado, que a su vez se contaminan desde las mismas superficies (6). El problema se agrava cuando estos microorganismos expresan genes de resistencia a múltiples antimicrobianos, convirtiendo el ambiente hospitalario en un reservorio de bacterias multirresistentes de difícil erradicación, aun mediante los procedimientos más prolijos de limpieza que emplee el hospital (6).
De las 15 cepas estudiadas, 14 presentaron el genotipo BLEE CTX-M y 11 presentaron los genotipos BLEE CTX-M y TEM (Figura 1 y 2). La mayor frecuencia del primero coincide con las mayores prevalencias con la que se reportan estas enzimas en el mundo (2,3) lo cual es importante debido a la amplia diversidad y rápida diseminación con que esta BLEE se presenta durante las IN. Algunos autores afirman que es común entre las bacterias productoras de CTX-M exhibir diferentes fenotipos de resistencia (2), en este estudio, 13/14 exhiben una marcada actividad cefotaximasa en las pruebas de cribado y solo la cepa de K. oxytoca presenta un patrón diferente; además, es de resaltar la alta actividad ceftazidimasa del gen CTX-M en las cepas 11, 12 y 14 (Tabla 1), que fueron negativas para el gen TEM. El fenotipo de resistencia donde los cultivos presentan ambos tipos de BLEE, es una estrategia bacteriana de coproducción cada vez más común y peligrosa entre las Klebsiella y E. coli, que a veces les permite enfrentar con mucha frecuencia y efectividad a otros grupos de antimicrobianos (4). En el caso del cultivo 7, que no muestra ninguno de los genes evaluados, pero muestra un fenotipo productor de BLEE en el cribado, su genotipo podría pertenecer al tipo SHV o a un gen cromosómico inducible AmpC, que en algunos casos, es difícil de detectar. Estos mecanismos también pueden ser responsables del fenotipo resistente en cultivos con genotipo TEM y CTX-M, sin embargo, para asegurar que estamos frente a un tipo específico de BLEE TEM o de BLEE CTX-M es necesario secuenciar, siendo esta una limitación muy importante en el trabajo realizado.
Al problema del reservorio bacteriano BLEE positivo se suman las complicaciones que hay en su control y tratamiento, debido a la facilidad con que se diseminan estos y otros genes de resistencia entre las Enterobacteriaceae (1,4). Aunque en el presente trabajo no se realizaron pruebas para determinar el patrón de clonalidad de las muestras, es importante mencionar que las relaciones genéticas entre las cepas seleccionadas se hubiesen podido establecer mediantes técnicas como la electroforesis de campos pulsados, y así determinar cuántos clones circulan en los ambientes hospitalarios o inferir los posibles focos de trasmisión. Este reporte también pretende captar la atención de las autoridades de salud, a fin de poner énfasis en la vigilancia de la trasmisión de estas bacterias y otros patógenos nosocomiales desde el ambiente, especialmente desde superficies inanimadas potencialmente contaminadas en áreas hospitalarias críticas, estableciendo programas de control que detecten la presencia de BLEE y otros mecanismos de resistencia tanto a nivel hospitalario como en otros escenarios, lo cual se hace muy poco en el Perú.
Contribuciones de autoría: MRJ y CRU han participado en la concepción del artículo, la recolección de datos, análisis e interpretación, su redacción, revisión crítica y aprobación de la versión final. RFC, LSL y ZAG han participado en la colección de datos, análisis e interpretación, revisión crítica del artículo, además de aprobar la versión final del manuscrito.
Fuentes de financiamiento: la PCR fue realizada con materiales y equipos del Laboratorio de Biología Molecular de la Dirección de Investigación del Hospital Regional de Lambayeque.
Conflictos de interés: los autores declaramos no tener conflicto de intereses.
Referencias Bibliográficas
1. Rivera A, Larrosa N, Mirelis B, Navarro F. Importancia de los controles de calidad para la detección de la resistencia a antibióticos β-lactámicos en enterobacterias. Enferm Infecc Microbiol Clin 2014;32(Supl 1):30-6. doi: 10.1016/S0213-005X(14)70147-8.
2. D’Andrea MM, Arena F, Pallecchi L, Rossolini GM. CTX-M-type β -lactamases: A successful story of antibiotic resistance. Int J Med Microbiol 2013;303(6-7):305-17. doi: 10.1016/j.ijmm.2013.02.008.
3. Guzmán-Blanco M, Labarca JA, Villegas MV, Gotuzzo E, On behalf of the Latin America Working Group on Bacterial Resistance. Extended spectrum -lactamase producers among nosocomial Enterobacteriaceae in Latin America. Braz J Infect Dis 2014; 18(4):421–33. doi: 10.1016/j. bjid.2013.10.005.
4. Kassakian SZ, Mermel LA. Changing epidemiology of infections due to extended spectrum beta-lactamase producing bacteria. Antimicrob Resist Infect Control. 2014;3(1):9. doi: 10.1186/2047-2994-3-9.
5. Freeman JT, Nimmo J, Gregory E, Tiong A, De Almeida M, McAuliffe GN, et al. Predictors of hospital surface contamination with Extended-spectrum β-lactamase-producing Escherichia coli and Klebsiella pneumoniae: patient and organism factors. Antimicrob Resist Infect Control 2014;3(1):5. doi: 10.1186/2047-2994-3-5.
6. López-Cerero L. Papel del ambiente hospitalario y los equipamientos en la transmisión de las infecciones nosocomiales. Enferm Infecc Microbiol Clin. 2014;32(7):459–64. doi: 10.1016/j.eimc.2013.10.004.
7. Rivera-Jacinto MA. Betalactamasas de espectro extendido en cepas de Escherichia coli y Klebsiella sp aisladas de reservorios inanimados en un hospital del norte del Perú. Rev Esp Quimioter 2012;25(2):161-63.
8. Guet-Revillet H, Le Monnier A, Breton N, Descamps P, Lecuyer H, Alaabouche I, et al. Environmental contamination with extended-spectrum b-lactamases: Is there any difference between Escherichia coli and Klebsiella spp? Am J Infect Control 2012;40(9):845-8. doi: 10.1016/j.ajic.2011.10.007.
9. European Society of Clinical Microbiology and Infectious Diseases. EUCAST guidelines for detection of resistance mechanisms and specific resistances of clinical and/or epidemiological importance. Version 1.0, 2013. Disponible en: http://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Resistance_mechanisms/EUCAST_detection_of_resistance_mechanisms_v1.0_20131211.pdf
10. Clinical and Laboratory Standards Institute (CLSI). Performance Standards for Antimicrobial Susceptibility Testing; 22th Informational Supplement. CLSI Document M100-S22. Wayne, PA: CLSI, 2012.
11. Mabilat C, Goussard S. PCR detection and identification of genes for extendedspectrum β-lactamases. In: DH Persing, Smith TF, Tenover FC, White TJ, editors. Diagnostic molecular microbiology: principles and applications. Washington, D.C: American Society for Microbiology; 1993: p. 553-559.
12. Arce-Gil Z, Llontop-Nuñez J, FloresClavo R, Fernández-Valverde D. Detección del gen CTX-M en cepas de Escherichia coli productoras de β-lactamasas de espectro extendido procedentes del Hospital Regional de Lambayeque; Chiclayo-Perú: Noviembre 2012-Julio 2013. Rev. cuerpo med HNAAA 2013;6(4):13-16.
Correspondencia: Marco Antonio Rivera Jacinto
Dirección: Av. Atahualpa 1050 – Edificio 1D. Lab.1D-105. Ciudad Universitaria,Universidad Nacional de Cajamarca, Cajamarca-Perú.
Teléfono: (+511) 976996558
Correo electrónico: mrivera@unc.edu.pe; marco_riverajacinto@yahoo.es
Recibido: 07-05-15
Aprobado: 02-09-15