
ARTÍCULO ORIGINAL
Detección de virus influenza A, B y subtipos A (H1N1) pdm09, A (H3N2) por múltiple RT-PCR en muestras clínicas
Detection of influenza A, B and subtypes A (H1N1) pdm09, A (H3N2) viruses by multiple QRT-PCR in clinical samples
Pool Marcos1,b, Maribel Huaringa1,c, Nancy Rojas1,a, Victoria Gutiérrez1,a, Sila Ruiton1,d, Emelda Gallardo1,d, Jorge Achata1,e, Marco Galarza2,b
1
Laboratorio de Referencia Nacional de Virus Respiratorios, Centro Nacional
de Salud Pública, Instituto Nacional de Salud. Lima, Perú.
2 Laboratorio de Referencia Nacional de Biología Molecular y
Biotecnología, Centro Nacional de Salud Pública, Instituto Nacional de
Salud. Lima, Perú.
a Biólogo; b magister en Biología molecular; c
tecnólogo médico; d técnico; e PhD en Biología
molecular
RESUMEN
Objetivos. Estandarizar la técnica de reacción en cadena de la polimerasa en tiempo real (RT-PCR) múltiple para la detección de virus influenza A, B y tipificación de subtipos A (H1N1) pdm09, A (H3N2) en muestras clínicas. Materiales y métodos. Se analizaron 300 muestras de hisopado nasofaríngeo. Esta metodología fue estandarizada en dos pasos: la primera reacción detectó el gen de la matriz del virus de influenza A, gen de la nucleoproteína del virus influenza B y el gen GAPDH de las células huésped. La segunda reacción detectó el gen de la hemaglutinina de los subtipos A (H1N1) pandémico (pdm09) y A (H3N2). Resultados. Se identificaron 109 muestras positivas a influenza A y B, de las cuales 72 fueron positivas a influenza A (36 positivas a influenza A (H1N1) pdm09 y 36 positivos a influenza A (H3N2)) y 37 muestras positivas a influenza B. 191 fueron negativas a ambos virus mediante RT-PCR en tiempo real multiplex. Se encontró una sensibilidad y especificidad del 100% al analizar los resultados de ambas reacciones. El límite de detección viral fue del rango de 7 a 9 copias/µL por virus. Los resultados no mostraron ninguna reacción cruzada con otros virus tales como adenovirus, virus sincitial respiratorio, parainfluenza (1,2 y 3), metapneumovirus, subtipos A (H1N1) estacional, A (H5N2) y VIH. Conclusiones. La RT-PCR múltiple demostró ser una prueba muy sensible y específica para la detección de virus influenza A, B y subtipos A (H1N1, H3N2) y su uso puede ser conveniente en brotes estacionales.
Palabras clave: Reacción en cadena de la polimerasa multiplex; Virus de la influenza A; Subtipo H1N1 del virus de la influenza A; Subtipo H3N2 del virus de la influenza A (fuente: DeCS BIREME).
ABSTRACT
Objectives. To describe the clinical and epidemiological characteristics of patients diagnosed with epidermolysis bullosa (EB) at the Instituto Nacional de Salud (INSN) in Lima, Peru; a National Reference Center for this disease. Material and methods. Observational, descriptive and transversal study. We reviewed the clinical histories and laboratory tests of patients diagnosed with EB treated in INSN from 1993 to 2015. Results. 93 patients were registered. The average age was 7.9 ± 5.6 years; 53.8% (n = 50) were boys. Clinical forms corresponded to dystrophic EB with 41 (44.1%) cases, simple EB with 39 (41.9%) union EB cases with 8 (8.6%) and Kindler syndrome with 4 (4.3%) cases. The clinical form could not be identified in a case. A total of 48 cases (51.6%) came from Lima and Callao, and 45 cases (48.4%) from other provinces of the country. Extracutaneous manifestations involved gastrointestinal (44.1%), ocular (37.6%), odontogenic (87.1%), and nutritional (79.6%) involvement, as well as pseudosindactilia (16.1%). Chronic malnutrition (71.6%), acute malnutrition (17.6%) and anemia (62.4%) were found. Mortality corresponded to 6 cases (6.5%). Conclusions. 93 cases of EB were reported in INSN, the predominant clinical presentation was the dystrophic form.
Key words: Multiplex polymerase chain reaction; Influenza A virus; Influenza A virus, H1N1 subtype; Influenza A virus, H3N2 subtype (source: MeSH NLM).
INTRODUCCIÓN
Los virus de influenza son los agentes causales de la gripe, infección respiratoria aguda que causa altas tasas de morbilidad y mortalidad relacionadas con cuadros de neumonía (1). En el mundo, la Organización Mundial de la Salud (OMS) reportó más de 134 510 casos y 816 muertes, y es considerada como una nueva pandemia (2). Debido a la capacidad de mutar y de adaptarse a casi cualquier circunstancia, este virus representa una amenaza para la humanidad. En Perú, hasta julio de 2009, se contaban ya más de 3500 casos de influenza A (H1N1) (2).
La mayoría de las pandemias de gripe se asocian con el tipo A, que representa el patógeno más extendido en humanos y animales. Estos se clasifican en subtipos basados en las variaciones antigénicas de la hemaglutinina (HA) y neuraminidasa (NA) que son glicoproteínas expresadas en la superficie del virus. Los subtipos más comunes del virus influenza A que causan enfermedad en humanos son H1N1pdm09, H1N1 y H3N2 (3).
El secuenciamiento completo del genoma del virus pandémico H1N1pdm09 reveló que se trataba de una reorganización del virus H1N1. Este incluía, al menos, tres secuencias virales diferentes que contenían seis genes de la gripe porcina de Norteamérica (H1N2) y dos genes de la gripe porcina Euro-Asiática (NA de H1N1) y Asiática (proteína de la matriz en H3N2) (4).
La gran variabilidad genética y antigénica que exhiben los virus influenza A se debe a la conformación particular de su genoma, a su alta tasa de mutación y a la reasociación frecuente de sus segmentos genómicos que les confiere un importante potencial pandémico.
Estos mecanismos de variación genética conducen a que la respuesta inmunológica de memoria pueda tener poco o ningún efecto cuando una nueva variante de virus infecta a la población (5).
El análisis filogenético de los virus pandémicos AH1N1 colectados de diferentes regiones del mundo durante el brote del 2009, demostró la presencia de variantes de A (H1N1) con mutaciones específicas en el gen de la NA (6,7).

El aislamiento viral es considerado el gold standard o método de referencia para la detección de virus respiratorios. Inicialmente, el aislamiento viral se realizó en huevos de gallina embrionados, posteriormente, se utilizaron líneas celulares tales como MDCK y Vero, para una mayor recuperación de virus en los medios de cultivo (8). Sin embargo, los avances tecnológicos en el diagnóstico han permitido utilizar otras metodologías que incluyen los anticuerpos monoclonales o métodos moleculares que resultaron ser herramientas poderosas para el diagnóstico de la infección vírica (9-11). Si bien es cierto, el aislamiento viral y las pruebas de inmunofluorescencia (directa o indirecta) son métodos efectivos y complementarios, son laboriosos y demandan un tiempo relativamente largo en comparación con los métodos moleculares (12,13). Desde el año 2004, en que se implementaron los métodos moleculares para la detección de influenza A, a la actualidad, la reacción en cadena de la polimerasa en tiempo real (RT-PCR) es considerado como un método rápido, sensible y específico para la detección de virus influenza (14).
La aparición del virus pandémico A (H1N1) en México y Estados Unidos durante el año 2009, promovió la necesidad de buscar nuevas estrategias para el diagnóstico oportuno mediante el uso de sondas tipo TaqMan, con mayor especificidad que las sondas comerciales tipo SYBR Green y Fret (15). Posteriormente, se implementan las corridas múltiples, que incluyen más de un gen en una misma corrida, de modo que se puede detectar simultáneamente los virus de la influenza A, B y los subtipos de influenza A, tales como H1N1pdm09 (16). En la actualidad, los métodos de detección de los principales virus respiratorios, se basan en el análisis de curvas de melting, cuyo principio está relacionado a temperaturas y fluorescencias que son emitidas en los análisis, de esta manera, se logra discriminar los diferentes tipos de virus influenza y, adicionalmente, poder identificar la resistencia a oseltamivir (17-19). La identificación rápida de los agentes etiológicos virales es indispensable para el monitoreo epidemiológico de esta patología, y brinda la posibilidad de disminuir el uso innecesario de antibióticos y la disminución de la resistencia bacteriana (20).
En el presente estudio se describe la estandarización y optimización de la RT-PCR, múltiple de dos pasos, como una herramienta diagnóstica para la detección de virus influenza A, B y la tipificación de los subtipos más importantes que circulan en nuestro país, tales como H1N1pdm09 y H3N2.
El primer paso de la múltiple RT-PCR consistió en primers y sondas para la matriz de influenza A, la nucleoproteína de influenza y GADH como control interno para células humanas. El segundo paso de la múltiple RT-PCR empleó primers y sondas para las hemaglutininas de los subtipos H1N1pdm09 y H3N2. En esta múltiple RT-PCR de dos pasos, se aumentó la sensibilidad y especificidad usando sondas específicas tipo TaqMan a partir de muestras de hisopado nasofaríngeo que permitirán, en forma rápida, diferenciar tipos y subtipos del virus influenza en Perú. La utilidad de este método es que es rápido, y preciso en la detección simultánea de virus influenza, que la hace comparable al aislamiento en cultivo celular usado en virología clínica.
MATERIALES Y MÉTODOS
MUESTRAS CLÍNICAS, AISLAMIENTO VIRAL Y EXTRACCIÓN DE ARN
Las muestras clínicas correspondieron a la vigilancia epidemiológica de influenza y otros virus respiratorios en todo el país durante el año 2013. Dichas muestras fueron hisopados nasofaríngeos (n=300) que se tomaron según los criterios establecidos en la Directiva Sanitaria 045-2012-MINSA/DGE V.01 y fueron procesados en el Laboratorio de Virus Respiratorios del Instituto Nacional de Salud (INS) de Perú. Estas muestras se almacenaron a -80 ºC hasta la extracción de ARN.
El aislamiento viral se realizó en las líneas celulares: Madin-Darby Canine Kidney (MDCK ATCC – CCL- 34, para aislamiento de virus influenza), Vero ATCC® CCL81™ (aislado de riñón de Cercopithecus aethiops, para aislamiento de virus sincitial) y Human Epidermoid carcinoma strain #2 (Hep2 ATCC – CCL – 23, para aislamiento de adenovirus). Se observó la aparición del efecto citopático en las líneas celulares que indicaban replicación viral. Los aislamientos virales positivos fueron, posteriormente, analizados para la identificación viral por inmunofluorescencia directa (IFD) y RT-PCR singlepex. La extracción de ARN se realizó usando el kit comercial QIAamp® Viral RNA mini kit (Qiagen) y se almacenó a -80 ºC hasta el procedimiento de RT-PCR múltiple.
DISEÑO DE PRIMERS Y SONDAS
En la Tabla 1 se muestran los primers y sondas empleados para la detección de los virus influenza A, B y tipificación de los subtipos A (H1N1) pdm09, A (H3N2). Estos primers y sondas TaqMan han sido considerados de publicaciones previas, e incluyen un control interno que es el gen humano GAPDH (14,21). Los primers y sondas fueron sintetizadas por BioResearch (México).
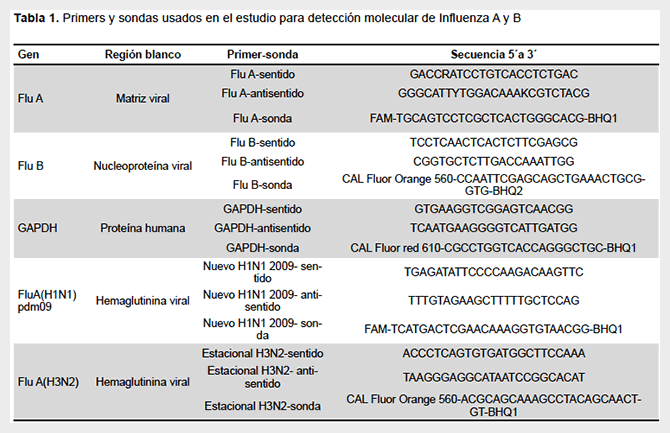
CONDICIONES DE LA RT-PCR MÚLTIPLE
El primer ensayo de RT-PCR empleó de primers y sondas correspondientes al gen de la matriz del virus influenza A, gen de la nucleoproteína del virus influenza B y el gen GAPDH de las células huésped como un control interno. El segundo ensayo utilizó primers y sondas específicas para hemaglutininas (H1 y H3) con el fin de tipificar los subtipos más destacados del virus influenza A (H1N1pdm09 y H3N2).
Simultáneamente, se realizó la retrotranscripción y PCR en tiempo real utilizando el kit Verso 1-step (Thermo Scientific). Los mix de RT-PCR (20 µL) comprendieron una mezcla de reacción de 15 µL [primers (0,25 µM sondas) 0,125 µM y 5µL de ARN], según condiciones del fabricante. La amplificación viral se realizó en el Termociclador RotorGene Q con las condiciones de transcripción reversa a 50 ºC x 30 min, seguida de la PCR con una activación inicial a 95 ºC × 15 min, 45 ciclos de una denaturación a 95 ºC × 15 s, anillamiento/ extensión a 60 ºC × 30 s. La adquisición y el análisis de los datos se realizaron utilizando el software del equipo RotorGene versión 6.0. En cada corrida se consideraron controles negativos y controles positivos para influenza A (H1N1) pdm09, influenza A (H3N2) e influenza B. Se consideró un Ct ≤35 como positividad al ensayo en las muestras de los pacientes.
EVALUACIÓN DE LÍMITE DE DETECCIÓN DE LA RTPCR MÚLTIPLE EN CONTROLES POSITIVOS
Se evaluó el parámetro de sensibilidad de la RT-PCR en tiempo real analizando el límite de detección de ARN viral. Se realizaron diluciones desde 10-1 a 10-6 de los ARN controles de virus influenza A, B y los subtipos A (H3N2) y A (H1N1) pdm09. Se utilizaron primers específicos para detectar secuencias blanco de cada virus. Las corridas de las muestras se realizaron por duplicado.
EVALUACIÓN DE REACCIÓN CRUZADA DE LA RTPCR MÚLTIPLE EN OTROS VIRUS
Se evaluó el parámetro de especificidad de la RT-PCR en tiempo real, analizando la posible reacción cruzada en otros virus ARN: adenovirus, parainfluenza (1,2 y 3), metapneumovirus, virus sincitial respiratorio, dengue (1,2,3 y 4) y VIH. Las corridas de las muestras se realizaron por duplicado.
EVALUACIÓN DE SENSIBILIDAD, ESPECIFICIDAD Y VALORES PREDICTIVOS DE LA RT-PCR MÚLTIPLE EN MUESTRAS CLÍNICAS
Se evaluaron los parámetros de sensibilidad y especificidad en 300 muestras de hisopado nasofaríngeo, de los cuales solo las muestras positivas a aislamiento fueron analizadas mediante la RT-PCR múltiple. Las corridas de las muestras se realizaron por duplicado. Se elaboró una tabla tetracórica para el cálculo de los parámetros de evaluación de la RT-PCR en tiempo real múltiple versus muestras de cultivo, con estos datos se hallaron los parámetros de sensibilidad, especificidad, valor predictivo positivo (VPP) y valor predictivo negativo (VPN).
RESULTADOS
EVALUACIÓN DE LA SENSIBILIDAD DE LA RT-PCR MÚLTIPLE EN CONTROLES POSITIVOS
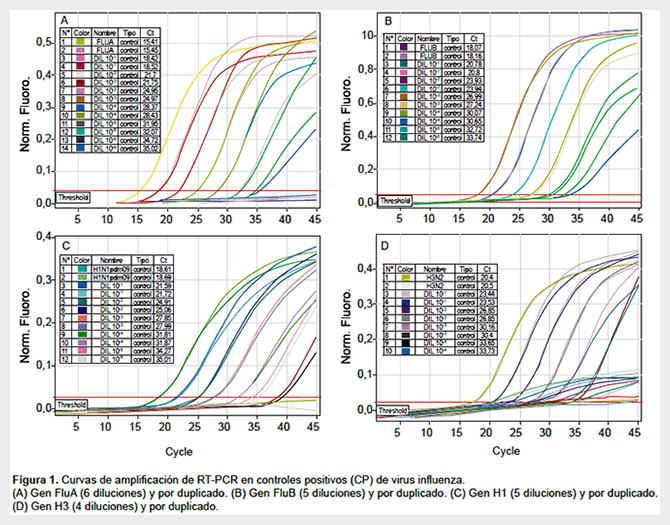
Los resultados de la sensibilidad de la RT-PCR múltiple mostraron los siguientes límites de detección: influenza A (9 copias/uL); influenza B (8 copias/uL); influenza A (H1N1) pdm09 (8 copias/uL), e influenza A (H3N2) con 7 copias/uL. Así mismo, se estableció el umbral (threshold) para cada subtipo del virus influenza mediante la curva de amplificación y la curva estandarizada con diluciones de cepas de los virus y el uso del software del equipo Rotor Gene Q versión 2.0.2. En la Figura 1 se muestran los Ct correspondientes a cada dilución versus la fluorescencia normalizada para la detección de cada virus influenza.
EVALUACIÓN DE LA ESPECIFICIDAD DE LA RT-PCR MÚLTIPLE EN OTROS VIRUS ARN
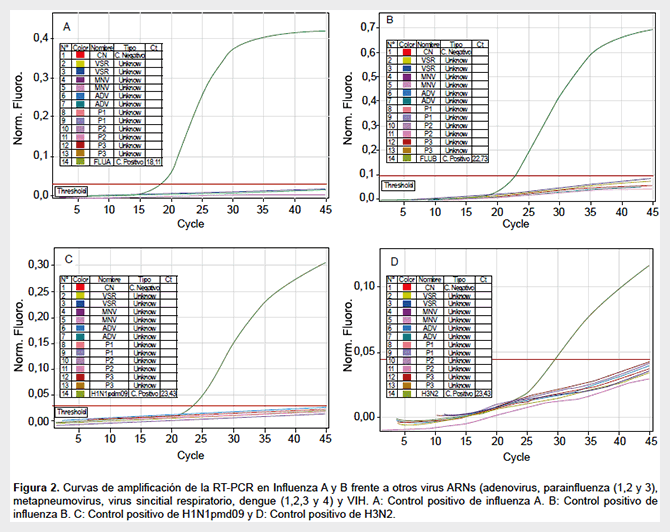
Los resultados de la especificidad de la RT-PCR múltiple no revelaron ninguna señal de amplificación cuando se analiza otros virus. Solamente se observó una señal positiva en los controles FluA (Ct=18,11), FluB (Ct=22,73), H1N1pdm09 (Ct=23,43), H3N2 (Ct=23,43) y en todas las muestras analizadas para el control humano GADPH (Ct=21,84). Las curvas de especificidad se muestran en la Figura 2.
EVALUACIÓN DE SENSIBILIDAD Y ESPECIFICIDAD DE LA RT-PCR MÚLTIPLE EN MUESTRAS CLÍNICAS
El aislamiento viral permitió la identificación de 109 cepas de virus influenza, las cuales fueron analizadas por RT-PCR y se identificaron 36 positivos a influenza A (H1N1) pdm09, 36 A (H3N2), 37 influenza B y 191 negativas a ambos virus (Tabla 2).

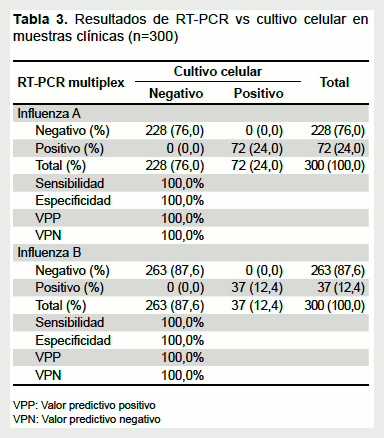
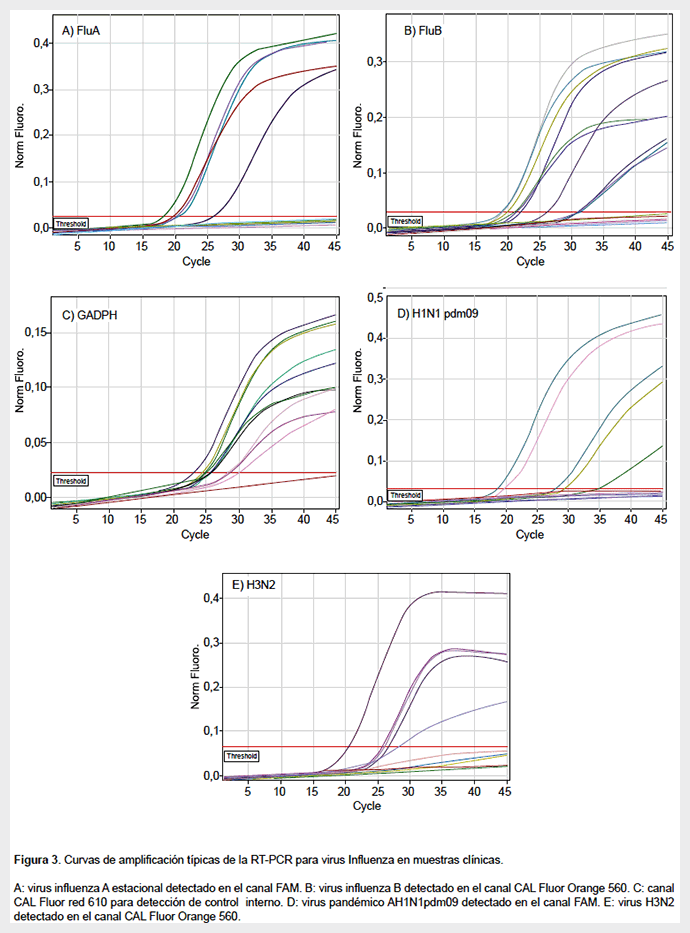
La comparación de la metodología RT-PCR múltiple con el cultivo celular (técnica Gold estándar) mostró una sensibilidad y especificidad del 100%. Los valores de sensibilidad y especificidad, según tipo de virus influenza o subtipo, estuvieron dentro del rango del intervalo de confianza de 95% (Tabla 3). Las curvas de amplificación de la RT-PCR en las muestras clínicas se observan en la Figura 3.
REPRODUCIBILIDAD DE LA RT-PCR MÚLTIPLE EN MUESTRAS CLÍNICAS
Las muestras se analizaron por duplicado, obteniéndose el mismo resultado para cada uno de los genes evaluados: FluA, H1N1, H3N2 y FluB, indicando un 100% de reproducibilidad de la RT-PCR múltiple.
DISCUSIÓN
El uso de metodologías moleculares es de gran ayuda en la actualidad para el diagnóstico de infecciones virales, ya que en solo horas podemos dar un diagnóstico rápido y posterior inicio de tratamiento (22,23). Su uso en las vigilancias epidemiológicas permite el análisis, interpretación y difusión de datos para la planificación, ejecución y evaluación de acciones en salud pública (24).
Las aplicaciones de PCR Múltiple benefician los diagnósticos en los laboratorios clínicos debido a su capacidad de detectar patógenos en una sola reacción de PCR en menos de 3 horas y hasta un panel de diez virus respiratorios (25).
Una de las principales limitaciones del aislamiento de los virus respiratorios en cultivos celulares, es el tiempo necesario de crecimiento e identificación que, por lo general, es de 4-7 días. Sin embargo, tiene la ventaja de detectar viabilidad de los virus presentes en las muestras, en cambio los métodos moleculares detectan ADN o ARN de microorganismos vivos o muertos (26).
Hay otros métodos capaces de detectar los virus respiratorios de modo más precoz, entre 1 y 3 días después de la inoculación de la línea celular y antes de la aparición del efecto citopático. El más común es el Shell vial, en el que las muestras son directamente centrifugadas sobre la monocapa celular para facilitar la adherencia y la penetración viral. Posteriormente, a las 24-48 horas se detecta la presencia de proteínas virales mediante inmunofluorescencia (27-29).
En este estudio se describe la estandarización de una RT-PCR múltiple para la detección de virus influenza A, B y la tipificación de los principales subtipos A (H1N1) pdm09 y A (H3N2), los cuales muestran una adecuada sensibilidad y especificidad. Cada ensayo representa un proceso de un solo paso, de modo que se evita contar con una PCR en tiempo real de varios pasos, es decir, para cada marcador viral. Otro punto muy importante es que a diferencia del PCR convencional múltiple, el cual es laborioso y presenta resultados falsos positivos, debido a la contaminación en el manejo de las muestras, el uso de sondas tipo TaqMan en un sistema de PCR en tiempo real múltiple es mucho más sensible y especifico, pero aun un poco más elevado en costos frente al método de PCR convencional (30).
Según el protocolo del Centers for Disease Control (CDC) emitido el 2009 para RT-PCR en la detección y caracterización del nuevo subtipo del virus de Influenza A (2009), se recomienda insumos y equipos que optimizan la prueba. El protocolo desarrollado es un múltiple RT-PCR in house que ha sido validado y optimizado tomando en consideración: robustez de los primers, sondas y enzima, límite de detección, repetitividad y reproducibilidad de la prueba. También se mejoró el ensayo a comparación de los RT-PCR múltiples citados por Suwannakarn et al. y Chen et al. reduciendo el volumen y componentes de la reacción, logrando reducciones costo-efectivas y mayor precisión comparado inclusive con una RT-PCR singleplex (32).
Los resultados de RT-PCR múltiple con el uso de sondas TaqMan, descritos anteriormente, son consistentes con estudios previos para la detección de diversos virus respiratorios. Proporcionan una sensibilidad y especificidad adecuadas (100%) en muestras de hisopado nasofaríngeo, siendo comparable con el PCR en tiempo real único (singleplex) para la detección del virus influenza (18,25,31-33). La estandarización de estos ensayos moleculares ayudará, en corto plazo, a la descentralización del diagnóstico de virus respiratorios en los laboratorios regionales que cumplan con la infraestructura y el equipamiento mínimo necesario.
En conclusión, la RT-PCR múltiple resultó ser un método rápido, sensible y específico para la detección simultanea de influenza A, B y la tipificación de los subtipos A (H1N1) pdm09, A (H3N2) en muestras de hisopado nasofaríngeo. Esta metodología constituye una herramienta muy útil en las muestras clínicas que podría ser usado sobre todo en los brotes estacionales, vigilancia y ensayos moleculares de rutina de los virus respiratorios más frecuentes en nuestro país.
Por otro lado, se hace necesario en nuestro país, el análisis completo de genomas de virus respiratorios para buscar nuevos targets que sirvan en la detección de los subtipos de influenza e identificar regiones de resistencia a medicamentos usados durante el tratamiento de los pacientes.
Agradecimientos: a la Dra. Josefina García por su apoyo en la revisión del manuscrito, y a la Blga. Merly Sovero por su apoyo en el soporte técnico.
Contribuciones de autoría: PM, MG y JA han participado en la concepción y diseño del artículo. Los procedimientos y resultados fueron realizados por PM, MH, NR, VG, SR y EG. Los análisis y discusiones fueron realizados por MG y PM. La redacción del artículo estuvo a cargo PM, MG y JA. La revisión crítica la realizó MG. La versión final estuvo a cargo de MG y PM. Todos aprobaron la versión final.
Fuentes de financiamiento: autofinanciado.
Conflicto de interés: los autores declaran no tener conflictos de interés.
REFERENCIAS BIBLIOGRÁFICAS
1. Oropeza Fernández S, Acosta Herrera B, Piñón Ramos A, Valdés Ramírez O, Savón Valdés C, Arencibia García A, et al. Diagnóstico molecular del virus influenza A (H1N1) 2009 y otros vírus respiratorios, durante la primera ola pandémica en Cuba. Rev Cubana Med Trop. 2011;63(2):147-54.
2. Talledo M, Zumaeta K. Los virus Influenza y la nueva pandemia A/H1N1. Rev peru biol. 2009;16(2): 227-38.
3. Hoffmann E, Stech J, Guan Y, Webster RG, Perez DR. Universal primer set for the full-length amplification of all influenza A viruses. Arch Virol. 2001;146(12):2275-89.
4. Gibbs AJ, Armstrong JS, Downie JC. From where did the 2009 ‘swine-origin’ influenza A virus (H1N1) emerge?. Virology J. 2009;6(1): 207. doi: 10.1186/1743-422X-6-207.
5. Salazar MI, López-Ortega O, León-Avila G, Ramírez-Gónzalez JE, Castro-Mussot ME. El origen de la variabilidad genética de los virus de la influenza. Gac Méd Méx. 2010;146(3):199-206.
6. Garten RJ, Davis CT, Russell CA, Shu B, Lindstrom S, Balish A, et al. Antigenic and genetic characteristics of swine-origin 2009 A (H1N1) influenza viruses circulating in humans. Science. 2009;325(5937):197-201. doi: 10.1126/science.
7. Itoh Y, Shinya K, Kiso M, Watanabe T, Sakoda Y, Hatta M, et al. In vitro and in vivo characterization of new swine-origin H1N1 influenza viruses. Nature. 2009;460(7258):1021-5. doi: 10.1038/nature08260.
8. Roth B, Mohr H, Enders M, Garten W, Gregersen JP. Isolation of influenza viruses in MDCK 33016PF cells and clearance of contaminating respiratory viruses. Vaccine. 2012;30(3):517-22. doi: 10.1016/j.vaccine.2011.11.063.
9. Leland DS, Ginocchio CC. Role of cell culture for virus detection in the age of technology. Clin Microbiol Rev. 2007;20(1):49-78. doi: 10.1128/CMR.00002-06.
10. Mancipe LF, Ramírez G, Jaime J, Vera V. Cultivos celulares como alternativa para el aislamiento y la producción de biológicos contra el Virus de Influenza. NOVA. 2011; 9(15).
11. Díez O, Batista N, Bordes A, Lecuona M, Lara M. Diagnóstico microbiológico de las infecciones del tracto respiratorio superior. Enferm Infecc Microbiol Clin. 2007;25(6):387-93.
12. Vallières E, Renaud C. Clinical and economical impact of multiplex respiratory virus assays. Diagn Microbiol Infect Dis. 2013;76(3):255-61. doi: 10.1016/j.diagmicrobio.2013.03.008.
13. Chi Hyun C, Chang Kyu L, Myung-Hyun N, Soo-Young Y, Chae Seung L,Yunjung C, et al. Evaluation of the AdvanSure™ real-time RT-PCR compared with culture and Seeplex RV15 for simultaneous detection of respiratory viruses. Diagn Microbiol Infect Dis. 2014;79(1):14-8.
14. Suwannakarn K, Payungporn S, Chieochansin T, Samransamruajkit R, Amonsin A, Songserm T, et al. Typing (A/B) and subtyping (H1/H3/H5) of influenza A viruses by multiplex real-time RT-PCR assays. J Virol Methods. 2008;152(1-2):25-31. doi: 10.1016/j.jviromet.2008.06.002.
15. Duchamp MB, Casalegno JS, Gillet Y, Frobert E, Bernard E, Escuret V, et al. Pandemic A (H1N1) 2009 influenza virus detection by real time RT‐PCR: is viral quantification useful?.Clin Microbiol Infect. 2010; 16(4):317-21. doi: 10.1111/j.14690691.2010.03169.x.
16. Xie Z, Pang YS, Liu J, Deng X, Tang X, Sun J, et al. A multiplex RT-PCR for detection of type A influenza virus and differentiation of avian H5, H7, and H9 hemagglutinin subtypes. Mol Cell Probes. 2006;20(3-4): 245-9.
17. Arvia R, Corcioli F, Ciccone N, Della Malva N, Azzi A. Detection of 12 respiratory viruses by duplex real time PCR assays in respiratory samples. Mol Cell Probes. 2015;29(6):408-13. doi: 10.1016/j.mcp.2015.08.006.
18. Khodadad N, Moattari A, Shamsi Shahr Abadi M, Kadivar MR, Sarvari J, Tavakoli F, et al. Prevalence of Influenza A (H1N1) pdm09 Virus Resistant to Oseltamivir in Shiraz, Iran, During 2012-2013. Jundishapur J Microbiol. 2015;29;8(8):e23690. doi: 10.5812/jjm.23690.
19. Radovanov J, Milošević V, Cvjetković IH, Ristić M, Djilas M, Nikolić N, et al., Influenza B viruses in the population of province of Vojvodina during the 2012/2013 season: Differentiation of B/Yamagata and B/Victoria lineages by real-tim RT-PCR, antigenic and phylogenetic characterization. Srp Arh Celok Lek. 2015;143(7-8):429-37.
20. Lucion MF, Juarez Mdel V, Viegas M, Castellano V, Romanin VS, Grobaporto M, et al. Virus respiratorio sincicial: Patrón clínico epidemiológico en niños internados en un hospital pediátrico durante los años 2000-2013.Arch Argent Pediatr. 2014;112(5):397-404. doi: 10.1590/S0325-00752014000500003.
21. Chen Y, Cui D, Zheng S, Yang S, Tong J, Yang D, et al. Simultaneous detection of influenza A, influenza B, and respiratory syncytial viruses and subtyping of influenza A H3N2 virus and H1N1 (2009) virus by multiplex real-time PCR. J Clin Microbiol, 2011;49(4):1653-6. doi: 10.1128/JCM.02184-10.
22. Read SJ, Burnett D, Fink CG. Molecular techniques for clinical diagnostic virology. J Clin Pathol. 2000; 53(7):502-6.
23. Cobo F. Application of molecular diagnostic techniques for viral testing. Open Virol J. 2012; 6(1):104-14.
24. Choi, B.C., The past, present, and future of public health surveillance. Scientifica, 2012. 2012. doi: 10.2174/1874357901206010104.
25. Parker J, Fowler N, Walmsley ML, Schmidt T, Scharrer J, Kowaleski J, et al. Analytical Sensitivity Comparison between Singleplex Real-Time PCR and a Multiplex PCR Platform for Detecting Respiratory Viruses.PloS one. 2015;10(11):e0143164. doi: 10.1371/journal.pone.0143164.
26. Hematian A, Sadeghifard N, Mohebi R, Taherikalani M, Nasrolahi A, Amraei M, et al. Traditional and modern cell culture in virus diagnosis. Osong Public Health Res Perspect. 2016;7(2):77-82. doi: 10.1016/j.phrp.2015.11.011.
27. Eiros JM, Ortiz de Lejarazu R, Tenorio A, Casas I, Pozo F, Ruiz G, et al. Diagnóstico microbiológico de las infecciones virales respiratorias. Enferm Infecc Microbiol Clin. 2009;27(3):168-77. doi: 10.1016/j.eimc.2008.03.004.
28. Jayakeerthi RS, Potula RV, Srinivasan S, Badrinath S. Shell Vial culture Assay for the rapid diagnosis of Japanese encephalitis, West Nile and Dengue-2 viral encephalitis.Virol J, 2006;3:2. doi: 10.1186/1743-422X-3-2.
29. Ribas Antúnez Mde L, Girón B, Monsalvez I, Morier L, Acosta G, Tejero Y, et al. Comparison of a modified shell vial culture procedure with conventional mouse inoculation for rabies virus isolation. Mem Inst Oswaldo Cruz. 2013;108(2):255-6.
30. Choudhary ML, Anand SP, Heydari M, Rane G, Potdar VA, Chadha MS, et al. Development of a multiplex one step RT-PCR that detects eighteen respiratory viruses in clinical specimens and comparison with real time RT-PCR.J Virol Methods. 2013;189(1):15-19. doi: 10.1016/j.jviromet.2012.12.017.
31. Nandhini G, Sujatha S. Epidemiology of influenza viruses from 2009 to 2013– A sentinel surveillance report from Union territory of Puducherry, India. Asian Pac J Trop Med, 2015;8(9):718-23. doi: 10.1016/j.apjtm.2015.07.028.
32. Li M, Xie Z, Xie Z, Liu J, Xie L, Deng X, et al. Simultaneous detection of four different neuraminidase types of avian influenza A H5 viruses by multiplex reverse transcription‐PCR using a GeXP analyser. Influenza Other Respir Viruses. 2016;10(2):141-9. doi: 10.1111/irv.12370.
33. Pretorius MA, Madhi SA, Cohen C, Naidoo D, Groome M, Moyes J, et al. Respiratory viral coinfections identified by a 10-plex real-time reverse-transcription polymerase chain reaction assay in patients hospitalized with severe acute respiratory illness-South Africa, 2009-2010. J Infect Dis. 2012;206 Suppl 1:S159-65. doi: 10.1093/infdis/jis538.
Correspondencia: Pool Marcos Carbajal
Dirección: Av. Defensores del Morro 2268, Chorrillos. Lima, Perú.
Teléfono: (+511) 748 0000 anexos 1428 o 1536
Correo electrónico:
pmarcos@ins.gob.pe
Recibido:
07/04/2016
Aprobado: 24/05/2017
En línea: 28/06/2017