Artículo Original
Diversidad genética de aislamientos peruanos de Leptospira spp. mediante electroforesis en gel de campo pulsado
Genetic diversity of peruvian isolates of Leptospira spp. through pulsed field gel electrophoresis
Paola Rivera1,a, Mónica Ticlla1,b, Lourdes Balda1,c, Dana Gonzalez1,d, Manuel Céspedes1,a
1 Laboratorio de Zoonosis Bacterianas, Instituto Nacional de Salud. Lima, Perú
a Biólogo magíster en Salud Pública; b biólogo magíster en Ciencias; c técnico de laboratorio; d biólogo
RESUMEN
Objetivos. Determinar la diversidad genética de aislamientos peruanos de Leptospira spp. mediante electroforesis en gel de campo pulsado (PFGE). Materiales y métodos. Se estandarizó la metodología de PFGE propuesta por Galloway y Levett (2008). Se elaboró una base de datos con los perfiles de PFGE de 65 cepas de referencia y se aplicó la técnica en 111 aislamientos de Leptospira spp. obtenidos en Perú entre 2002 y 2010. Resultados. Se determinó gran diversidad genética de serovares de Leptospira spp. circulantes en nuestro país. Se identificaron 57 serovares, 47 en 97 aislamientos patógenos. Los serovares más frecuentes fueron Icterohaemorrhagiae/Copenhageni (n=24) y Canicola (n=7). Las especies más frecuentes fueron L. santarosai (49,5%) y L. interrogans (37,1%). La distribución de especies, clusters y serovares varió según la fuente del aislamiento, el contexto ambiental y la procedencia. Conclusiones. Existe gran diversidad de serovares circulantes en el Perú, la cual está relacionada a la especie, el reservorio, el contexto ambiental y la procedencia del aislamiento. Se evidencia las relaciones genéticas y epidemiológicas entre aislamientos de diferentes fuentes, lo cual está relacionada a la especie, el reservorio, el contexto ambiental y la procedencia del aislamiento.
Palabras clave: Leptospira; Electroforesis en gel de campo pulsado; Variación genética (fuente: DeCS BIREME).
ABSTRACT
Objectives. Determine the genetic diversity of Peruvian isolations of Leptospira spp. through Pulsed Field Gel Electrophoresis (PFGE). Materials and methods. The PFGE methodology proposed by Galloway and Levett (2008) was standardized. A database including the PFGE profiles of 65 reference strains was prepared, and the technique was applied in 111 isolates of Leptospira spp. obtained in Peru between 2002 and 2010. Results. A great generic diversity of serovars of circulating Leptospira spp. was determined in our country. 57 serovars were identified, 47 out of 97 pathogen isolates. Most frequent serovars were Icterohaemorrhagiae/Copenhageni (n=24) and Canicola (n=7). The most frequent species were L. santarosai (49,5%) and L. interrogans (37,1%). The distribution of species, clusters and serovars changed according to the source of isolate, the environmental context and the origin. Conclusions. There is great diversity of circulating serovars in Peru. There are genetic and epidemiological relations among isolates of different sources, and this is related to species, reservoir, environmental context and the origin of the isolate.
Key words: Leptospira; Electrophoresis, gel, pulsed-field; Genetic variation (source: MeSH NLM).
INTRODUCCIÓN
La leptospirosis tiene una distribución mundial y afecta principalmente zonas tropicales con altos índices de precipitación (1). En el Perú, la incidencia anual de leptospirosis es 6,3 por millón de habitantes, cifras muy superiores a las reportadas en Venezuela, Paraguay, Colombia y Chile (2,3). En tanto, la incidencia en la región Loreto es de 72,7 lo cual refleja la endemicidad de la enfermedad en la zona. En el 2012, como consecuencia de las inundaciones, se han producido casos de hemorragia pulmonar grave y defunciones debido a leptospirosis (4).
La leptospirosis es una enfermedad febril aguda causada por espiroquetas patógenas del género Leptospira, transmitida al hombre por contacto directo o indirecto con orina de animales portadores (5). La unidad taxonómica del género es el serovar, según la prueba de aglutinación cruzada y absorción (CAAT). Los serovares relacionados antigénicamente constituyen serogrupos (6). Históricamente, el género Leptospira se dividió en dos especies: L. interrogans (cepas patógenas) y L. biflexa (cepas saprofíticas) (1). Actualmente el género Leptospira se clasifica en 20 genomospecies en base al estudio de hibridación del ácido desoxirribonucleico ADN-ADN. Estas, a su vez, se clasifican en especies patógenas, intermedias y saprofíticas mediante el secuenciamiento del gen 16S rARN (7,8).
La identificación de los serovares circulantes de Leptospira tiene importancia epidemiológica pues permite identificar fuentes de infección o reservorios, virulencia de cepa y distribución geográfica, ya que los serovares varían en las diferentes regiones y dependen de la ecología del medio que los alberga (5). Sin embargo, son pocos los laboratorios que cuentan con los recursos necesarios para mantener los esquemas de identificación serológica convencional: la CAAT y la prueba de microaglutinación (MAT), las cuales son técnicas que permiten identificar leptospiras a nivel de serovar y serogrupo respectivamente (6), y que, además, presentan un gran número de limitaciones, como la necesidad de tener una colección de cepas referenciales y sus respectivos antisueros, lo cual dificulta la identificación de la diversidad de leptospiras (8). No obstante y gracias al avance de técnicas de análisis de ADN se ha hecho posible el estudio de la epidemiología molecular de Leptospira spp. en perspectivas globales. Estas técnicas incluyen la digestión del ADN cromosómico con enzimas de restricción (REA), ribotipificación, análisis de polimorfismos de fragmentos de restricción (RFLP), electroforesis en gel por campo pulsado (PFGE) (9,10), entre otros.
La Electroforesis en gel de campo pulsado (PFGE) es el gold standard para la genotipificación de microorganismos (11) y permite la tipificación de aislamientos de Leptospira spp. a nivel de serovar (9,12). Además, proporciona información crucial para los estudios de epidemiología molecular ya que permite evidenciar relaciones genéticas entre aislamientos y así contribuir en la vigilancia e investigación de brotes.
En nuestro país son pocos los estudios epidemiológicos basados en métodos moleculares. Se ha evidenciado que mamíferos silvestres (roedores, marsupiales y murciélagos) son reservorios potenciales en la transmisión de leptospirosis en la Amazonía peruana (13,14), que las concentraciones de leptospiras virulentas en aguas superficiales urbanas son mayores que en las rurales, y que la especie L. interrogans prevalece en zonas urbanas, mientras que L. santarosai prevalece en zonas rurales (15). Asimismo, se ha reportado una nueva especie, Leptospira licerasiae serovar Varillal (7).Sin embargo, no se conoce la diversidad genética de la leptospira obtenidos de zonas endémicas. Por ello, el presente estudio tuvo como objetivo determinar la diversidad genética de 111 aislamientos de Leptospira spp. a través de PFGE, obtenidos en el Perú entre 2002 y 2010.
MATERIALES Y MÉTODOS
CEPAS REFERENCIALES DE LEPTOSPIRA
Se contó con un total de 65 cepas referenciales de Leptospira spp. proveídas por el Instituto Oswaldo Cruz (IOC-FioCruz); el Laboratorio de los Servicios Veterinarios Nacionales (NVSL), y los Centros para el Control y la Prevención de Enfermedades (CDC). Estos incluyeron seis especies patógenas (L. interrogans, L. kirschneri, L. noguchii, L. weilii, L. borgpetersenii y L. santarosai); dos intermedias (L. fainei y L. licerasiae), y dos saprofíticas (L. biflexa y L. meyeri) (los perfiles de PFGE están disponibles en Anexos en www.rpmesp.ins.gob.pe).
AISLAMIENTOS DE LEPTOSPIRA
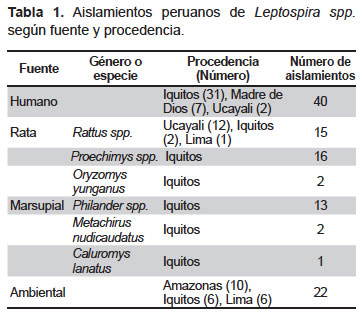
Se seleccionaron 111 aislamientos peruanos de Leptospira spp. (40 de humanos, 49 de animales y 22 ambientales) obtenidos del cepario del Laboratorio de Zoonosis Bacterianas del Instituto Nacional de Salud (INS) a partir del Programa de Vigilancia de Leptospira en el Perú (periodo 2002-2010). Solo se tomaron en cuenta los aislamientos de Leptospira spp. de fuente conocida (Tabla 1).

Los aislamientos de Leptospira spp. fueron conservados en medio semisólido Fletcher a temperatura ambiente y se mantuvieron viables en el tiempo mediante resiembras realizadas cada seis meses. Para su reactivación, estos fueron repicados en medio líquido Ellinghausen-McCullough-Johnson-Harris (EMJH) con 4% de suero de conejo e incubados a 28-30 ºC por un promedio de dos semanas. Se realizó un primer pasaje en medio líquido EMJH y se dejó crecer a 28-30 ºC por una semana. A partir de este se efectuó la prueba de 8-azaguanina y microaglutinación (MAT). Se hizo un segundo pasaje para proceder con la tipificación de los aislamientos mediante la técnica PFGE estandarizada.
Prueba de 8-azaguanina. Útil para diferenciar cepas saprofíticas y patógenas (16), se sembraron los aislamientos en EMJH líquido con 225 ug/mL de 8-azaguanina y se incubaron a 28- 30 ºC.
Prueba de microaglutinación (MAT). Se realizó según el manual de procedimientos de diagnóstico de Leptospira del Laboratorio de Zoonosis Bacterianas del INS (17). Se incubaron las cepas aisladas (antígenos) con un panel de serogrupos de Leptospira referenciales (antisueros de conejo contra antígenos específicos) y luego se examinaron con microscopio de campo oscuro para observar aglutinación. La MAT se llevó a cabo con los siguientes serogrupos: Andamana, Australis, Autumnalis, Ballum, Bataviae, Canicola, Celledoni, Cynopteri, Djasiman, Grippotyphosa, Hebdomadis, Icterohaemorrhagiae, Javanica, Mini, Panama, Pomona, Pyrogenes, Sejroe, Semaranga, Shermani, Tarassovi y Varillal. Títulos ≥ 800 fueron considerados significativos.
PFGE Y ANÁLISIS DE LOS PERFILES
La extracción de ADN y la PFGE se realizó según metodología descrita por Galloway y Levett (12) con ciertas modificaciones para su aplicación en el Laboratorio de Referencia Nacional de Zoonosis Bacterianas. Así mismo, se utilizaron las fórmulas y procedimientos estandarizados por la Red PulseNet USA para la preparación de reactivos para PFGE (18).
Cepas bacterianas. Se centrifugaron 8 mL de cada cultivo a 4500 rpm por 20 min. Luego fueron resuspendidas en 1mL de buffer de suspensión (BS) y centrifugadas a 4500 rpm por 10 min por dos veces. Se resuspendieron las células en BS a una DO de 0,600-0,900 a 610 nm.
Preparación de los bloques de agarosa. Se tomaron 100 μL de cada suspensión celular y se adicionaron 5 uL de proteinasa K (20 mg/mL). Se tomaron 100 uL de agarosa (1% SeaKem Gold agarose, 1% sodium dodecyl sulfate SDS en buffer Tris-EDTA) y se mezclaron con el mix bacteriano. El mix se puso en moldes y se dejó solidificar a temperatura ambiente por 15 min.
Extracción de ADN. Se empleó una concentración final de proteinasa K de 0,53 mg/mL, para lo cual se sumergieron los bloques de agarosa en 1,5 mL de buffer de lisis (50mM Tris-HCL, 50 mM EDTA, pH 8,0; 1% N-lauroylsarcosine sodium salt) con 40 uL de proteinasa K (20 mg/mL) y fueron incubados en baño maría a 54 ºC por dos horas. Luego cada bloque fue transferido a un tubo cónico que contenía 10 mL de agua estéril previamente calentada y lavado dos veces en baño maría a 50 ºC por 10 min. Los lavados fueron repetidos cuatro veces usando buffer de lavado TE (10 mM Tris, 1 mM EDTA, pH 8,0). Los bloques de agarosa fueron almacenados en 5 mL de buffer TE a 4ºC.
Digestión de restricción. Se cortó una parte de cada bloque y se digirió con 30 U de enzima de restricción endonucleasa Not I (Fermentas, BioRad) en baño maría a 37 ºC por seis horas. La Salmonella serotipo Braenderup H9812 fue digerida con 50U de Xba I para su uso como marcador estándar (19).
Electroforesis en gel de campo pulsado. Se colocaron los bloques de agarosa que contenían el ADN digerido, en los pocillos del gel agarosa 1% (SeaKem Gold en buffer TBE 0,5x). Se realizó la corrida utilizando el equipo CHEF – MAPPER (Bio-Rad Laboratories) por 18 h a 14 ºC con 2,3 L de buffer TBE 0,5x recirculante y bajo las siguientes condiciones: tiempo inicial de 2,16 s, tiempo final de 35,07 s, ángulo de 120º y gradiente de 6 V/cm. Se tiñeron los geles con bromuro de etidio (1ug/mL) por 20 min, seguidos por uno o dos ciclos de 20 min de desteñido en agua y se documentaron con el Bio-Rad Gel Doc 2000. Para los aislamientos no tipificables por PFGE, se repitió el procedimiento y se agregó tiourea al buffer de corrida en una concentración final de 100 μM, para lo cual se utilizó el protocolo propuesto por Ribeiro et al. (20).
Análisis de los perfiles de PFGE. Las imágenes de los geles fueron analizadas mediante el programa GelCompar II. Los dendogramas fueron creados utilizando el análisis de agrupamiento UPGMA basado en el coeficiente de similitud de bandas con una optimización de 1,5% y tolerancia de posición de 1,0%. La base de datos con los perfiles de PFGE de las 65 cepas referenciales de Leptospira spp. sirvió como una biblioteca de búsqueda para la comparación e identificación de serovares de los aislamientos estudiados. Se comparó con los resultados obtenidos por Galloway y Levett y el IOC-Fiocruz para las cepas referenciales (12,21), y por Ganoza et al. y Matthias et al. para algunos de nuestros aislamientos (7,15).
Criterios de agrupamiento e interpretación. Las clonas genéticas (clusters) representan aislamientos que se asocian formando grupos en el dendograma (22). Asimismo, los perfiles de PFGE que no difirieron entre ellos en más de tres bandas fueron considerados un mismo serovar (12) y a partir de ellos se evaluó la diversidad genética de la población (H) y el poder de discriminación de la técnica (D). Los patrones PFGE con un número menor a cinco bandas no fueron considerados en el dendograma y fueron designados no tipificables.

DIVERSIDAD GENÉTICA (H) Y EVALUACIÓN DE LA TÉCNICA
Para evaluar la diversidad genética se utilizó la fórmula de Nei (23) (Figura 1). Mientras el valor se aproxime más a 1 la diversidad genética será mayor.
La técnica se evaluó a través de tres indicadores: i) Poder de tipificación: determinación del porcentaje de aislamientos a los que se les pudo asignar un tipo específico sin ambigüedad. ii) Reproducibilidad: se corrió una cepa referencial en cada gel de corrida. iii) Poder de discriminación: se usó el índice de diversidad de Simpson (D) para métodos de tipificación bacterial. Mientras el valor se aproxime más a 1, la capacidad de la técnica de distinguir entre dos cepas no relacionadas será mayor (24) (Figura 2).
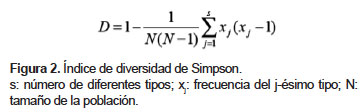
RESULTADOS
Según la prueba de 8-azaguanina, todos los aislamientos de humanos y animales fueron clasificados como patógenos; de los aislamientos ambientales, 14 fueron saprofíticos. Por medio de la MAT, 81 ais-lamientos (74,8%) reaccionaron a títulos altos con el panel de serogrupos referenciales, 12 (10,8%) reaccionaron a títulos bajos y 16 (14,4%) no reaccionaron.
ELECTROFORESIS EN GEL DE CAMPO PULSADO
A partir de los 111 aislamientos se generaron 107 patrones de bandas (cuatro cepas no fueron tipificables). La digestión de restricción de ADN con la enzima Not I resultó en 5-25 bandas, con un promedio de 14.
Análisis de las clonas genéticas. No se incluyeron los perfiles de PFGE de los aislamientos saprofíticos (n=10) ya que formaban grupos externos de lejana relación genética con respecto a los aislamientos patógenos (n=97) y a la vez no se asociaban con ninguna cepa referencial. Las clonas patógenas identificadas se muestran en la Tabla 2.
En el dendograma (disponible en Anexos en www.rpmesp.ins.gob.pe) se puede observar la formación de tres clusters importantes (I, II y III). El cluster I forma un grupo externo y se asocia con cepas referenciales de la especie L. noguchii, identificándose el serovar Proechimys en tres aislamientos. El cluster II se asocia con cepas referenciales de la especie L. interrogans (n=36) y se divide en tres grupos (IIA, IIB y IIC). El cluster IIA incluye cinco aislamientos de serovares desconocidos pero identificados como serogrupo Bataviae por MAT. El cluster IIB incluye 24 aislamientos pertenecientes al serovar Icterohaemorrhagiae/Copenhageni; y el cluster IIC incluye siete aislamientos humanos pertenecientes al serovar Canicola. El cluster III se subdivide en cinco grupos (IIIA-IIIE). El cluster IIIA incluye dos aislamientos humanos pertenecientes al serovar Varillal de la especie L. licerasiae. Los clusters IIIB, IIID y IIIE están conformados por un solo aislamiento respectivamente y no se asocian con ninguna cepa referencial. El cluster IIIC se asocia con las cepas referenciales de la especie L. santarosai (n=48) y se subdivide a su vez en cinco subgrupos (IIIC1 – IIIC5). El subgrupo IIIC1 incluye siete aislamientos de serovares desconocidos pero identificados como serogrupo Sejroe, en reacción cruzada con Hebdomadis, por MAT. El subgrupo IIIC2 está conformando por 16 aislamientos, dentro de los cuales se incluyen cinco aislamientos ambientales de serovar desconocido (ser-7). El subgrupo IIIC4 incluye nueve aislamientos, dentro de los cuales se puede identificar al serovar Borincana en un aislamiento. Tanto el subgrupo IIIC3 como el subgrupo IIIC5 incluyen aislamientos de serovares desconocidos.
Análisis de los serovares. Se encontraron 57 serovares (47 patógenos y 10 saprofíticos) con un coeficiente de similitud ≥ a 78,4%. El análisis con la base de datos referencial de PFGE permitió identificar 37/107 de los aislamientos a nivel de serovar. El más frecuente fue Icterohaemorrhagiae/Copenhageni.
ANÁLISIS GENÉTICO DE LOS AISLAMIENTOS PATÓGENOS
Aislamientos ambientales. El 87,5% (7/8) se asoció con L. santarosai y uno con una especie no identificada, probablemente intermedia ya que forma un cluster con L. licerasiae. El cluster genético predominante fue el III-C2 en el 62,5%, identificándose un mismo serovar (ser-7) en cinco aislamientos, cuatro de ellos procedentes de Alto Tuntus-Amazonas y uno de Varillal-Iquitos.
Aislamientos en marsupiales. Los marsupiales de los géneros Philander, Caluromys y Metachirus se agruparon con la especie L. santarosai (12/16), L. noguchii (3/16) y L. interrogans (1/16) con predominio del cluster III-C2 (37,5%). Se identificaron 14 serovares, encontrándose en su mayoría un aislamiento para cada uno de estos.
Aislamientos en ratas. Se agruparon con la especie L. interrogans (16/33), L. santarosai (12/33), L. noguchii (4/33) y una especie no identificada. El serovar predominante fue Icterohaemorrhagiae/Copehnageni (48,5%), el cual se asoció en su mayoría a roedores del género Rattus (93,7%) y a uno del género Proechimys. L. santarosai se asoció a roedores del género Proechimys y Oryzomys, y L. noguchii y una especie no identificada solo se asociaron a roedores del género Proechimys, identificándose el serovar Proechimys en dos aislamientos.
Aislamientos en humanos. Se agruparon con la especie L. interrogans (19/40), L. santarosai (17/40), L. licesariae serovar Varillal (2/40), L. noguchii serovar Proechimys (1/40) y una especie no identificada; con predominio de los serovares Icterohaemorrhagiae/Copehnageni (20,0%) y Canicola (17,5%).
ANÁLISIS SEGÚN PROCEDENCIA
El número de aislamientos patógenos de Amazonas (n=6) y Lima (n=2) fue muy bajo para asociarlo al departamento, en ambos casos los resultados se asociaron a la fuente. En el caso de Madre de Dios (n=7), todos los aislamientos fueron de humanos y se asociaron con las especies L. santarosai (n=6) y L. interrogans serovar Canicola (n=1), con predominio de los clusters III-C3 (n=3) y III-C4 (n=3), e identificándose el serovar Borincana en un aislamiento. Asimismo, todos los aislamientos de ratas de Ucayali (12/14) y 1 de humano pertenecieron al serovar Icterohaemorrhagiae/Copenhageni L. interrogans; el otro aislamiento humano se asoció con L. santarosai cluster III-C3.
Los aislamientos de Loreto - Iquitos (n=68) se ubicaron en 12 de las 13 clonas genéticas descritas, siendo los de mayor proporción el cluster III-C2 (17,6%) y el cluster II-B serovar Icterohaemorrhagiae/Copenhageni (14,7%). Estos se agruparon en su mayoría con las especie L. santarosai (51,5%) y L. interrogans (30,9%). Los serovares predominantes en aislamientos humanos fueron Icterohaemorrhagiae/Copenhageni (22,6%), Canicola (19,4%), ser-5 serogrupo Sejroe, en reacción cruzada con Hebdomadis (12,9%) y ser-39 serogrupo Bataviae (9,7%). Cabe resaltar que los clusters III-C2 y III-C3 de L. santarosai reunieron aislamientos de las cuatro fuentes del estudio, y del mismo modo el cluster III-C3 reunió aislamientos de 4/5 departamentos del estudio.
DIVERSIDAD GENÉTICA (H) Y EVALUACIÓN DE LA TÉCNICA
El índice de diversidad genética de Nei para toda la población fue 0,95 y para los aislamientos de Iquitos fue de 0,98.
El poder de tipificación de la técnica para todos los aislamientos de Leptospira spp. fue de 96,4% (cuatro aislamientos saprofíticos no fueron tipificables). El poder de tipificación solo para los aislamientos patógenos fue de 100%, y para los aislamientos saprofíticos fue de 71,4% siendo necesario el uso de tiourea para el 70% de estos. En relación a la reproducibilidad, las ocho cepas referenciales incluidas en las corridas dieron los mismos perfiles que los obtenidos en la base de datos (coeficiente de similitud de 100%). El poder de discriminación de la técnica de PFGE aplicada a nuestra muestra de aislamientos de Leptospira spp. fue de 0,99.
DISCUSIÓN
La distribución de especies de Leptospira varió según la fuente del aislamiento. Los aislamientos ambientales se asociaron en su mayoría con L. santarosai y con una especie no identificada probablemente intermedia. Es importante resaltar la identificación de un serovar (ser-7) en 5/8 muestras ambientales, aislado en su mayoría de zonas rurales. Nuestros resultados difieren de los reportados por Ganoza et al. quienes no identificaron L. santarosai en muestras de agua de zonas rurales. Sin embargo, coincidimos en la identificación de L. santarosai en aislamientos urbanos y de especies intermedias en aislamientos rurales (15).
Similar a otras investigaciones (15,25), se identificó el serovar Icterohaemorrhagiae/Copenhageni L. interrogans como el más común en los roedores del género Rattus. Asimismo, se identificaron las especies L. santarosai y L. noguchii en ratas espinosas (Proechimys spp.), y L. santarosai en ratas arroceras (Oryzomys yunganus). Ambas especies han sido previamente aisladas a partir de ratas espinosas en Panamá (26). La identificación de L. santarosai, L. noguchii y L. interrogans ya ha sido previamente descrita en marsupiales peruanos del género Philander (26). La gran diversidad de serovares identificados en el presente estudio sostiene lo enunciado por Liceras y Zulzer, quienes identificaron seis nuevos serovares en marsupiales (27).
La prevalencia de L. interrogans y L. santarosai en aislamientos humanos coincide con lo descrito por Ganoza et al. (15), quienes además indicaron que L. interrogans estuvo mayormente asociada a zonas urbanas y periurbanas, mientras que L. santarosai, L. noguchii y otras especies a zonas rurales, resultados que también coinciden con los nuestros. Además, se identificó L. licerasiae serovar Varillal, la cual es una reciente especie aislada por Matthias et al. en aislamientos humanos y de roedores en la Amazonía peruana (7). El serovar Icterohaemorrhagiae/Copenhageni L. interrogans, al igual que en los roedores, fue el serovar más común, seguido del serovar Canicola, sugiriendo que las ratas y los perros son hospederos de leptospiras de importancia en nuestro país. La identificación de una gran diversidad de serovares L. santarosai indica, además, la existencia de otras fuentes potenciales de transmisión como ratas espinosas, marsupiales y fuentes ambientales como aguas y suelos contaminados con leptospiras patógenas, reservorios descritos previamente por algunos autores (13,15,27). En la Amazonia peruana, la Leptospira también ha sido identificada en diversas especies de mamíferos de vida salvaje como murciélagos (14), sajinos (Tayassu tajacu) (28) y primates (Potos flavus) (13).
De acuerdo a la distribución por procedencia se identificaron las especies L. interrogans y L. santarosai en Lima, Iquitos, Madre de Dios y Ucayali. En Iquitos, además, se identificaron L. noguchii, L. licerasiae y otras especies desconocidas. En Amazonas se identificó L. santarosai y una especie desconocida, probablemente por tratarse de aislamientos ambientales. En Madre de Dios no se identificó el serovar Icterohaemorrhagiae a pesar de tratarse de aislamientos humanos, lo cual estaría de acuerdo con lo reportado por Céspedes et al., quienes en un estudio de seroprevalencia en la zona no hallaron anticuerpos contra el serogrupo Icterohaemorrhagiae (29). La gran diversidad de leptospiras patógenas en la Amazonia peruana es la única que ha sido estudiada mediante métodos moleculares en nuestro país (14,15); sin embargo, estos estudios solo se basaron en la identificación de genomospecies mediante el secuenciamiento del gen 16S rRNA. En el presente estudio se presenta la diversidad de serovares de Leptospira spp. mediante PFGE, la cual fue mayor en L. santarosai, e identificamos dos clusters patógenos de importancia: los serogrupos Bataviae L. interrogans y Sejroe L. santarosai.
Los resultados de la MAT coincidieron, en su mayoría, con los obtenidos por PFGE a nivel de serogrupo; sin embargo, en algunos aislamientos se observó reacción cruzada entre serogrupos de cercana relación genética, y entre algunos clusters y determinados serogrupos. Como los resultados de ambas técnicas se complementan se recomienda el uso de ambas en la identificación de serovares de Leptospira.
Aunque nuestra base de datos no contiene todos los serovares de Leptospira existentes, esta nos permitió identificar los serovares de Leptospira más comunes, como Icterohaemorrhagiae/Copenhageni y Canicola de la especie L. interrogans. La diversidad genética para el total de aislamientos estudiados fue de 0,95, lo cual confirma la elevada diversidad genética de variedades de Leptospira spp. circulantes en nuestro país. Esta alta diversidad genética de Leptospira indica una gran capacidad de generar nuevos genotipos mediante mutación al azar o fenómenos de recombinación genética y, en consecuencia, una mejor capacidad de adaptación a diferentes condiciones ambientales (30).
Desde el punto de vista patogénico, la leptospirosis parece variar según el contexto ambiental y biológico. Si bien es cierto, distintas especies de Leptospira producen síndrome febril, L. interrogans (serovares Icterohaemorrhagiae y Canícola) ha sido asociado a zonas urbanas y a formas más graves de la enfermedad como hemorragia pulmonar y muerte (15,31). En nuestro estudio, estos dos serovares fueron identificados como los predominantes, por lo que la implementación de medidas de prevención y control enfocadas en ratas y perros tendría un gran impacto en la salud pública del país.
La técnica de PFGE demostró un alto poder de tipificación, reproducibilidad y poder de discriminación. La mayoría de las cepas saprofíticas mostraron degradación de ADN, por lo que fue necesario el uso de tiourea para mejorar los resultados (20); sin embargo, muchos de ellas no produjeron un número suficiente de bandas que permitieran una caracterización confiable y otras no pudieron ser tipificadas. Es necesario esclarecer que el poder de tipificación de estas cepas no está relacionado con la técnica sino más bien a la condición de ser saprofíticas, premisa que no ha sido previamente descrita. Asimismo, aunque la técnica de PFGE es el gold estándar para realizar subtipificación molecular de bacterias (9,11,12); se recomienda que estos resultados sean comparados con los de otras técnicas de subtipificación molecular para nuestra misma población.
Una de las limitaciones que tiene la técnica es la de no poder diferenciar entre los serovares Icterohaemorrhagiae y Copenhageni; sin embargo, su cercana relación genética y la incapacidad de las técnicas serológicas y moleculares para diferenciarlas ya han sido reportadas previamente (32,33). Asimismo, la PFGE solo permite identificar especies en caso de que el perfil del aislamiento se encuentre en la base de cepas referenciales. De no ser así, la técnica nos sugiere una determinada especie por la manera en que se asocia con sus cepas de referencia, ya que el poder de discriminación de esta corresponde a la dupla especie-serovar (12). Este hecho, junto a los resultados obtenidos en estudios previos en nuestro país, nos sugiere la distribución de genomospecies descritas en el presente estudio.
El avance de los métodos moleculares ha tenido gran impacto en la vigilancia epidemiológica de nuestro país, la cual hasta hace pocos años se basaba en estudios de seroprevalencia, y es precisamente gracias a ellos que se ha identificado la diversidad de variedades, reservorios y relaciones entre el medioambiente, los hospederos y los serovares de Leptospira. Sin embargo, los métodos de tipificación molecular solo son posibles tras el aislamiento de la bacteria y la obtención de cultivos puros, requisitos que solamente son posibles en poblaciones con alta prevalencia de la enfermedad y tras la vigilancia y seguimiento de casos.
En conclusión, los resultados presentados aquí dan soporte a los estudios de epidemiología molecular previos realizados en nuestro país y, además, permiten evidenciar las relaciones genéticas y epidemiológicas entre aislamientos humanos, de roedores, marsupiales y ambientales patógenos, así como la gran diversidad de serovares circulantes, la cual está estrechamente relacionado a la especie, el reservorio, el contexto ambiental y la procedencia del aislamiento. Aplicaciones futuras de esta técnica requerirán la expansión del número de serovares incluidos en la base de datos. Así mismo, será necesario la confirmación o identificación de las especies a las cuales pertenecieron los aislamientos mediante el secuenciamiento del gen 16S rRNA o la técnica de MLST.
Agradecimientos: a la Dra. Renee Galloway (CDC) por sus recomendaciones. Al Blgo. Henri Bailón por sus sugerencias y apoyo técnico en el uso del programa Gel Compar II. Al personal del Laboratorio de Zoonosis Bacterianas del INS por el apoyo logístico brindado.
Contribuciones de autoría: PR, MT y MC participaron en la concepción y diseño del trabajo. PR y LB en la recolección de datos. PR en el análisis e interpretación de datos y redacción del manuscrito. MT y MC realizaron la asesoría técnica o administrativa, DQ y MC obtuvieron el financiamiento, MC realizó la revisión crítica y la aprobación final del artículo.
Fuentes de financiamiento: Instituto Nacional de Salud.
Conflictos de interés: los autores declaran no tener conflictos de interés en la publicación de este artículo.
Referencias Bibliográficas
1. Johnson RC, Faine S. Leptospira, En: Krieg NR, Holt JG. Bergey’s manual of systematic bacteriology. Vol. 1. Baltimore: Williams & Wilkins; 1984. p. 62-7.
2. Céspedes M, Tapia R, Balda L, Gonzales D, Ticlla M. Informe de situación de la leptospirosis en el Perú 2007 [Internet]. Lima: Instituto Nacional de Salud; 2006 [citado el 8 de febrero del 2012]. Disponible en: http://www.ins.gob.pe/repositorioaps/0/4/jer/-1/Leptopirosis/InformesituaciondeleptospirosisPer%C3%BA2007.pdf
3. Pappas G, Papadimitriou P, Siozopoulou V, Christou L, Akritidis N. The globalization of leptospirosis: worldwide incidence trends. Int J Infect Dis. 2008;12(4):351-7.
4. Ministerio de Salud del Perú, Dirección Regional de Salud Loreto. Situación de la Leptospirosis en Loreto, a la S.E 22 del 2012. Informe ejecutivo [Internet]. Loreto: MINSA; 2012 [citado el 8 de febrero del 2012]. Disponible en: http://www.diresaloreto.gob.pe/portal/files/Informe_Leptospirosis_22.pdf
5. Levett PN. Leptospirosis. Clin Microbiol Rev. 2001;14(2):296-326.
6. Dikken H, Kmety E. Serological typing methods of leptospires. En: Bergan T, Norris JR. Methods in Microbiology. Vol.11. New York: Academic Press; 1978. p. 259-307.
7. Matthias MA, Ricaldi JN, Cespedes M, Diaz MM, Galloway RL, Saito M, et al. Human leptospirosis caused by a new, antigenically unique Leptospira associated with a rattus species reservoir in the Peruvian Amazon. PLoS Negl Trop Dis. 2008;2(4):e213.
8. Slack AT, Khairani-Bejo S, Symonds ML, Dohnt MF, Galloway RL, Steigerwalt AG, et al. Leptospira kmetyi sp. nov., isolated from an environmental source in Malaysia. Int J Syst Evol Microbiol. 2009;59(Pt 4):705-8.
9. Herrmann JL, Bellenger E, Perolat P, Baranton G, Saint Girons I. Pulsed-field gel electrophoresis of NotI digests of leptospiral DNA: a new rapid method of serovar identification. J Clin Microbiol. 1992;30(7):1696-702.
10. Herrmann JL, Baril C, Bellenger E, Perolat P, Baranton G, Saint Girons I. Genome conservation in isolates of Leptospira interrogans. J Bacteriol. 1991;173(23):7582-8.
11. Tenover FC, Arbeit RD, Goering RV, Mickelsen PA, Murray BE, Persing DH, et al. Interpreting chromosomal DNA restriction patterns produced by pulsed-field gel electrophoresis: criteria for bacterial strain typing. J Clin Microbiol. 1995;33:2233-9.
12. Galloway RL, Levett PN. Evaluation of a modified pulsed-field gel electrophoresis approach for the identification of Leptospira serovars. Am J Trop Med Hyg. 2008;78(4):628-32.
13. Bunnell JE, Hice CL, Watts DM, Montrueil V, Tesh RB, Vinetz JM. Detection of pathogenic Leptospira spp. infections among mammals captured in the Peruvian Amazon basin region. Am J Trop Med Hyg. 2000;63(5-6):255-8.
14. Matthias MA, Díaz MM, Campos KJ, Calderon M, Willig MR, Pacheco V, et al. Diversity of bat associated Leptospira in the peruvian amazon inferred by bayesian phylogenetic analysis of 16s ribosomal DNA sequences. Am J Trop Med Hyg. 2005;73(5):964-74.
15. Ganoza CA, Matthias MA, Collins-Richards D, Brouwer KC, Cunningham CB, Segura ER, et al. Determining risk for severe leptospirosis by molecular analysis of environmental surface waters for pathogenic Leptospira. PLoS Med. 2006;3(8):e308.
16. Hartskeerl R, Smits H, Korver H, Terpstra W. International course on laboratory methods for the diagnosis of Leptospirosis. Netherlands: Royal Tropical Institute Department of Biomedical Research; 2001.
17. Instituto Nacional de Salud. Manual de procedimientos bacteriológico y serológico para el diagnostico de la Leptospirosis. Lima: Instituto Nacional de Salud; 2002.
18. Centers for Disease Control and Prevention. Quality assurance (QA)/quality control (QC) manual for the standardized pulsed-field gel electrophoresis (PFGE) technique used by PulseNet Laboratories for foodborne disease surveillance. Atlanta: CDC; 2005.
19. Hunter SB, Vauterin P, Lambert-Fair MA, Van Duyne MS, Kubota K, Graves L, et al. Establishment of a universal size standard strain for use with the PulseNet standardized pulsed-field gel electrophoresis protocols: converting the national databases to the new size standard. J Clin Microbiol. 2005;43(3):1045-50.
20. Ribeiro RL, Machry L, Brazil JM, Ramos TM, Avelar KE, Pereira MM. Technical improvement to prevent DNA degradation of Leptospira spp. in pulsed field gel electrophoresis. Lett Appl Microbiol. 2009;49(2):289-91.
21. Machry L, Ribeiro RL, Brazil JMV, Balassiano IT, Oliveira ICM, Avelar KES, et al. Caracterização de cepas de referência de Leptospira sp utilizando a técnica de pulsed field gel electrophoresis. Rev Soc Bras Med Trop. 2010;43(2):166-9.
22. Maslow JN, Mulligan ME, Arbeit RD. Molecular epidemiology: application of contemporary techniques to the typing of microorganisms. Clin Infect Dis. 1993;17(2):153-62.
23. Nei M, Tajima F. Genetic drift and estimation of effective population size. Genetics. 1981;98(3):625-40.
24. Hunter PR. Reproducibility and indices of discriminatory power of microbial typing methods. J Clin Microbiol. 1990;28(9):1903-5.
25. Galloway RL, Levett PN. Application and validation of PFGE for serovar identification of Leptospira clinical isolates. PLoS Negl Trop Dis. 2010;4(9):e824.
26. Faine S, Adler B, Bolin C, Perolat P. Leptospira y Leptospirosis. 2da ed. Melboure, Australia: MediSci; 1999.
27. Liceras de Hidalgo JL, Sulzer KR. Six new leptospiral serovars isolated from wild animals in Peru. J Clin Microbiol. 1984;19(6):944-5.
28. Jori F, Galvez H, Mendoza P, Cespedes M, Mayor P. Monitoring of leptospirosis seroprevalence in a colony of captive collared peccaries (Tayassu tajacu) from the Peruvian Amazon. Res Vet Sci. 2009;86(3):383-7.
29. Céspedes M, Ormaeche M, Condori P, Balda L, Glenny M. Prevalencia de leptospirosis y factores de riesgo en personas con antecedentes de fiebre en la provincia de Manu, Madre de dios, Perú. Rev Peru Med Exp Salud Publica. 2003;20(4):180-5.
30. Betancor L, Gadea MP, Flores K. Genética Bacteriana. En: Instituto de Higiene, Facultad de Medicina (UDELAR). Temas de Bacteriología y Virología Médica. 3ra Ed. Montevideo: Oficina del Libro FEFMUR; 2008. p. 65-90.
31. de Faria MT, Calderwood MS, Athanazio DA, McBride AJ, Hartskeerl RA, Pereira MM, et al. Carriage of Leptospira interrogans among domestic rats from an urban setting highly endemic forleptospirosis in Brazil. Acta Trop. 2008;108(1):1-5. 32. Tamai T, Sada E, Kobayashi Y. Restriction endonuclease DNA analysis of Leptospira interrogans serovars Icterohaemorrhagiae and Copenhageni. Microbiol Immunol. 1988;32(9):887-94.
33. Majed Z, Bellenger E, Postic D, Pourcel C, Baranton G, Picardeau M. Identification of variable-number tandem-repeat loci in Leptospira interrogans sensu stricto. J Clin Microbiol. 2005;43(2):539-45.
Correspondencia: Paola Andrea Rivera Ramirez
Dirección: Av. Defensores del Morro 2268, Lima 9, Perú
Teléfono: (511) 617-6200 anexo 1429
Correo electrónico: p_rivera_ramirez@hotmail.com
Recibido: 03-05-12
Aprobado: 31-10-12