
ARTÍCULO ORIGINAL
Multidrogorresistencia de Salmonella Infantis en Perú: un estudio mediante secuenciamiento de nueva generación
Multidrug resistance of Salmonella Infantis in Peru: a study through next generation sequencing
Willi Quino1,a,c, Carmen Verónica Hurtado1,b, Oscar Escalante-Maldonado1,b,d, Diana Flores-León2,1,b,e, Orson Mestanza1,b,f, France Vences-Rosales1,b, María Luz Zamudio1,b, Ronnie G. Gavilán1,b,g
1 Instituto Nacional de Salud. Lima, Perú.
2 Universidad Alas Peruanas. Lima, Perú.
a Tecnólogo médico; b biólogo; c magister en Microbiología; d doctor en Ciencias Médicas; e magister en Bioquímica, Biología Molecular y Biomedicina; f magister en Bioinformática; g doctor en Bioquímica y Biología Molecular
RESUMEN
Objetivos. Describir los patrones fenotípicos y genotípicos de la resistencia antimicrobiana de Salmonella Infantis en Perú. Materiales y Métodos. Se analizaron 297 cepas de Salmonella sp. remitidas al Instituto Nacional de Salud (INS) en el periodo 2014-2016. Las cepas fueron caracterizadas fenotípicamente mediante pruebas microbiológicas, serológicas y de susceptibilidad antimicrobiana. En base a los patrones de resistencia antimicrobiana se seleccionaron 46 cepas que fueron caracterizadas genéticamente mediante secuenciamiento de nueva generación. Resultados. Se identificaron 193/297 (65,0%) cepas de Salmonella Infantis, de la cuales 143 (74,1%) fueron multidrogorresistentes productoras de betalactamasas de espectro extendido (BLEE). Con el secuenciamiento genómico se evidenció un nuevo perfil para Salmonella Infantis, además, se identificó la presencia de 15 diferentes determinantes genéticos de resistencia a los antimicrobianos codificados en cromosoma bacteriano y cinco codificados en un megaplásmido. Los patrones de resistencia fenotípicos y genotípicos coincidieron, a excepción de la ceftazidima. Asimismo, las 46 cepas presentaron resistencia y/o sensibilidad disminuida a las quinolonas. Conclusiones. Salmonella Infantis se ha convertido en una de las serovariedades más frecuentemente referidas al INS, la cual incluye cepas multidrogoresistentes productoras de BLEE con resistencia a las quinolonas. Finalmente, se reafirma la relevancia del secuenciamiento de nueva generación en la caracterización de nuevas variantes de patógenos de importancia para la salud pública y su uso potencial en los sistemas de vigilancia de resistencia antimicrobiana.
Palabras clave: Salmonella; Resistencia a múltiples fármacos; Secuenciación completa de genoma (fuente: DeCS BIREME).
ABSTRACT
Objectives. To describe the phenotypic and genotypic patterns of the antimicrobial resistance of Salmonella Infantis in Peru. Materials and Methods. Two hundred and ninety-seven strains of Salmonella sp. submitted to the National Institute of Health (INS, in Spanish) during 2014-2016 were analyzed. The strains were phenotypically characterized by microbiological, serological, and antimicrobial susceptibility tests. Based on antimicrobial resistance patterns, 46 strains were selected and genetically characterized by next generation sequencing. Results. 193/297 (65%) strains of Salmonella Infantis were identified, of which 143 (74.1%) were multidrug-resistant producers of extended spectrum beta-lactamases (ESBL). The genomic sequencing evidenced a new profile for Salmonella Infantis; additionally, it identified the presence of 15 different genetic determinants of antimicrobial resistance coded in bacterial chromosome and five coded in a megaplasmid. The phenotypic and genotypic resistance patterns matched, with the exception of ceftazidime. Moreover, the 46 strains presented resistance and/or decreased sensitivity to quinolones. Conclusions. Salmonella Infantis has become one of the sero-varieties most frequently referred to the INS, which includes ESBLproducing multidrug-resistant strains with resistance to quinolones. Finally, the relevance of next generation sequencing is reasserted in the characterization of new variants of pathogens that are important for public health, and their potential use in antimicrobial resistance surveillance systems.
Keywords: Salmonella; Multidrug resistance; Complete genome sequencing (source: MeSH NLM).
INTRODUCCIÓN
Salmonella enterica subespecie enterica es causante de la salmonelosis en los seres humanos y es considerada como una de las etiologías más importantes debido a su impacto en la salud pública y en la sanidad animal. Existen más de 2500 serovariedades de Salmonella enterica subespecie enterica (1), siendo las serovariedades Enteritidis y Typhimurium las de mayor importancia tanto en países desarrollados como en desarrollo (2). Sin embargo, recientemente la serovariedad Infantis (Salmonella Infantis) ha tomado protagonismo debido a que presenta frecuentemente un fenotipo resistente a los antimicrobianos (3). Las circunstancias de aparición y propagación de la resistencia a los antimicrobianos son complejas. Epidemiológicamente, se ha descrito que una de las causas más importantes es el uso generalizado de agentes antimicrobianos en animales de consumo humano (4).
Recientes estudios usando técnicas de biología molecular, han mejorado el entendimiento de la presencia de resistencia en los antimicrobianos para Salmonella, la que está asociada con genes de resistencia específicos a ciertos antimicrobianos y a genes de resistencia con objetivos múltiples (5). La técnica del secuenciamiento de nueva generación permite obtener la información genómica completa de los genes vinculados a la resistencia antimicrobiana (6), además tiene aplicabilidad en los sistema de vigilancia para predecir la susceptibilidad antimicrobiana a Salmonella (7).
En Perú se presenta un panorama similar. En el 2011, se presentó el último reporte donde la Salmonella Infantis se ubicó como la tercera serovariedad más frecuente (3). La importancia del estudio de la Salmonella Infantis no se da sólo por la frecuencia de casos sino por la presencia de resistencia a más de dos antimicrobianos (8) y la producción de betalactamasas de espectro extendido (BLEE) (3).
A nivel hospitalario, las bacterias productoras de BLEE son causantes del incremento de morbilidad y mortalidad. Las consecuencias de ignorar su presencia, puede condicionar al fracaso del tratamiento debido a un uso inapropiado de antibióticos, lo que conllevaría a aumentar la resistencia y la diseminación de este tipo de microorganismos. Asimismo, en Perú se han identificado pocas investigaciones que usen el secuenciamiento de nueva generación.
El objetivo del presente estudio es describir por primera vez los perfiles fenotípicos y genéticos de resistencia a los antibióticos de Salmonella Infantis, en muestras remitidas al Laboratorio de Referencia Nacional de Enteropatógenos del Instituto Nacional de Salud (INS) durante el periodo 2014-2016.

MATERIALES Y METODOS
DISEÑO Y POBLACIÓN DEL ESTUDIO
El presente estudio es una investigación descriptiva transversal. La población estuvo conformada por 297 cepas de Salmonella spp. de origen humano aisladas de heces, sangre y orina. Las cepas fueron remitidas de Lima, Apurímac, Cajamarca, Ancash, Lambayeque y Cuzco al Laboratorio de Referencia Nacional de Enteropatógenos del INS, durante el periodo 2014-2016 para la determinación de serovariedades y el estudio de susceptibilidad de antimicrobianos de Salmonella spp.
IDENTIFICACIÓN BIOQUÍMICA DE Salmonellla Infantis
Las 297 muestras fueron reaisladas en Caldo Tripticasa de Soya (Merck, Germany) a 37 °C durante 6 a 8 horas. Posteriormente, fueron sembradas por agotamiento en placas de Agar Salmonella Shigella (Merck, Germany) y se incubaron a 37 °C durante 18 a 24 horas. El género Salmonella fue confirmado utilizando pruebas bioquímicas convencionales. Estas cepas fueron analizadas con antisueros de aglutinación O y H (Bio Merieux, Francia) siguiendo el esquema de Kauffmann–White (9), para la identificación de la serovariedad.
IDENTIFICACIÓN MOLECULAR DE Salmonella Infantis
Las cepas identificadas por serología como Salmonella Infantis fueron seleccionadas. El ADN de dichas cepas fue extraído utilizando el kit DNesay Blood &Tissue (Qiagen, Germany) según las instrucciones del fabricante. La concentración de ADN y la pureza se evaluó mediante un espectrofotómetro (DENOVIX, EEUU). La identificación molecular se realizó mediante Reacción en cadena de Polimerasa (PCR) convencional, teniendo como blanco el gen fljB (10).
SUSCEPTIBILIDAD ANTIMICROBIANA
Se evaluó la susceptibilidad a los antimicrobianos de las cepas de Salmonella Infantis, mediante el método de difusión en disco siguiendo los lineamientos del Clinical and Laboratory Standards Institute (CLSI) (11). Los antimicrobianos utilizados fueron: ampicilina, AMP (10 μg), sulfametoxazol-trimetoprima, STX (25 μg), ácido nalidíxico, NAL (30 μg), nitrofurantoina, NIT (300 μg), (10 μg), cefotaxima, CTX (30 μg), ceftazidima, CAZ (30 μg), amoxicilina / ácido clavulánico, AMC (20/10 μg), tetraciclina, TCY (30 μg), cloranfenicol, CHL (30 μg) y ciprofloxacina, CIP (5 μg).
CONFIRMACIÓN DE LA PRODUCCIÓN DE BLEE
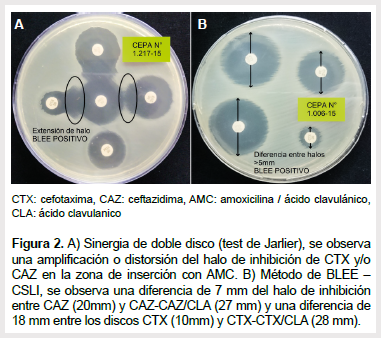
Se utilizaron dos métodos para determinar las cepas de Salmonella Infantis productoras de BLEE. Primero, la prueba de sinergia de doble disco (DDST), la cual fue realizada siguiendo el método descrito por Jarlier (12). Segundo, la prueba confirmatoria de BLEE-CLSI, la cual fue realizada siguiendo el protocolo de la CLSI (11).
SECUENCIAMIENTO Y ANÁLISIS GENÓMICO
Se seleccionaron aleatoriamente cepas de Salmonella Infantis según el perfil fenotípico de resistencia. Las muestras de ADN genómico obtenidas previamente fueron cuantificadas mediante fluorometría (Quibit 3.0 Invitrogen, Malasia). La elaboración de las librerías de secuenciamiento se realizó usando el Kit preparación de librería Nextera XT (Illumina, EEUU), y el secuenciamiento genómico utilizando el secuenciador de nueva generación MiSeq (Illumina, EEUU).
La calidad de las secuencias obtenidas de cada cepa fueron evaluadas mediante el programa Fastqc v0.11.5 (13). Los adaptadores y bases nitrogenadas de baja calidad fueron removidas usando el programa Trimommatic v0.38 (14). Se ensamblo de novo las secuencias correspondientes a cada cepa empleando el pipeline a5_pipeline.pl. Se identificó la especie y serotipo de cada cepa usando el programa Multi-locus Sequence Typing (MLST) v2.10 (16). Se utilizó un genoma de referencia de Salmonella Infantis (Genbank id CP016412 y CP016413) para identificar los contigs del cromosoma y plásmido de cada cepa secuenciada usando el programa Bandage v0.8.1(17), basado en el algoritmo BLAST (18) con parámetros de 80% de identidad, 20 pares de base (pb) de cobertura mínima. Para identificar los genes de resistencia se descargó la base de datos CARD (19) y genes del integrón 1, 2 y 3 (20), y se construyó una base de datos local.
Se realizó la predicción de secuencias codificantes para cada librería usando el programa Prodigal v2.6.3 (21). Se identificaron los genes homólogos delas secuencias a partir delos genes del genoma de referencia, con el algoritmo BLAST con <79% de identidad y una cobertura <80% de alineamiento a la referencia. El código empleado para la anotación se encuentra disponible en https://github.com/OrsonMM/Blast-score-ratio-for-genomics. Todas las secuencias obtenidas durante el estudio han sido depositadas en la base de datos del NCBI (GenBank).
CONSIDERACIONES ÉTICAS
El protocolo de estudio fue aprobado por el Comité Institucional de Ética en Investigación del INS, además, el INS otorgó la autorización para el uso de muestras biológicas criopreservadas.
RESULTADOS
IDENTIFICACIÓN DE Salmonella Infantis
En el estudio se analizaron 297 cepas de Samonella spp., de las cuales se identificaron un total de 193 cepas (65,0%) como Salmonella Infantis mediante métodos serológicos y microbiológicos. Las cepas fueron confirmadas como Salmonella Infantis mediante ensayos de PCR para la amplificación del gen fljB (Figura 1).

PERFIL DE RESISTENCIA ANTIMICROBIANA
A partir de las 193 cepas confirmadas como Salmonella Infantis se identificó 143 (74,1%) productoras de BLEE al ser resistentes a la ampicilina y cefalosporinas de tercera generación mediante métodos fenotípicos (Figura 2). Se encontraron 13 patrones de resistencia fenotípica diferentes (Tabla 1).
ANÁLISIS BIOINFORMÁTICO DE LOS GENOMAS DE Salmonella Infantis
Se seleccionaron un total de 46 cepas Salmonella Infantis de forma aleatoria. A partir del set de datos se obtuvieron en promedio: 3,2 millones de lecturas, 88 contigs, guanina-citocina 52,0%, 46X. El alineamiento con el genoma de referencia permitió identificar y separar los contigs pertenecientes al cromosoma (48Mpb) y los contigs pertenecientes al plásmido (249 – 354 Kpb) para cada una de nuestras muestras.
El análisis de MLST identificó que 42 de las cepas secuenciadas corresponden al sequence type 32 (ST32). Las cuatro cepas restantes no coincidieron con el sequence type (ST) reportados para Salmonella Infantis, al presentar incongruencias para los genes aroC, hisD y purE (Tabla 2).
Mediante la anotación sobre los contigs del plásmido se logró identificar el integrón clase 1 conformada por los genes Int1, aadA1, QaceΔ1 y sul1 y, el integrón clase 2 conformada por los genes Int2 y dfrA1. Además, en los contigs cromosómicos la anotación detectó genes asociados a resistencia contra diversos antimicrobianos. En la Figura 4 se observa la distribución de los genes asociados a la resistencia de los 10 antimicrobianos utilizados en este estudio y de otros cinco grupos de antimicrobianos.
Se compararon los resultados obtenidos por los patrones de resistencia fenotípicos con la caracterización molecular obtenidos por el secuenciamiento. Los resultados, en general, muestran una correlación entre la presencia/ausencia de los genes y la detección fenotípica de la resistencia. Esta correlación se aprecia gráficamente en la Figura 3. Se evidencia claramente en el caso de CHL donde las 38 cepas que poseen el gen floR son resistentes y las ocho cepas que no poseen el gen floR son sensibles. En el caso de CIP, las 46 cepas poseen el gen gyrA con la mutación D87Y y todas mostraron una sensibilidad disminuida. Sin embargo, se encontró un caso discordante con CAZ, a pesar de que las 46 cepas poseen los genes asociados a su resistencia, sólo seis cepas fueron resistentes, 12 intermedias y 28 sensibles.
DISCUSIÓN
La Salmonella Infantis es un patógeno emergente con multidrogoresistencia reportado en diferentes regiones del mundo (22). En el ámbito local y regional existen pocos reportes de esta serovariedad. Por lo cual, el presente trabajo ofrece la primera descripción de los perfiles fenotípicos y genéticos de resistencia a los antimicrobianos de cepas Salmonella Infantis remitidas al INS.
En este estudio el 65,0% de las cepas remitidas al INS fueron confirmadas como Salmonella Infantis, esta alta ocurrencia podría estar relacionada con el incremento de casos asociados a esta serovariedad en el país durante los últimos años, tal como se reportó en el 2011 a partir de un grupo reducido de cepas evaluadas (5). Asimismo, se describe por primera vez que el 74,1% de las Salmonellas Infantis corresponden a un perfil multidrogorresistente, agrupándose en 13 patrones diferentes, con resistencia a más de cinco antimicrobianos y la presencia fenotípica de BLEE. El perfil BLEE fue reportado en una cepa de Salmonella Infantis del 2013 aislada de un paciente procedente de Lima (23). La presencia de cepas productoras de BLEE dentro de esta serovariedad podría explicar el incremento de casos de Salmonella Infantis en las muestras remitidas al INS.
En estudios previos usando genes de MLST se asigna esta serovariedad dentro del genotipo ST32 (24). Sin embargo, en el presente estudio se identificaron 42 cepas con este patrón; las otras cepas confirmadas serológicamente y molecularmente como Salmonella Infantis presentaron ST no característicos en la base de datos (25), estas deben ser considerados para la actualización de la base de datos MLST.
Además, el secuenciamiento genómico es también importante para la caracterización de cepas emergentes con relevancia clínica. Utilizando esta metodología se encontró que las 46 cepas estudiadas, presentaban un megaplásmido asociado a la multidrogoresistencia a CHL, SXT, TCY, CAZ y CTX (26). La literatura describe que el megaplásmido puede contener los integrones clase 1, 2 y 3 (20,27), sin embargo, las cepas incluidas en el estudio presentan el integrón de clase 1 y 2. Estos están formados por genes dfrA1 y sulf1 que dan resistencia a SXT (27), el gen tetC que da resistencia a TCY (28), el gen CTX-M-65 que da resistencia a CAZ y CTX (29). Por otro lado, el secuenciamiento genómico permitió detectar genes cromosómicos (Figura 4) vinculados con la resistencia de otros antimicrobianos como NAL, CIP, NIT, AMP (30).
Podemos observar que los perfiles fenotípicos de multidrogoresistencia fueron confirmados con la presencia de los genes encontrados en el megaplásmido y el cromosoma. Tres casos resumen claramente lo expuesto. Primero, el gen floR, asociado a la resistencia a CHL está presente en 38 y ausente en 8 cepas y las mismas cepas mostraron resistencia y sensibilidad fenotípica respectivamente. Segundo, el gen gyrA, asociado a la resistencia a las quinolonas (NAL y CIP) tiene la mutación D87Y para las 46 cepas y todas ellas mostraron resistencia a NAL y sensibilidad disminuida a CIP. Tercero, los genes asociados a la resistencia (plasmídicos y cromosómicos) a CAZ están presentes en las 46 cepas, pero sólo seis mostraron resistencia fenotípica. Esto sugiere que habría otros mecanismos de resistencia asociados a CAZ que aún no están descritos en la literatura.
Los resultados obtenidos podrían esclarecer la ineficacia de los antimicrobianos en el tratamiento para la salmonelosis y contribuir a la generación de evidencia para establecer un nuevo esquema, aplicable a la salud pública en el tratamiento de dicha enfermedad considerando los perfiles de resistencia descritos.
El estudio presenta la limitación de no haber analizado muestras de las 24 regiones de Perú, lo que hubiera mejorado su representatividad. A pesar de ello, las cepas recibidas han permitido detectar la circulación de los genes responsables de la multidrogoresistencia de Salmonella Infantis productoras de BLEE. Además, los recursos económicos con los que se disponía permitieron secuenciar un número limitado de cepas. Pese a estas limitaciones el estudio brinda información sobre un mayor número de cepas que los disponibles actualmente en las bases de datos.
En conclusión, se logró describir los patrones de resistencia en las muestras remitidas al Instituto Nacional de Salud; además, el uso del secuenciamiento permitió identificar genes de resistencia asociados a otros grupos de antimicrobianos que no estaban incluidos en las recomendaciones de la CLSI (11). El secuenciamiento es la herramienta de elección ya que brinda información necesaria para determinar el patrón de resistencia completo, lo que podría servir para trazar una apropiada estrategia de salud pública en Perú.
Agradecimientos: Al Fondo Nacional de Desarrollo Científico, Tecnológico y de Innovación Tecnológica (FONDECYT) y al Instituto Nacional de Salud por el apoyo en el financiamiento del proyecto.
Contribución de los autores: QW, HV, ZML y GGR participaron en la concepción, delineación de hipótesis y diseño del estudio. WQ, EMO, HV, FLD, MO, VRF participaron en el análisis, interpretación de datos y redacción del artículo. RGG, EMO, ZML participaron en la revisión crítica del artículo.
Fuentes de financiamiento: La investigación fue financiada por el Fondo Nacional de Desarrollo Científico, Tecnológico y de Innovación Tecnológica (FONDECYT) y por el Instituto Nacional de Salud en el marco del proyecto Epidemiología molecular y genética de la salmonelosis multirresistente, aprobado con RD. N° 191-2016-OGITT-OPE/INS.
Conflicto de interés: Todos los autores declaran que no tienen ningún conflicto de interés en relación con esta publicación.
REFERENCIAS BIBLIOGRÁFICAS
1. Andino A, Hanning I. Salmonella enterica: Survival, Colonization, and Virulence Differences among Serovars. Scientific World Journal. 2015;2015:520179. doi: 10.1155/2015/520179.
2. Rodrigue DC, Tauxe RV, Rowe B. International increase in Salmonella enteritidis: a new pandemic?. Epidemiol Infect. 1990;105(1):21-7.
3. Zamudio ML, Meza A, Bailón H, Martinez-Urtaza J, Campos J. Experiencias en la vigilancia epidemiológica de agentes patógenos transmitidos por alimentos a través de electroforesis en campo pulsado (PFGE) en el Perú. Rev Peru Med Exp Salud Publica. 2011;28:128-35.
4. Su L-H, Chiu C-H, Chu C, Ou JT. Antimicrobial resistance in nontyphoid Salmonella serotypes: a global challenge. Clin Infect Dis. 2004;39(4):546-51.
5. Poppe C, Martin LC, Gyles CL, Reid-Smith R, Boerlin P, McEwen SA, et al. Acquisition of resistance to extended-spectrum cephalosporins by Salmonella enterica subsp. enterica serovar Newport and Escherichia coli in the turkey poult intestinal tract. Appl Environ Microbiol. 2005;71(3):1184-92.
6. McDermott PF, Tyson GH, Kabera C, Chen Y, Li C, Folster JP, et al. Whole-Genome Sequencing for Detecting Antimicrobial Resistance in Nontyphoidal Salmonella. Antimicrob Agents Chemother. 2016;60(9):5515-20. doi: 10.1128/AAC.01030-16.
7. Ellington MJ, Ekelund O, Aarestrup FM, Canton R, Doumith M, Giske C, et al. The role of whole genome sequencing in antimicrobial susceptibility testing of bacteria: report from the EUCAST Subcommittee. Clin Microbiol Infect. 2017;23(1):2-22. doi: 10.1016/j.cmi.2016.11.012.
8. Silva C, Betancor L, García C, Astocondor L, Hinostroza N, Bisio J, et al. Characterization of Salmonella enterica isolates causing bacteremia in Lima, Peru, using multiple typing methods. PLoS One. 2017;12(12):e0189946. doi: 10.1371/journal.pone.0189946.
9. Hudzicki J. Kirby-Bauer Disk Diffusion Susceptibility Test Protocol [Internet]. Washington, DC: American Society for Microbiology; 2016 [citado 12 de septiembre de 2018]. Disponible en: http://www.asmscience.org/content/education/protocol/protocol.3189.
10. Kardos G, Farkas T, Antal M, Nógrády N, Kiss I. Novel PCR assay for identification of Salmonella enterica serovar Infantis. Lett Appl Microbiol. 2007;45(4):421-5.
11. Performance Standards for Antimicrobial Susceptibility Testing; Twenty-Fourth Informational Supplement (M100-S24) [Internet]. Washington, DC: AACC; 2014 [citado 7 de septiembre de 2018]. Disponible en: https://www.aacc.org/store/books/9200/performance-standards-for-antimicrobial-sus-ceptibility-testing.
12. Jarlier V, Nicolas MH, Fournier G, Philippon A. Extended broad-spectrum beta-lactamases conferring transferable resistance to newer beta-lactam agents in Enterobacteriaceae: hospital prevalence and susceptibility patterns. Rev Infect Dis. 1988;10(4):867-78.
13. Andrews S. FastQC: a quality control tool for high throughput sequence data [Internet]. Cambridge: Babraham Bioinformatics; 2010. Disponible en: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/.
14. Bolger AM, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 2014;30(15):2114-20. doi: 10.1093/ bioinformatics/btu170.
15. Coil D, Jospin G, Darling AE. A5-miseq: an updated pipeline to assemble microbial genomes from Illumina MiSeq data. Bioinformatics. 2015;31(4):587-9. doi: 10.1093/bioinformatics/btu661.
16. Larsen MV, Cosentino S, Rasmussen S, Friis C, Hasman H, Marvig RL, et al. Multilocus sequence typing of total-genome-sequenced bacteria. J Clin Microbiol. 2012;50(4):1355-61. doi: 10.1128/JCM.06094-11.
17. Wick RR, Schultz MB, Zobel J, Holt KE. Bandage: interactive visualization of de novo genome assemblies. Bioinformatics. 2015;31(20):3350-2. doi: 10.1093/bioinformatics/btv383
18. Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. Basic local alignment search tool. J Mol Biol. 1990;215(3):403-10. doi: 10.1016/S0022-2836(05)80360-2.
19. Jia B, Raphenya AR, Alcock B, Waglechner N, Guo P, Tsang KK, et al. CARD 2017: expansion and model-centric curation of the comprehensive antibiotic resistance database. Nucleic Acids Res. 2017;45(D1):D566-D573. doi: 10.1093/nar/gkw1004.
20. Asgharpour F, Rajabnia R, Ferdosi Shahandashti E, Marashi MA, Khalilian M, Moulana Z. Investigation of Class I Integron in Salmonella infantis and Its Association With Drug Resistance. Jundishapur J Microbiol. 2014;7(5):e10019. doi: 10.5812/jjm.10019.
21. Hyatt D, Chen G-L, Locascio PF, Land ML, Larimer FW, Hauser LJ. Prodigal: prokaryotic gene recognition and translation initiation site identification. BMC Bioinformatics. 2010;11:119. doi: 10.1186/1471-2105-11-119.
22. Franco A, Leekitcharoenphon P, Feltrin F, Alba P, Cordaro G, Iurescia M, et al. Emergence of a Clonal Lineage of Multidrug-Resistant ESBL-Producing Salmonella Infantis Transmitted from Broilers and Broiler Meat to Humans in Italy between 2011 and 2014. PLoS One. 2015;10(12):e0144802. doi: 10.1371/journal.pone.0144802.
23. Silva C, Betancor L, García C, Astocondor L, Hinostroza N, Bisio J, et al. Characterization of Salmonella enterica isolates causing bacteremia in Lima, Peru, using multiple typing methods. PLoS One. 2017;12(12):e0189946. doi: 10.1371/journal.pone.0189946.
24. Pethplerdprao P, Supa-amornkul S, Panvisavas N, Chaturongakul S. Salmonella enterica multilocus sequence typing and its correlation with serotypes. Food Biotechnology. 2017;31(2):73-9. doi: 10.1080/08905436.2017.1301820.
25. Urwin R, Maiden MCJ. Multi-locus sequence typing: a tool for global epidemiology. Trends Microbiol. 2003;11(10):479-87.
26. Aviv G, Tsyba K, Steck N, Salmon-Divon M, Cornelius A, Rahav G, et al. A unique megaplasmid contributes to stress tolerance and pathogenicity of an emergent Salmonella enterica serovar Infantis strain. Environ Microbiol. 2014;16(4):977-94. doi: 10.1111/1462-2920.12351.
27. Asgharpour F, Mahmoud S, Marashi A, Moulana Z. Molecular detection of class 1, 2 and 3 integrons and some antimicrobial resistance genes in Salmonella Infantis isolates. Iran J Microbiol. 2018;10(2):104-110.
28. Lynne AM, Rhodes-Clark BS, Bliven K, Zhao S, Foley SL. Antimicrobial Resistance Genes Associated with Salmonella enterica Serovar Newport Isolates from Food Animals. Antimicrob Agents Chemother. 2008 Jan;52(1):353-6. doi: 10.1128/AAC.00842-07.
29. Tate H, Folster JP, Hsu C-H, Chen J, Hoffmann M, Li C, et al. Comparative Analysis of Extended-Spectrum-β-Lactamase CTX-M-65-Producing Salmonella enterica Serovar Infantis Isolates from Humans, Food Animals, and Retail Chickens in the United States. Antimicrob Agents Chemother. 2017;61(7). pii: e00488-17. doi: 10.1128/AAC.00488-17.
30. Li L, Dai X, Wang Y, Yang Y, Zhao X, Wang L, et al. RNA-seq-based analysis of drug-resistant Salmonella enterica serovar Typhimurium selected in vivo and in vitro. PLoS One. 2017;12(4):e0175234. doi: 10.1371/journal.pone.0175234.
Correspondencia: Willi Quino Sifuentes
Dirección: Cápac Yupanqui 1400, Jesús María, Lima, Perú
Teléfono: 511-7481111 anexo 2117
Correo electrónico: wquino@ins.gob.pe
Recibido: 14/09/2018
Aprobado: 30/01/2019
En línea: 08/03/2019