
ARTÍCULO ORIGINAL
Caracterización molecular de la nucleoproteína del virus de la rabia en canes procedentes de Arequipa, Perú
Molecular characterization of the rabies virus nucleoprotein in dogs from Arequipa, Peru
Carina Rosario Mantari Torpoco1,a, Alfredo Miguel Berrocal Huallpa2,b, Abraham Omar Espinoza-Culupú3,4,c, Ricardo Luis López-Ingunza1,d
1 Laboratorio de Zoonosis Virales, Instituto Nacional de Salud. Lima, Perú.
2 Laboratorio de Sistemática y Diversidad Vegetal, Museo de Historia Natural, Universidad Nacional Mayor de San Marcos. Lima, Perú.
3 Laboratorio de Bacteriología, Instituto Butantan. São Paulo, Brasil.
4 Programa de Posgrado Interunidades en Biotecnología. Universidade de São Paulo, Brasil.
a Médico veterinario; b biólogo, magíster en Mejoramiento genético de plantas; c biólogo, magíster en Biología molecular, d médico veterinario, magíster en Medicina veterinaria preventiva.
El artículo es parte de la tesis de Carina Mantari Torpoco para obtener el grado de magíster en Biología Molecular de la Universidad Nacional Mayor de San Marcos, Lima, Perú.
RESUMEN
Objetivos. Caracterizar la nucleoproteína (N) y establecer el origen del virus de la rabia en canes procedentes de Arequipa. Materiales y métodos. Se analizaron 30 muestras de tejido nervioso procedentes de los departamentos de Arequipa y Puno. Se extrajo el ARN total de las muestras y se sintetizó ADNc para amplificar el gen de la nucleoproteína, secuenciarlo y realizar el análisis bioinformático. Resultados. Se obtuvo la formación de un grupo definido con respecto al grupo externo (European bat lyssavirus). Este grupo fue clasificado en dos subgrupos, uno constituido por muestras procedentes de Puno y Arequipa (subgrupo A), y otro por muestras de Puno (subgrupo B), observándose una identidad nucleotídica de 99,9% en el subgrupo A. Conclusiones. Los agrupamientos de las secuencias virales muestran que los casos de rabia canina notificados en Arequipa son el resultado de la expansión de rabia canina procedente de la región endémica de Puno.
Palabras clave: Virus ARN; Virus de la Rabia; Secuenciación de Nucleótidos de Alto Rendimiento; Perros; Perú (fuente: DeCS BIREME).
ABSTRACT
Objective. To characterize the nucleoprotein (N) and establish the origin of the rabies virus in dogs coming from Arequipa. Materials and Methods. Thirty samples of nervous tissue from the departments of Arequipa and Puno were analyzed. Total RNA was extracted from the samples and cDNA was synthesized to amplify the nucleoprotein gene, sequence it, and perform bioinformatics analysis. Results. A defined group was formed with respect to the external group (European bat lyssavirus). This group was classified into two subgroups, one constituted by samples coming from Puno and Arequipa (subgroup A), and another one by samples from Puno (subgroup B), exhibiting a nucleotide identity of 99.9% in subgroup A. This group was classified in two subgroups, one constituted by samples coming from Puno and Arequipa (subgroup A), and another one by samples from Puno (subgroup B), observing a nucleotide identity of 99.9% in subgroup A. Conclusions. The groupings of viral sequences show that the cases of canine rabies reported in Arequipa are the result of the expansion of canine rabies from the endemic region of Puno.
Keywords: RNA Virus; rabies virus; high-performance nucleotide sequencing; dogs; Peru (source: MeSH NLM).
INTRODUCCIÓN
La rabia es una enfermedad neurológica mortal que afecta una amplia variedad de hospederos mamíferos incluyendo al hombre (1), y su transmisión está asociada a especies hospederos, comúnmente carnívoros y quirópteros (2,3). Aunque la vacunación y la terapia con anticuerpos es efectiva en el tratamiento posexposición, se estima al menos 59 000 muertes humanas anuales causada por rabia canina, siendo las áreas más afectadas África y Asia (4,5).
El virus de la rabia (VR), especie del género Lyssavirus, familia Rabdoviridae, orden Mononegavirales; es un virus ARN de cadena simple, sentido negativo, con una estructura en forma de bala, de unos 12Kb de longitud que codifica cinco proteínas estructurales: nucleoproteína (N), fosfoproteína (P), proteína de la matriz (M), glicoproteína (G) y ARN polimerasa dependiente de ARN (L) (6).
Actualmente el análisis molecular es utilizado como una herramienta en la investigación epidemiológica de la rabia (7,8), siendo la nucleoproteína uno de los genes más empleados al permitir la determinación de especies, origen de variantes del virus, dinámica de distribución geográfica espacial e identificación del ancestro común (9). De esta manera, estudios previos, en epidemiología molecular de la rabia en el Perú, utilizaron el gen N para la caracterización molecular y distribución de linajes en reservorios silvestres, así como para la identificación de posibles nuevos reservorios de la enfermedad (10).
En Perú, los principales reservorios del VR son los canes (rabia urbana) y murciélagos hematófagos, Desmodus rotundus (rabia silvestre), los cuales mantienen ciclos independientes de transmisión (10). Según los reportes del Laboratorio de Zoonosis Virales del Instituto Nacional de Salud (INS), 80 casos de rabia urbana fueron notificados en los departamentos de Madre de Dios, Piura, y Puno, durante el periodo 2010-2014; cinco casos en humanos, nueve en diversas especies (bovino, llama, y gato) y 66 casos en canes, este último con un promedio de 14 casos al año (11).
Sin embargo, tras una ausencia de 15 años de rabia canina en eldepartamento de Arequipa, dos casos fueron notificados en diciembre del 2014 en la provincia de Camaná (Arequipa), cifra que aumentó a 19 casos durante el 2015, dando inicio a la expansión de rabia canina en el sur del país (12,13). En Perú, si bien la rabia canina está focalizada en Puno, departamento fronterizo con Bolivia, el cual es endémico a rabia canina (14); la cercanía geográfica de Arequipa, sugirió considerar a Bolivia y Puno como posibles orígenes del brote. Adicionalmente la ausencia de control sanitario en el ingreso de canes en estas áreas fronterizas aumenta dicha posibilidad.
El objetivo de este estudio fue caracterizar la nucleoproteína (N) y establecer el origen del virus de la rabia en canes procedentes de Arequipa durante el periodo 2014-2017.

MATERIALES Y MÉTODOS
MUESTRAS
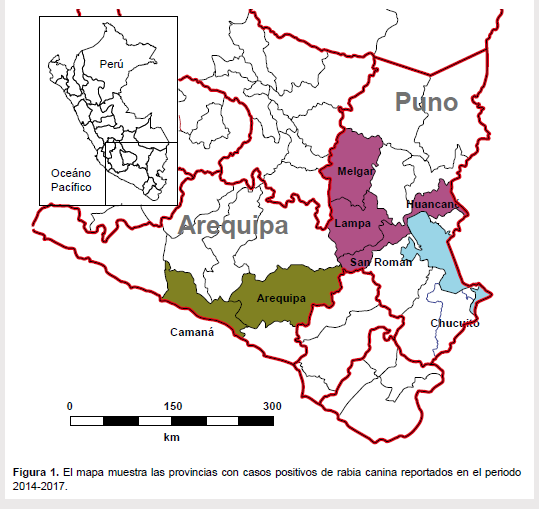
Un total de 30 muestras de tejido nervioso de canes procedentes de los departamentos de Arequipa (n=16) y Puno (n=14) fueron remitidas al laboratorio de Zoonosis Virales del Instituto Nacional de Salud (INS) durante el periodo 2014-2017, como parte del programa de vigilancia del Ministerio de Salud (Figura 1). Todas estas muestras fueron evaluadas por la prueba de inmunofluorescencia directa (IFD) para su diagnóstico como positivas al VR (15). La cepa estándar "Challenge Virus Standard, CVS" fue utilizada como control positivo en las pruebas de IFD y posteriormente en la RT-PCR.
EXTRACCIÓN DE ARN Y TRANSCRIPCIÓN REVERSA (RT)
Para la extracción del ARN total se empleó la técnica trizol-cloroformo, utilizando TRI-reagent (sigma) y siguiendo las instrucciones del fabricante. Se usó entre 30 y 50 mg de tejido nervioso de cada muestra. Posteriormente, se cuantificó el ARN utilizando la relación de absorbancia 260/280 en el nanodrop y se determinó la integridad en geles de agarosa al 1% en buffer TBE (tris base 450 mM, ácido bórico 450 mM, EDTA 10 mM) y teñido con bromuro de etidio (BrEt).
El ADN complementario (ADNc) fue sintetizado a partir de 300 ng de ARN totalutilizando el kit Cloned Avian Myeloblastosis Virus (AMV) Reverse Transcriptase (Invitrogen), bajo las siguientes condiciones de ciclado: 65 °C por cinco minutos, seguidos de 42 °C por 60 minutos y 85 °C por cinco minutos.
RT- PCR
Un total de 100 ng de ADNc fue usado como molde para amplificar el gen de la nucleoproteína, en un volumen final reacción de 25 µl, con buffer de PCR 1X (Invitrogen), MgCl21 mM, dNTPs 0,2 mM, 0,2 µM de cada primer, y dos unidades de la enzima Platinum® Taq DNA Polymerase (Invitrogen). Con la finalidad de obtener el gen completo de la nucleoproteína, se utilizaron dos pares de oligonucleótidos previamente reportados (Lys 001 5’ACGCTTAACGAMAAA3’ y 921B 5´YGTGTTCAAYCTHATYCACTT3’, y 550F 5’ATGTGYGCTAAYTGGAGYAC3’ y 304 5’TTGACGAAGATCTTGCTCAT3) (10,16,17). El programa de amplificación para la PCR fue el siguiente: 94 °C por dos minutos, con 40 ciclos de 94 °C por 30 segundos 37 °C por 30 segundos, y 72 °C por 90 segundos, seguidos de una extensión final de 72 °C por siete minutos (17).
Los productos finales de la PCR fueron corridos mediante electroforesis en geles de agarosa al 1% en buffer TBE 1X durante una hora a 100 V, teñidos con bromuro de etidio (BrEt) y visualizados en el transiluminador.
SECUENCIAMIENTO Y ANÁLISIS FILOGENÉTICO
Los productos amplificados de la PCR fueron purificados con QIAquick PCR Purification Kit (QIAGEN, Alemania) y secuenciados utilizando dos pares de oligonucleótidos específicos (Lys001-921B y 550F-304). El secuenciamiento fue bidireccional empleando BigDye Terminator v3.1 Cycle Sequencing Kit (Applied Biosystems), con modificaciones en las condiciones de ciclaje: 96 °C por un minuto; 35 ciclos de 96 °C por diez segundos, 50 °C por 45 segundos, y 60 °C por un minuto. El secuenciamiento se realizó en el analizador genético Applied Biosystems 3500 DNA analyzer.
Los cromatogramas obtenidos del secuenciamiento se visualizaron y editaron con el programa MEGA 6.0 (18), luego ensamblados con el software libre CAP 3 (19), y posteriormente alineados con ClustalW (20) para la identificación de cambios nucleotídicos y aminoacídicos. Para la identificación de la región codificante ubicada en la posición (71-1423) se usó la cepa de referencia PV (GenBank acceso N° M13215.1).
Por otro lado se recuperaron secuencias parciales de la nucleoproteína del VR (posición 1157-1476), almacenadas en el GenBank y procedentes de Bolivia (accesión AY340753.1, AY340756.1 AY340757.1 AY340755.1 AY340721.1 AY340724.1 AY340722.1 AY340723.1 AY340727.1 AY340728.1 AY340703.1 AY340746.1 AY340747.1 AY340785.1 AY340766.1 AY340767.1 AY340704.1) (21) que se utilizaron para identificar las posibles relaciones filogenéticas entre las muestras procedentes de Perú y Bolivia. La especie European bat 2 lyssavirus (GenBank acceso N° KY688156.1) se usó como grupo externo para poder enraizar el árbol.
El árbol filogenético fue construido empleando el método de máxima verosimilitud con un valor bootstrap de 1000 réplicas, y las sustituciones nucleotídicas y aminoacídicas fueron calculadas con los métodos de Kimura 2-parameter y Jones-Taylor-Thornton respectivamente, para este análisis se utilizó el programa Mega 6.0 (18).
CONSIDERACIONES ÉTICAS
Todas las muestras fueron colectadas como parte del programa de vigilancia del Ministerio de Salud, obtenidas de canes infectados naturalmente. No se utilizaron animales con fines experimentales ni se obtuvieron muestras de procedimientos experimentales.
RESULTADOS
Un total de 30 muestras del VR proveniente de diferentes distritos de los departamentos de Puno y Arequipa, fueron incluidos en el presente estudio (Tabla 1).
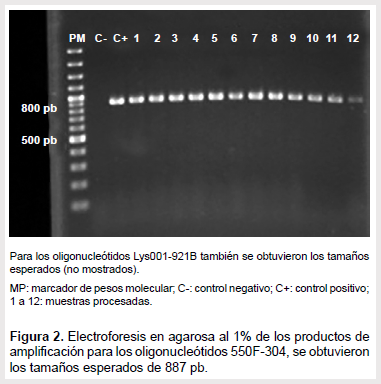
Mediante RT-PCR y usando los oligonucleótidos Lys001-921B y 550F-304 se logró amplificar dos regiones parciales de la nucleoproteína de tamaños 1011 pb y 887 pb respectivamente, como se muestra en la Figura 2, y estas fueron secuenciadas utilizando el método Sanger.
Los resultados de secuenciamiento corresponden a dos regiones amplificadas con los oligonucleótidos Lys001-921B y 550F-304, las cuales fueron ensamblados y editados para obtener la secuencia nucleotídica de la región codificante de la nucleoproteína, con un tamaño de 1352 nucleótidos (posición 71-1423).
La comparación de las secuencias nucleotídicas obtenidas en este estudio con respecto a la cepa PV permitió identificar sustitución de aminoácidos en las posiciones C40G, C59G, S61N, V95L, G106D, P135S, S157N, V322I, I410M (Anexo 1- visualizar en versión electrónica).
El análisis filogenético de la nucleoproteína del VR de las 30 muestras analizadas reveló la formación de un grupo respecto a European bat 2 lyssavirus (grupo externo) (GenBank acceso N° KY688156.1). Este grupo se dividió en un subgrupo A formado por las muestras de la provincia de Melgar, Lampa, San Román, Huancané (Puno) y las provincias de Camaná y Arequipa (Arequipa), y el subgrupo B formado por las muestras procedentes de la provincia de Chucuito, limítrofes con Bolivia (Figura 3). La distancia genética del VR entre los subgrupos A y B fue de 1,25%, y las distancias intragrupo de A y B fueron 0,12% y 0,0% respectivamente.
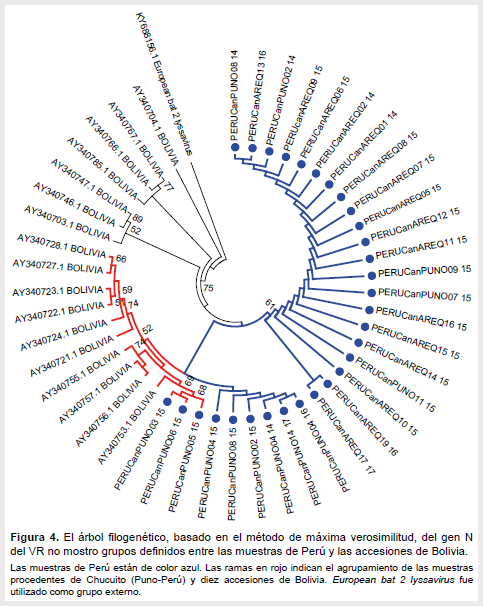
En relación al análisis filogenético entre las 30 muestras de Perú y las 17 accesiones de Bolivia, no se formaron grupos definidos, sin embargo se puede observar un agrupamiento interno entre las muestras de Chucuito (subgrupo B) con diez accesiones de Bolivia (accesión AY340753.1, AY340756.1 AY340757.1 AY340755.1 AY340721.1 AY340724.1 AY340722.1 AY340723.1 AY340727.1 AY340728.1)(Figura 4).
DISCUSIÓN
El presente estudio permitió determinar el origen del brote de rabia canina en el departamento de Arequipa mediante la caracterización molecular de la nucleoproteína del VR en canes procedentes del departamento de Arequipa y Puno.

En función al análisis de agrupamiento basado en la similitud y cambios a nivel de nucleótidos, y en relación al grupo externo European bat 2 lyssavirus se observó la formación de un grupo que incluye los aislados del VR circulando en canes, el cual fue dividido en dos subgrupos. El subgrupo «A» formado por muestras procedentes de las provincias Puno y Arequipa, ubicadas al noreste de Chucuito, que comparten una identidad nucleotídica del 99,9%, característica que permitió relacionarla con el posible origen del brote en Arequipa; y el subgrupo B que agrupa muestras procedentes de la provincia de Chucuito limítrofe con Bolivia, el cual presentó una identidad del 100%.
Si bien la similitud entre estos subgrupos fue de 98,75%, la sustitución en el sitio C40G nos permitió diferenciarlos; este agrupamiento estaría relacionado con la circulación localizada (geográfica) del virus, característica del VR para mantener una relación específica con su hospedero y/o ubicación geográfica. Hallazgos similares relacionados con la filogeografía del VR también fueron observados en estudios de caracterización de la nucleoproteína del VR en cánidos del norte y noreste de Brasil, que establecen la formación de grupos como resultado de la adaptación de subpoblaciones de virus a regiones geográficas (22,23); así también estudios filogenéticos del VR en canes de la India y reportes de carnívoros estudiados en Brasil mencionan que el agrupamiento genético está determinado geográficamente (24,25).
Con respecto al análisis filogenético entre las muestras de Perú y las accesiones de Bolivia no se formaron grupos definidos, sin embargo, diez accesiones de Bolivia se agruparon con las muestras de la provincia de Chucuito-Puno (subgrupo B), lo que indicaría una alta similitud del VR circulante en las regiones limítrofes entre ambos países (Figura 4). Hallazgo a considerar pues si bien los casos de rabia humana y canina, en América Latina y el Caribe, han disminuido (14); la rabia canina aún sigue siendo endémica en Bolivia, país que reportó un promedio de 330 casos entre el 2015 y 2016, cifra que se incrementó a 954 casos en el 2017 (13). Dicha situación aumenta el riesgo de transmisión de rabia canina en el sur este del Perú, debido a que el departamento de Puno, límite fronterizo con Bolivia, mantiene un tránsito y comercio activo entre ambos países. Sumado a esto, la falta de regulación en el control de ingreso y salida de mascotas facilita el movimiento de canes con rabia, considerada aún como una fuerte amenaza para las áreas libres de rabia canina (26,27).
En el análisis filogenético se observó también el agrupamiento de los aislados del VR obtenidos en el periodo 2016-2017 con los aislados recuperados al inicio del brote, esta alta similitud nucleotídica posiblemente se deba a: la adaptación del virus para mantenerse en la población hospedera como ha sido reportada en estudios anteriores (24,28,29); al comportamiento del movimiento de la población de canes en Arequipa (30); o debido a la limitada variedad de especies reservorio en el área de la epizootia (31).
En estos últimos años el departamento de Arequipa viene reportando un alto número de casos positivos de rabia canina, 58 casos en el 2016 y 48 casos en el 2017 (11), persistencia probablemente relacionada a diversos factores: económicos, políticos, sociales y culturales (30,32-34). En este contexto el uso de herramientas moleculares es necesaria como soporte de la investigación epidemiológica, ya que la identificación y/o caracterización del VR circulante nos permitirá establecer vínculos filogeográficos (33), conocer la dinámica del virus en el área epizoótica, y contribuir al diseño de estrategias para el control y prevención de rabia canina. Así mismo, el análisis molecular de la región de la nucleoproteína servirá como referencia para el desarrollo de métodos diagnósticos basados en el diseño de sondas que permitan la identificación de la variante de rabia canina circulante en el país.
Una de las limitaciones del estudio fue la selección de muestras, ya que estuvo limitada a la vigilancia pasiva que realiza el Ministerio de Salud, así también, la poca información detallada de los datos epidemiológicos correspondientes a las accesiones de Bolivia no permitió establecer nexos epidemiológicos con mayor robustez.
En conclusión, el análisis molecular determinó una alta similitud nucleotídica de las muestras obtenidas del brote de rabia canina en Arequipa con las muestras procedentes de los distritos de Melgar, Lampa o San Román, áreas que mantienen una transmisión sostenida de rabia canina hace 20 años, lo que permitió determinar el posible origen de los casos de rabia canina en Arequipa.
Contribuciones de los autores: CM ha participado en la concepción y diseño de la investigación además de la generación, análisis de datos y la redacción del artículo. AB realizó el análisis e interpretación de datos, y apoyo en la redacción. AEC realizó el análisis, interpretación y apoyo en la redacción. RL realizó la concepción y diseño de la investigación, la revisión crítica del artículo, y la aprobación final del mismo.
Fuentes de financiamiento: autofinanciado.
Conflictos de interés: ninguno.
Agradecimientos: Al MSc. Blgo. Víctor Jiménez Vázquez por su apoyo en la edición de imágenes para el artículo.
REFERENCIAS BIBLIOGRÁFICAS
1. World Health Organization. Rabies 2018 [Internet]. WHO; 2018 [citado el 27 de octubre de 2018]. Disponible en: http://www.who.int/ rabies/en/.
2. Ding NZ, Xu DS, Sun YY, He H Bin, He CQ. A permanent host shift of rabies virus from Chiroptera to Carnivora associated with recombination. Sci Rep. 2017;7(1):1–9. doi: http://dx.doi.org/10.1038/s41598-017-00395-2s.
3. Badrane and Noël Tordo. Host Switching in Lyssavirus History from the Chiroptera to the Carnivora Orders. 2001;75(17):8096-104.
4. Hampson K, Coudeville L, Lembo T, Sambo M, Kieffer A, Attlan M, et al. Estimating the Global Burden of Endemic Canine Rabies. Plos Negl Trop Dis. 2015;9(4):e003709. doi: 10.1371/journal.pntd.0003709.
5. Kessels JA, Recuenco S, Navarro-Vela AM, Deray R, Vigilato M, Ertl H, et al. Pre-exposure rabies prophylaxis: a systematic review. Bull World Health Organ. 2017;95(3):210-219.
6. Wunner WH, Conzelmann KL. Rabies Virus. En Alan Jackson, editor. Rabies, Scientific Basis of the Disease and Its Management. 3ra ed. USA. Academic Press; 2013. p 17-24.
7. Smith JS, Orciari LA, Yager PA, David Seidel H, Warner CK. Epidemiologic and Historical Relationships among 87 Rabies Virus Isolates as Determined by Limited Sequence Analysis. J Infect Dis. 1992;166(2):296-307.
8. Kissi B, Tordo N, Bourhy H. Genetic polymorphism in the rabies virus nucleoprotein. Virology. 1995;209:526-537.
9. Johnson N, McElhinney LM, Smith J, Lowings P, Fooks AR. Phylogenetic comparison of the genus Lyssavirus using distal coding sequences of the glycoprotein and nucleoprotein genes. Arch Virol. 2002;147(11):2111-23.
10. Condori-Condori RE, Streicker DG, Cabezas-Sanchez C, Velasco-Villa A. Enzootic and epizootic rabies associated with vampire bats, Peru. Emerg Infect Dis. 2013;19(9):1463-9.
11. Laboratorio de Referencia Nacional de Zoonosis Virales, Centro Nacional de Salud Pública, Instituto Nacional de Salud I. Situación de la rabia en el Perú, INS, 2015-2017. Bol Inst Nac Salud. 2018;24(3-4):45-51.
12. Vargas E. Situación de la rabia en el Perú. Año 2015 a la SE 7. Bol Epidemiol. 2015;24(7):146-150.
13. Vargas I. Reporte de Vigilancia de Rabia, Perú, SE 52-2017. Bol Epidemiol. 2017;26(52):1630-1632.
14. Vigilato MAN, Clavijo A, Knobl T, Silva HMT, Cosivi O, Schneider MC, et al. Progress towards eliminating canine rabies: Policies and perspectives from Latin America and the Caribbean. Philos Trans R Soc B Biol Sci. 2013;368(1623):20120143. doi: 10.1098/rstb.2012.0143.
15. López R, Condori-Condori RE, Díaz A. Manual de procedimientos para el diagnóstico de la rabia. Lima: Ministerio de Salud-Instituto Nacional de Salud; 2002. Serie de Normas Técnicas: 31.
16. Streicker DG. Viral host shifts: ecological dynamics, cross-species transmission and host adaptation in bat rabies [dissertation]. Georgia: University of Georgia; 2011. 159 p.
17. Velasco-Villa A, Kuzmin I, Orciari LA. Detection of rabies virus nucleic acid in human and animal diagnostic fresh specimens by reverse-trans-cription polymerase chain reaction (RT-PCR). 2da ed. Atlanta: Centers for Disease Control and Prevention; 2012.
18. Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6: Molecular evolutionary genetics analysis version 6.0. Mol Biol Evol. 2013;30(12):2725-9.
19. Huang X, Madan A. CAP3: A DNA Sequence Assembly Program. Genome Res. 1999;9(9):868-877. doi: http://dx.doi.org/10.1101/ gr.9.9.868.
20. Thompson JD, Gibson TJ, Plewniak F, Jeanmougin F, Higgins DG. The CLUSTAL X windows interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Res. 1997;25(24):4876–82.
21. Favi M, Nina A, Yung V, Fernández J. Characterization of rabies virus isolates in Bolivia. Virus Res. 2003;97(2):135-40.
22. De Souza DN, Carnieli P, Macedo CI, de Novaes Oliveira R, de Carvalho Ruthner Batista HB, Rodrigues AC, et al. Phylogenetic analysis of rabies virus isolated from canids in North and Northeast Brazil. Arch Virol. 2017;162(1):71-7.
23. Carnieli P, Fahl W de O, Castilho JG, Oliveira R de N, Macedo CI, Durymanova E, et al. Characterization of Rabies virus isolated from canids and identification of the main wild canid host in Northeastern Brazil. Virus Res. 2008;131(1):33-46.
24. Nagarajan T, Nagendrakumar SB, Mohanasubramanian B, Rajalakshmi S, Hanumantha NR, Ramya R, et al. Phylogenetic analysis of nucleoprotein gene of dog rabies virus isolates from Southern India. Infect Genet Evol. 2009;9(5):976-82.
25. Kobayashi Y, Inoue N, Sato G, Itou T, Santos HP, Brito CJC, et al. Phylogenetic characterization of rabies virus isolates from Carnivora in Brazil. J Vet Med Sci. 2007;69(7):691-6.
26. Fèvre EM, Bronsvoort BMDC, Hamilton KA, Cleaveland S. Animal movements and the spread of infectious diseases. Trends Microbiol. 2006;14(3):125-31.
27. Bourhy H, Dacheux L, Strady C, Mailles A. Rabies in Europe in 2005. Euro Surveill. 2005;10:213-216. doi: https://doi.org/10.2807/es-m.10.11.00575-en.
28. Bourhy H, Reynes JM, Dunham E, Dacheux L, Larrous F, Huong V, et al. The origin and phylogeography of dog rabies virus. J Gen Virol. 2008;89(2673-2681.
29. Bourhy H, Kissi B, Smreczak M, Sadkowska-Todys M, Kulonen K, Tordo N, et al. Ecology and evolution of rabies virus in Europe. J Gen Virol. 1999;80(10):2545-57.
30. Castillo-Neyra R, Zegarra E, Monroy Y, Bernedo R, Cornejo-Rosello I, Paz-Soldan V, et al. Spatial Association of Canine Rabies Outbreak and Ecological Urban Corridors, Arequipa, Peru. Trop Med Infect Dis. 2017;2(3):38. doi: https://doi.org/10.3390/tropicalmed2030038.
31. Nadin-Davis S. Molecular Epidemiology. En Alan Jackson, editor. Rabies, Scientific Basis of the Disease and Its Management. 3ra ed. USA. Academic Press; 2013. p 123-124.
32. Wallace RM, Undurraga EA, Blanton JD, Cleaton J, Franka R. Elimination of Dog-Mediated Human Rabies Deaths by 2030: Needs Assessment and Alternatives for Progress Based on Dog Vaccination. Front Vet Sci. 2017;4:9. doi: 10.3389/fvets.2017.00009.
33. Fahrion AS, Taylor LH, Torres G, Müller T, Dürr S, Knopf L, et al. The Road to Dog Rabies Control and Elimination-What Keeps Us from Moving Faster?. Front Public Health. 2017;5:103. doi: 10.3389/fpubh.2017.00103
34. Mørk T, Prestrud P. Arctic Rabies – A Review. Acta vet scand. 2004;45(45):1-9.
Correspondencia: Carina Mantari Dirección: Cápac Yupanqui 1400, Jesús María, Lima, Perú.
Teléfono: 7481111 - anexo 2147
Email: cmantari@ins.gob.pe
Recibido: 17/09/2018
Aprobado: 27/02/2019
En línea: 21/03/2019