Artículo original
Polimorfismo genético de la Apolipoproteína E en una población peruana
Genetic polymorphism of Apolipoprotein E in a peruvian population
Victoria Marca1,a, Oscar Acosta2,b, Mario Cornejo-Olivas1,3,c, Olimpio Ortega1,b, Doris Huerta2,4,d, Pilar Mazzetti1,4,e
1 Servicio de Neurogenética, Instituto Nacional de Ciencias Neurológicas. Lima, Perú.
2 Centro de Investigación de Bioquímica y Nutrición, Facultad de Medicina, Universidad Nacional Mayor de San Marcos. Lima, Perú.
3 Washington Universtity International Fogarty Scholar´s Program. Lima, Perú.
4 Facultad de Medicina Humana, Universidad Nacional Mayor de San Marcos. Lima, Perú.
a Ingeniera Química; b Biólogo, genetista; c Médico neurólogo; d Biólogo, Magíster en Bioquímica; e Médico neurólogo, Magíster en Gestión Pública
RESUMEN
Objetivos. Determinar las frecuencias genotípicas y alélicas del gen APOE en una muestra poblacional peruana. Materiales y métodos. Estudio transversal analítico en 189 trabajadores voluntarios, aparentemente sanos, del Instituto Nacional de Ciencias Neurológicas en Lima, Perú, divididos en cinco grupos según departamento de origen y ascendencia en dos generaciones. El ADN genómico fue amplificado mediante PCR-RFLP. Se realizó la detección de los fragmentos resultantes por electroforesis en gel de poliacrilamida al 12 %. Resultados. El alelo ε3 es el más frecuente en todos los grupos (93,9 %), con bajas frecuencias de los alelos ε4 (5 %) y ε2 (1,1 %). El análisis de heterocigosidad (H) en cada grupo muestra una diversidad intermedia entre 10 y 20 %. Las diversidades genéticas poblacional (Ht) e intrapoblacional (Hs), son 14,4 y 14,3 % respectivamente, sugiriendo proximidad genética entre los grupos estudiados para el polimorfismo ApoE. Conclusiones. Las frecuencias alélicas del gen ApoE encontradas muestra que el alelo ε3 tiene una de las frecuencias más altas y, el alelo ε4, una de las más bajas respecto a otros grupos poblacionales del mundo, con posibles implicancias en el riesgo para enfermedades neurológicas, cardiovasculares y otras en nuestro país.
Palabras clave: Alelos; Apolipoproteínas E; Frecuencia de los genes (fuente: DeCS BIREME).
ABSTRACT
Objectives. To determine the allelic and genotypic frequencies of the APOE gene in a sample of a population group in Peru. Materials and methods. Cross-sectional analytic study in 189 apparently healthy volunteers, workers of the Instituto Nacional de Ciencias Neurológicas in Lima, Perú, divided into 5 groups by birth department and two generations ancestry. Genomic DNA was amplified using PCR-RFLP. The resulting fragments were detected by 12 % polyacrylamide gel electrophoresis. Results. The ε3 allele is the most frequent in all the groups (93.9 %), with low ε4 (5 %) and ε2 (1.1 %) allele frequencies. The analysis of heterozygosity (H) for each group displays intermediate diversity between 10 and 20%. Population genetic diversity (Ht) and diversity within populations (Hs) are 14.43 % and 14.31% respectively, suggesting genetic proximity between the studied groups for the ApoE polymorphism. Conclusions. Allele frequencies of the ApoE gene found show that allele ε3 has one of the highest frequencies and ε4 allele one of the lowest compared to other population groups in the world, with possible implications in the risk of neurological, cardiovascular and other diseases in our country.
Key words: Alleles; Apolipoproteins E; Gene frequency (source: MeSH NLM).
INTRODUCCIÓN
La apolipoproteína E (ApoE) es una glicoproteína de 34 kDa y 229 aminoácidos que se encuentra formando parte de los quilomicrones, lipoproteínas de muy baja densidad (VLDL) y lipoproteínas de alta densidad (HDL), participa en el transporte y el metabolismo de lípidos plasmáticos al actuar como ligando de las lipoproteínas de baja densidad (LDL) y de los receptores de ApoE (1,2). El gen de la apolipoproteína E en el humano está localizado en el cromosoma 19 y la variación genética en este locus está determinada por tres alelos: ε2, ε3 y ε4, cuya combinación genera seis genotipos, siendo el alelo ε3 el más frecuente en todas las poblaciones mientras que el ε4 es el alelo considerado ancestral, presente en primates como homocigotos E4/E4 y en seres humanos en los que por mutación aparecen posteriormente los alelos E2 y E3 (3,4). Las diferencias en los residuos 112 y 158 de la secuencia aminoacídica -determinada por la transición citosina/timina en la secuencia del gen y el consiguiente intercambio entre una arginina y la cisteína- son la base para la formación de las tres isoformas proteicas: ε2 (Cys112, Cys158), ε3 (Cys112, Arg158) y ε4 (Arg112, Arg158) (5).
Diversos estudios clínicos y epidemiológicos han aportado evidencias sobre la relación entre los alelos y los genotipos de la ApoE con diferentes condiciones patológicas, principalmente del metabolismo de los lípidos, enfermedades cardiovasculares y neurológicas, entre otras (5,6). La presencia de, por lo menos, un alelo ε4 se considera un factor de riesgo para la enfermedad de Alzheimer y otras demencias (7).
El locus ApoE es polimórfico y muestra una variación alélica que va del 0 al 20 % para el E2; 60 al 90 % para el E3 (el más frecuente) y 10 al 20 % para el E4 en diferentes poblaciones humanas, con algunas excepciones (7,8). Dado que la población peruana es de origen multiétnico y presenta un alto grado de mestizaje, y que no existe información sobre la prevalencia de polimorfismos de ApoE en esta población, se realizó un estudio para determinar las frecuencias genotípicas y alélicas del gen APOE en una muestra poblacional peruana.
MATERIALES Y MÉTODOS
Se realizó un estudio transversal analítico. Se calculó el tamaño de muestra para proporciones con una frecuencia esperada de 7,4 % (mestizos mexicanos) para el alelo E4 (8) y un 13 % por posibles pérdidas o fallas técnicas. Para el análisis molecular se incluyeron 189 muestras de ADN de individuos voluntarios, aparentemente sanos, residentes en Lima, de un total de 750 trabajadores del Instituto Nacional de Ciencias Neurológicas que siguen evaluaciones médicas de rutina por ser trabajadores de salud; todos ellos sin relación de parentesco, con información de su origen según departamento y ascendencia hasta dos generaciones (tanto el participante como sus padres y cuatro abuelos eran nacidos en el departamento considerado de origen). Todos los participantes firmaron el consentimiento informado aprobado por el Comité de Ética Institucional.
Para la evaluación genética, el ADN genómico fue extraído de muestras de epitelio bucal, utilizando buffer de lisis y shock térmico (100 °C) y ulterior resuspensión en Tris EDTA. Se determinaron los diferentes genotipos del ApoE mediante la técnica PCR-RFLP amplificándose una secuencia de 227 pb, empleando cebadores (primers) específicos que flanquean al gen: 5’ TCCAAGGAGCTGCAGGCGGCGCA 3’ y 5’ ACAGAATTCGCCCCGGCCTGGTACACTGCCA 3’ con las siguientes condiciones: 94 °C por 3 min, 30 ciclos (94 °C por 30 min para la denaturación, 65 °C por 30 min para el alineamiento y 70 °C por 1,5 min para la extensión) y, finalmente, 70 °C por 10 min (9). Los productos amplificados fueron digeridos con la enzima HhaI por cuatro horas, sometidos a electroforesis en geles de poliacrilamida al 12 % y teñidos con nitrato de plata, generándose bandas características para cada genotipo: ε2/ε2 (91, 83, y 35 pb), ε3/ε3 (91, 48 y 35 bp), ε4/ε4 (72, 48 y 35 bp), ε2/ε3 (91, 83, 48 y 35 pb), ε2/ ε4 (91,83, 72, 48 y 35 pb) y ε3/ε4 (91, 72, 48 y 35 pb).
Las frecuencias genotípicas y alélicas fueron obtenidas por conteo directo. Se aplicó el test exacto (10) para evaluar las frecuencias observadas según lo esperado bajo la hipótesis del equilibrio de Hardy-Weinberg. Se realizó un análisis de diversidad genética: H (heterocigosidad para cada grupo poblacional), Ht (diversidad genética en la población total), Hs (diversidad genética promedio intrapoblacional) y Gst (diferenciación genética entre poblaciones respecto a la diversidad genética total) según lo descrito por Nei et al. (11).
Para comparar las frecuencias alélicas de la ApoE en nuestra población (n=189) con las informadas en otras poblaciones del mundo, se usó la prueba X2 o el test exacto de Fisher según corresponda (α = 0,05) considerando las categorías presencia y ausencia del alelo ε4. Para el análisis estadístico se usó el paquete SPSS versión 15.0 y el programa de libre acceso Arlequín v3.11 para genética poblacional (cmpg.unibe. ch/software/ arlequin3/) (12).
RESULTADOS
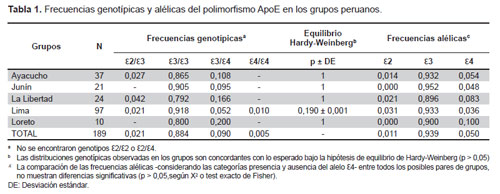
El grupo estudiado está constituido por 66 varones (34,9 %) y 123 mujeres (65,1 %), con edades entre 18 y 80 años (43,2 ± 13,1), diez personas (5,3 %) tuvieron 60 o más años. Los participantes fueron categorizados en grupos según origen por departamento de nacimiento y ascendencia en las dos últimas generaciones; se encuentran representados cinco departamentos de nuestro país de las tres áreas geográficas: 121 personas de la costa (Lima y La Libertad), 58 de la sierra (Ayacucho y Junín) y diez de la selva (Loreto). La distribución de las frecuencias genotípicas y alélicas del gen APOE encontrada en los cinco grupos se describen en Tabla 1.
Las distribuciones genotípicas observadas en los grupos, son concordantes con lo esperado bajo la hipótesis de equilibrio de Hardy-Weinberg (p > 0,05), según test exacto de Guo y Thompson. El genotipo ε3/ε3 es el más frecuente en todos los grupos, representando el 88,4 % del total. Los genotipos ε2/ε2 y ε2/ε4 no se encontraron en ninguno de los grupos, y el genotipo ε4/ε4 alcanza solo un 0,5 % de total de individuos estudiados. El alelo ε3 es el más frecuente en todos los grupos (93,9 %), seguido del ε4 (5 %) y del ε2 (1,1 %), sin diferencias significativas (p>0,05) al comparar las frecuencias alélicas entre pares de grupos.
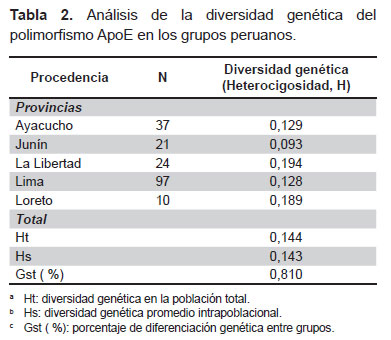
La evaluación de la diversidad genética en cada grupo (heterocigosidad H) nos muestra que el grupo de La Libertad es el más diverso (19,4 %) para el polimorfismo ApoE, siendo el grupo de Junín el menos diverso (9,3 %). Tomados los cinco grupos como una población única para evaluar su diversidad genética (Ht), el valor es 14,4 %, muy similar a la diversidad genética promedio intrapoblacional (HS=14,3 %). La diferenciación genética entre los cinco grupos (Gst) es menor al 1 %, lo que indica similitud entre ellos, como se ve en la Tabla 2. En la Tabla 3 se comparan las frecuencias alélicas del gen ApoE en la población de estudio con las descritas para otras poblaciones del mundo, encontrándose diferencias significativas (p < 0,05) con la mayoría de poblaciones al comparar el alelo ε4.
DISCUSIÓN
El gen ApoE ha sido estudiado en muchas poblaciones, pues la presencia de al menos un alelo ε4 está considerado como factor asociado o de riesgo en enfermedades neurodegenerativas y cardiovasculares, entre otras enfermedades (13-15). La composición étnica de la población peruana es compleja, con importante mestizaje, conformada por poblaciones originarias diversas con componente ulterior europeo, africano y asiático. No hemos encontrado trabajos previos de la distribución del polimorfismo de este gen en nuestro medio.
Respecto a la distribución de genotipos en los cinco grupos, el ε3/ε3 es el más frecuente; encontramos una baja frecuencia de ε4/ε4, solo presente en Lima y no encontramos ε2/ε2 y ε2/ε4 en ninguno de los grupos. Estos resultados concuerdan con lo observado en otras poblaciones amerindias nativas y mestizas (16,17) y sigue la tendencia observada para la distribución de los genotipos ApoE en el mundo, es decir: ε3/ε3 > ε3/ε4 > ε2/ε3 (8,18).
Respecto a la distribución de alelos, el ε3 es el más frecuente en los cinco grupos (93,9 %), siendo una de las frecuencias más altas en el mundo mientras que el alelo ε2 es el menos frecuente (1,1 %) (Tabla 3). Considerando la alta frecuencia de ε3 y la baja frecuencia de ε2 en nuestra muestra, al igual que en nativos de Sudamérica, ello podría orientar a una historia evolutiva en común tal y como han propuesto otros investigadores (8,16,18,19). Encontramos una frecuencia de 5 % para el alelo ε4, similar a poblaciones de Asia y la India; aunque mucho menor si la comparamos con poblaciones de Africa y Oceanía (8,16). Ello probablemente este en relación con la ancestralidad del alelo ε4. Los alelos ε2 y ε3 se encuentran cada vez más representados en las diferentes poblaciones humanas en la medida en que acceden a alimentación de mejor calidad y constantemente disponible (4,8).
La diversidad genética en esta población peruana (Ht), y la diversidad genética promedio intrapoblacional (Hs) son 14,43 % y 14,31 % respectivamente, valores menores a los descritos en poblaciones de África (47 y 44 %), Europa (35 y 34 %), Asia (27 y 26 %), Norteamérica (30 y 29 %), Sudamérica (37 y 34 %) y Oceanía (49 y 44 %) (8); todo ello sugiere una diversidad relativamente baja en esta población peruana para el polimorfismo ApoE. La diferenciación genética entre los cinco grupos (Gst) es tan solo del 0,81 %, considerada baja comparado con las poblaciones sudamericanas y de Oceanía (7,9 y 9,1 % respectivamente) e inclusive por norteamericanas y de la India con valores entre 1,2 y 4,6 % (8), lo cual sugiere similitud o proximidad genética entre los grupos estudiados.
Las frecuencias alélicas del gen ApoE encontradas en nuestro estudio, siguen la tendencia observada en otras poblaciones y países, es decir: ε3 > ε4 > ε2 (8,18). La frecuencia del alelo ε3 es de 93,9 %, una de las más altas descritas; solo los xavante en el Brasil (98 %) y la región de Koch en la India (96,8 %) tienen una frecuencia algo mayor (8,16).
Al analizar la frecuencia del alelo ε4 por categorías y su presencia y ausencia en esta población peruana, comparada con las del continente americano, no encontramos diferencias con los xavante de Brasil, una población colombiana y mestizos de México y Costa Rica. Por el contrario, encontramos diferencias (p < 0,05) con los nativos wai wai y mestizos de Brasil, una población chilena, nativos cayapa de Ecuador, una población cubana y norteamericanos de Pensilvania (8, 16, 20-22).
Cuando se compara la presencia del alelo ε4 en esta población peruana con la de otros continentes (Tabla 3), se observan diferencias con poblaciones de África, Europa y Oceanía (23-27). Con respecto a las poblaciones asiáticas, las frecuencias alélicas son diferentes a la de India y Japón; no encontramos diferencias con China, Corea, Taiwan e Irán.
El alelo ε4 tiene una de las frecuencias más bajas respecto a la reportadas en otras regiones del mundo, lo cual puede tener implicancias en el riesgo de enfermedades como el Alzheimer, que es el tipo de demencia más frecuente en nuestro país (28). Las diferencias étnicas observadas significan que cada grupo poblacional debe conocer sus propias características genéticas para respaldar las decisiones de salud pública.
Un estudio alemán sobre extracción de ADN en tejido óseo de cinco momias de la costa peruana, que analiza el gen APOE, encontró los tres alelos representados, aunque por el tamaño de la muestra no se puede inferir distribución de frecuencias (29).
El grupo estudiado es relativamente pequeño y no incluye personas de todas las etnias que forman parte de nuestro país, limitando la representatividad de la muestra para la población peruana, pues solo se ha incluido a personas de cinco departamentos de las tres regiones del país, sin relación de parentesco y con permanencia familiar en el lugar de origen por dos generaciones. Si bien faltan incluir personas de otros lugares, los índices de diversidad genética indican similaridad para el polimorfismo ApoE estudiado. Otra limitante es la ausencia de descarte sistemática de deterioro cognitivo u otras condiciones neurológicas asociadas al alelo 4 de ApoE en las evaluaciones de salud de rutina. En futuros trabajos se requiere una muestra mucho mayor, que incluya todos los departamentos y grupos étnicos del país.
En conclusión, el genotipo ε3/ε3 y el alelo ε3 son los más frecuentes, y los alelos E4 y E2 son poco frecuentes en la población estudiada. Esta característica se observa también en cada uno de los cinco grupos determinados por el lugar de nacimiento. En comparación a otras regiones del mundo el alelo E3 tiene una de las frecuencias más altas y el alelo E4 una de las más bajas. Los resultados de esta muestra poblacional peruana contribuyen a un mejor conocimiento de la variabilidad geográfica y étnica del gen APOE en el mundo (3, 30). Este estudio nos aproxima a la distribución del polimorfismo del gen APOE en población peruana, y puede aportar información útil para estudios de asociación genética con diversas enfermedades, su interacción con otros genes y factores ambientales en nuestro medio.
AGRADECIMIENTOS
Al biólogo Raúl Tito, por su apoyo logístico. A los Drs. Silvia Montano (NAMRU-6, Lima) y Joe Zunt (UW, Seattle), coordinadores del Programa Fogarty en el Perú por su apoyo en la formación de investigadores. Al Dr. Ignacio F. Mata por la revisión crítica del informe final.
Contribución de autoría
VM, OA, MCO, OO, DH y PM participaron en la concepción del estudio, recolección de resultados, análisis e interpretación de datos, redacción del manuscrito, revisión crítica del artículo y la aprobación de la versión final a publicar. PM y MCO contribuyeron en recolección de datos y diseño del estudio, VM y OO contribuyeron en el procesamiento de las muestras, OA y DH contribuyeron en la creación de la base de datos y el análisis estadístico para genética de poblaciones.
Fuentes de financiamiento
Este trabajo ha recibido el apoyo del National Institutes of Health Fogarty International Clinical Research Fellows Program at Vanderbilt University (R24 TW007988) y el Instituto Nacional de Ciencias Neurológicas.
Conflictos de interés
Los autores declaran no tener conflictos de interés en la publicación de este artículo.
REFERENCIAS BIBLIOGRÁFICAS
1. Mahley RW. Apolipoprotein E: cholesterol transport protein with expanding role in cell biology. 1988;240(4852):622-30.
2. Jiang Q, Lee CY, Mandrekar S, Wilkinson B, Cramer P, Zelcer N, et al. ApoE promotes the proteolytic degradation of Abeta. Neuron. 2008;58(5):681-93.
3. Fullerton SM, Clark AG, Weiss KM, Nickerson DA, Taylor SL, Stengard JH, et al. Apolipoprotein E variation at the sequence haplotype level: implications for the origin and maintenance of a major human polymorphism. Am J Hum Genet. 2000;67(4):881-900.
4. Corbo RM, Scacchi R. Apolipoprotein E (APOE) allele distribution in the world. Is APOE*4 a ‘thrifty’ allele? Ann Hum Genet. 1999;63(Pt 4):301-10.
5. Smith JD. Apolipoprotein E4: an allele associated with many diseases. Ann Med. 2000;32(2):118-27.
6. Bennet AM, Di Angelantonio E, Ye Z, Wensley F, Dahlin A, Ahlbom A, et al. Association of apolipoprotein E genotypes with lipid levels and coronary risk. JAMA. 2007;298(11):1300-11.
7. St George-Hyslop PH, Petit A. Molecular biology and genetics of Alzheimer’s disease. C R Biol. 2005;328(2):119- 30.
8. Singh PP, Singh M, Mastana SS. APOE distribution in world populations with new data from India and the UK. Ann Hum Biol. 2006;33(3):279-308.
9. Hixson JE, Vernier DT. Restriction isotyping of human apolipoprotein E by gene amplification and cleavage with HhaI. J Lipid Res. 1990;31(3):545-8.
10. Guo SW, Thompson EA. Performing the exact test of Hardy-Weinberg proportion for multiple alleles. Biometrics. 1992;48(2):361-72.
11. Nei M. Analysis of gene diversity in subdivided populations. Proc Natl Acad Sci USA. 1973;70(12):3321-3.
12. Excoffier L, Laval G, Schneider S. Arlequin (version 3.0): an integrated software package for population genetics data analysis. Evol Bioinform Online. 2005;1:47-50.
13. Verghese PB, Castellano JM, Holtzman DM. Apolipoprotein E in Alzheimer’s disease and other neurological disorders. Lancet Neurol. 2011;10(3):241-52.
14. Payami H, Kaye J, Heston LL, Bird TD, Schellenberg GD. Apolipoprotein E genotype and Alzheimer’s disease. Lancet. 1993;342(8873):738.
15. Cattin L, Fisicaro M, Tonizzo M, Valenti M, Danek GM, Fonda M, et al. Polymorphism of the apolipoprotein E gene and early carotid atherosclerosis defined by ultrasonography in asymptomatic adults. Arterioscler Thromb Vasc Biol. 1997;17(1):91-4.
16. de Andrade FM, Coimbra CE, Jr., Santos RV, Goicoechea A, Carnese FR, Salzano FM, et al. High heterogeneity of apolipoprotein E gene frequencies in South American Indians. Ann Hum Biol. 2000;27(1):29-34.
17. Demarchi DA, Salzano FM, Altuna ME, Fiegenbaum M, Hill K, Hurtado AM, et al. APOE polymorphism distribution among Native Americans and related populations. Ann Hum Biol. 2005;32(3):351-65.
18. Aceves D, Ruiz B, Nuno P, Roman S, Zepeda E, Panduro A. Heterogeneity of apolipoprotein E polymorphism in different Mexican populations. Hum Biol. 2006;78(1):65-75.
19. Cavalli-Sforza LL, Menozzi P, Piazza A. The history and geography of human genes. Princeton, NJ: Princeton University; 1994. 1088 p.
20. Quiroga P, Calvo C, Albala C, Urquidi J, Santos JL, Perez H, et al. Apolipoprotein E polymorphism in elderly Chilean people with Alzheimer’s disease. Neuroepidemiology. 1999;18(1):48-52.
21. Jacquier M, Arango D, Villareal E, Torres O, Serrano ML, Cruts M, et al. APOE epsilon4 and Alzheimer’s disease: positive association in a Colombian clinical series and review of the Latin-American studies. Arq Neuropsiquiatr. 2001;59(1):11-7.
22. Gamboa R, Vargas-Alarcon G, Medina-Urrutia A, Cardoso-Saldana G, Hernandez-Pacheco G, Zamora- Gonzalez J, et al. Influence of the apolipoprotein E polymorphism on plasma lipoproteins in a Mexican population. Hum Biol. 2001;73(6):835-43.
23. Scacchi R, Corbo RM, Rickards O, Mantuano E, Guevara A, De Stefano GF. Apolipoprotein B and E genetic polymorphisms in the Cayapa Indians of Ecuador. Hum Biol. 1997;69(3):375-82.
24. Ganguli M, Chandra V, Kamboh MI, Johnston JM, Dodge HH, Thelma BK, et al. Apolipoprotein E polymorphism and Alzheimer disease: The Indo-US Cross-National Dementia Study. Arch Neurol. 2000;57(6):824-30.
25. Haddy N, De Bacquer D, Chemaly MM, Maurice M, Ehnholm C, Evans A, et al. The importance of plasma apolipoprotein E concentration in addition to its common polymorphism on inter-individual variation in lipid levels: results from Apo Europe. Eur J Hum Genet. 2002;10(12):841-50.
26. Hu P, Qin YH, Jing CX, Lu L, Hu B, Du PF. Does the geographical gradient of ApoE4 allele exist in China? A systemic comparison among multiple Chinese populations. Mol Biol Rep. 2011;38(1):489-94.
27. Bazrgar M, Karimi M, Fathzadeh M, Senemar S, Peiravian F, Shojaee A, et al. Apolipoprotein E polymorphism in Southern Iran: E4 allele in the lowest reported amounts. Mol Biol Rep. 2008;35(4):495-9.
28. Custodio N, Montesinos R, Escobar J, Bendezú L. Prevalencia de demencia en una población urbana de Lima-Perú: estudio puerta a puerta. An Fac med. 2008;69(4):233-8.
29. Wiechmann I. Molecular genetic analysis of the polymorphic apolipoprotein E in modern and ancient human DNA samples. Anthropol Anz. 2000;58(1):93-8.
30. Eisenberg DT, Kuzawa CW, Hayes MG. Worldwide allele frequencies of the human apolipoprotein E gene: climate, local adaptations, and evolutionary history. Am J Phys Anthropol. 2010;143(1):100-11.
Correspondencia:
María Victoria Marca Ysabel
Dirección: Jr. Ancash 1271, Lima 1, Perú.
Teléfono: (511) 4117779
Correo electrónico: peru.neurogenetica@gmail.com
Recibido: 27-07-11
Aprobado: 26-10-11