Revisión
Mecanismos moleculares de resistencia antibiótica en Escherichia coli asociadas a diarrea
Molecular mechanisms of antibiotic resistance in Escherichia coli- associated diarrhea
Susan Mosquito1,2,a, Joaquim Ruiz3,4,5,b, José Luis Bauer2,c, Theresa J. Ochoa1,6,7,d
1 Instituto de Medicina Tropical Alexander von Humbold, Universidad Peruana Cayetano Heredia. Lima, Perú.
2 Facultad de Ciencias y Filosofía Alberto Cazorla Talleri, Universidad Peruana Cayetano Heredia. Lima, Perú.
3 Centre de Recerca en Salut Internacional de Barcelona, Hospital Clinic, Universitat de Barcelona. Barcelona, España.
4 Institut d’investigacions Biomèdiques August Pi i Sunyer. Barcelona, España.
5 Centro de Investigación Biomédica en Red de Epidemiología y Salud Pública. Barcelona, España.
6 School of Public Health, University of Texas. Texas, EE.UU.
7 Facultad de Medicina Alberto Hurtado, Universidad Peruana Cayetano Heredia. Lima, Perú.
a Bióloga, MSc. en Microbiología; b Biólogo, PhD en Biología; c Microbiólogo, PhD en Ciencias con mención en Microbiología; d Médico, Pediatra Infectólogo
RESUMEN
La resistencia antibiótica es un problema emergente a nivel mundial presente en diversas bacterias, en especial en la Escherichia coli, que tiene altos porcentajes de resistencia hacia ampicilina, trimetoprim-sulfametoxazol, tetraciclina, cloramfenicol y ácido nalidíxico, lo que supone grandes complicaciones en el tratamiento antibiótico cuando este es requerido. Este aumento de resistencia antibiótica se debe a la adquisición de diferentes mecanismos moleculares de resistencia mediante mutaciones puntuales a nivel cromosómico o transferencia horizontal de material genético entre especies relacionadas o diferentes, facilitada por algunos elementos genéticos tales como los integrones. Esta revisión discute los efectos de los mecanismos moleculares de resistencia más comunes en E.coli: inactivación enzimática, alteraciones en el sitio blanco y alteraciones de la permeabilidad. El conocer los mecanismos de resistencia implicados, como lo recomienda la Organización Mundial de la Salud, permitirá optimizar la vigilancia de resistencia y las políticas de control y uso de antibióticos a nivel nacional.
Palabras clave: Resistencia bacteriana a antibióticos; Agentes antibacterianos; Escherichia coli (fuente: DeCS BIREME).
ABSTRACT
Antibiotic resistance is an emerging problem worldwide present in many bacteria, specially in Escherichia coli, which has high percentages of resistance to ampicilline, thrimethoprim-sulfamethoxazole, tetracycline, chloramphenicol and nalidixic acid, which implies important complications in antibiotic treatment when required. The increasing antibiotic resistance is due to the acquisition of different molecular mechanisms of resistance through point chromosomal mutations and /or horizontal transfer of genetic material between related or different species facilitated by some genetic elements such as integrons. This review discusses the effects of the most common molecular mechanisms of antibiotic resistance in E. coli: enzymatic inactivation, changes in the target site and permeability disturbances. Getting to know the mechanisms of resistance which are involved, as the World Health Organization recommends, will allow us to improve the surveillance of the antibiotic resistance, the control policies and the antibiotic utilization at a national level.
Key words: Drug resistance, bacterial; Anti-bacterial agents; Escherichia coli (source: MeSH NLM).
INTRODUCCIÓN
La resistencia antibiótica es un fenómeno biológico natural debido a las mutaciones y a la gran capacidad de las bacterias de transferir horizontalmente su material genético, existiendo una clara correlación entre el uso de antibióticos y la resistencia bacteriana (1). Se constituye así en un problema a nivel mundial que supone mayor sufrimiento humano, pérdida en la productividad y mortalidad. La relación antibiótico-bacteria se ve alterada por otros múltiples factores como la farmacocinética de la droga, la dosis, la duración del tratamiento, el tamaño de inóculo bacteriano, etc., por lo que para optimizar el uso de estos fármacos, se realizan supervisiones periódicas de la resistencia como parte transcendental de la política de control de la resistencia antibiótica (1,2). Según la OMS (Organización Mundial de la Salud), existen principalmente dos métodos de supervisión de resistencia: in vivo (fracasos terapéuticos) o in vitro (estudios en el laboratorio, en “tubos de ensayo”). Dentro de los métodos in vitro, están los fenotípicos (Kirby-Bauer o concentración mínima inhibitoria) y los moleculares cuando se hacen pruebas genéticas. La vigilancia molecular, se basa en la identificación de genes que codifican los mecanismos de resistencia en las bacterias y que representan el futuro de la vigilancia de resistencia antibiótica. Estos genes surgen ante el uso de un antibiótico específico, sin embargo, pueden generar también resistencia cruzada, afectando otros antibióticos de la misma clase o con el mismo mecanismo de acción, incluso a compuestos de familias diferentes (1). Asimismo, en numerosas ocasiones genes de resistencia a diferentes antimicrobianos se encuentran asociados en una misma estructura genética (integrones) (3), lo que da lugar al fenómeno de coselección de resistencia (mantenimiento en la bacteria de genes capaces de conferir resistencia a un antimicrobiano, mediante el uso de un agente antimicrobiano no emparentado). De esta manera conocer el bagaje de genes de resistencia contribuye a tomar mejores políticas de uso antibiótico que intenten controlar la diseminación de estos genes.
En el Perú, la vigilancia de resistencia se realiza de manera in vitro mediante métodos fenotípicos, donde se determina la sensibilidad o resistencia antibiótica, siendo muy pocos los estudios de investigación realizados para la búsqueda de genes de resistencia antibiótica y aun menos los dirigidos hacia enteropatógenos específicamente. Dentro de los pocos estudios realizados en el país, se muestra por ejemplo la diseminación de las beta-lactamasas de espectro extendido (BLEE) tipo CTX-M en Escherichia coli aisladas de niños sanos menores de cuatro años (4).
Para la mayoría de episodios diarreicos asociados con las E. coli diarrogénicas, no es recomendado el tratamiento antibiótico debido a que el uso excesivo de antibióticos está relacionado con el aumento de resistencia y, en el caso específico de la E. coli O157 relacionado con el desarrollo del síndrome urémico-hemolítico (SUH) (5). Sin embargo, en casos específicos como la diarrea del viajero causada por E. coli enterotoxigénica (ECET), su uso es recomendado (6). Por otro lado, el estudio de los mecanismos moleculares de resistencia en E. coli comensales, propias de la flora intestinal, es importante debido a que estas bacterias estarían actuando como reservorio de los genes de resistencia antibiótica distribuidos en la comunidad (7) siendo un reflejo de la exposición comunitaria a los antibióticos.
Dada la creciente resistencia antibiótica en E. coli y su impacto actual a nivel mundial, así como el creciente número de publicaciones sobre resistencia antibiótica a nivel nacional, consideramos de importancia revisar los principales mecanismos moleculares de resistencia en las E. coli con la finalidad de proporcionar a los microbiólogos, biólogos, médicos, epidemiólogos y demás personas del área biomédica, información actualizada y resumida sobre estos mecanismos para poder así conocer e interpretar mejor este problema en la literatura médica.
MECANISMOS MOLECULARES DE RESISTENCIA A ANTIBIÓTICOS
El fenotipo de resistencia antibiótica es perceptible gracias a la presencia de uno o más mecanismos moleculares de resistencia antibiótica en la bacteria. Dentro de los tipos de mecanismos moleculares de resistencia destacan por su relevancia: inactivación enzimática, alteraciones en el sitio blanco y alteraciones de la permeabilidad (8).
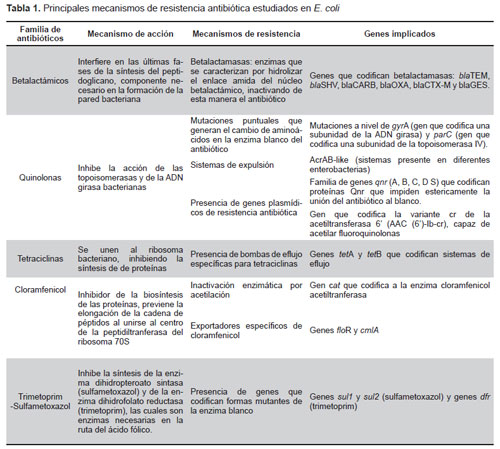
Las enterobacterias a nivel mundial presentan alta resistencia hacia ampicilina (beta-lactámico), trimetoprim-sulfametoxazol, tetraciclina, cloramfenicol y ácido nalidíxico por lo que se debe determinar la prevalencia de los diferentes genes relacionados a nivel molecular, como se ve en la Tabla 1.
ANTIBIÓTICOS BETA-LACTÁMICOS
La ampicilina es un beta-lactámico de espectro moderado, cuyo mecanismo de acción se da al interferir en las últimas fases de la síntesis del peptidoglicano, componente necesario en la formación de la pared bacteriana. Uno de los principales mecanismos de resistencia hacia beta-lactámicos es la hidrólisis enzimática, que es debida a la presencia de enzimas llamadas “beta-lactamasas” que se caracterizan por hidrolizar el enlace amida del núcleo beta-lactámico, inactivando de esta manera el antibiótico antes de que genere cualquier efecto. Estas enzimas constituyen una amplia familia que según la clasificación de Bush 2010 (9), se dividen en los grupos 1, 2 y 3, que a su vez están divididos en 16 subgrupos. El número de beta-lactamasas actualmente descrito es sumamente elevado, incrementándose de manera continua (10). Dentro de las más de 890 beta-lactamasas que actualmente se han caracterizado, las familias más comunes dentro de las enterobacterias son: blaTEM, blaSHV, blaOXA-1 y blaCARB. Las dos primeras pertenecientes al grupo 2b, es decir son penicilasas, inhibidas por el ácido clavulánico y que en algunos casos también tienen acción contra cefalosporinas de tercera generación. En el caso de blaOXA-1, pertenece al grupo 2d donde están las penicilasas que se caracterizan por la hidrólisis de cloxacilina (oxacilina); por su parte blaCARB pertenece al grupo 2c, el cual se caracteriza por tener penicilasas con acción hidrolítica contra carbenicilina (9). La clasificación de las betalactamasas se resume en la Tabla 2.
En los últimos años se ha descrito la creciente importancia clínica en enterobacterias de la familia CTX-M (11), un grupo de betalactamasas de espectro extendido que afecta la actividad de cefalosporinas de tercera y cuarta generación. Un ejemplo de la emergencia de estas beta-lactamasas CTX-M es un estudio realizado en 32 cepas de E. coli BLEE resistentes a cefalosporinas aisladas de pacientes pediátricos hospitalizados en Túnez, se informó una frecuencia del 97 % de blaCTX-M-15 (12).
Otro grupo de betalactamasas descritas por primera vez en Pseudomonas aeruginosa y descritas actualmente en E. coli son las blaGES (13) que se caracterizan por hidrolizar a la ceftazidima (14). En el caso de E. coli esta enzima ha sido relacionada con el integrón clase 3, clase menos frecuente respecto a la clase 1 y la clase 2 (13).
Un estudio en Suecia en E. coli comensales resistentes a ampicilina aisladas de 128 niños menores de un año, muestran que blaTEM (27/32) es predominante sobre blaSHV (5/32) y blaOXA (1/32) (15). En el caso de las E.coli diarrogénicas los estudios de beta-lactamasas son escasos, sin embargo, un estudio realizado en Uruguay determinó la presencia de la beta-lactamasa TEM-116, identificada como una beta-lactamasa de espectro extendido en trece cepas de E. coli Enteropatógena (16). Otro estudio reciente (17) en cepas de E. coli peruanas aisladas de niños menores de dos años confirma que blaTEM es predominante en la población (30,5 %), respecto al resto de las beta-lactamasas estudiadas: blaSHV (4,5 %), blaCARB (1,9 %) y blaOXA (5,8 %).
ANTIBIÓTICOS QUINOLONAS
El mecanismo de acción de las quinolonas es la inhibición de la acción de las topoisomerasas tipo II (ADN girasa y topoisomerasa IV). En bacterias Gram-negativas la ADN girasa, es el blanco principal mientras que la topoisomerasa IV actuaría como diana secundaria. La ADN girasa es una enzima compuesta por cuatro subunidades dos tipo A y dos tipo B codificadas por gyrA y gyrB respectivamente, y que es la encargada de catalizar el superenrrollamiento negativo del ADN. En el caso de la topoisomerasa IV, blanco principal en el caso de las Gram positivas, se trata también de una enzima con cuatro subunidades codificadas por parC y parE, homólogas a gyrA y gyrB respectivamente.
Entre los mecanismos moleculares de resistencia a estos antibióticos se encuentran las alteraciones en los blancos de quinolonas, las bombas de expulsión activa y la transferencia de genes de resistencia plasmídicos (18). El mecanismo mayormente descrito es el primero, donde mediante una mutación puntual, hay un cambio en el codón 83 y se codifica otro aminoácido de manera que se modifica la enzima blanco, con lo que se logra una alta resistencia a quinolonas como el ácido nalidíxico. La alta resistencia hacia fluoroquinolonas, tales como la ciprofloxacina, se relaciona con más de una mutación a nivel de gyrA (normalmente además de la posición 83 se afecta la posición 87) o con mutaciones además en gyrA a nivel de otros genes como parC (codones 80 o 84 de manera usual). A lo largo de los años se han informado las posiciones en donde ocurren las mutaciones y los cambios de aminoácidos que conlleva cada mutación (18), que están resumidas en la Tabla 3.
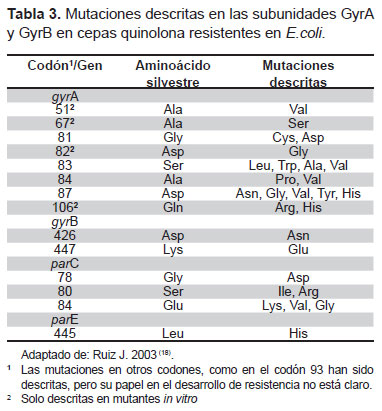
En un estudio reciente de E. coli aisladas de heces de pacientes hospitalizados en EE.UU., 353 cepas mostraron sensibilidad disminuida hacia fluoroquinolonas, teniendo el 85 % una o más mutaciones a nivel de gyrA y 46 % una o más mutaciones a nivel de parC (19).
Otro tipo de mecanismos de resistencia a quinolonas es el relacionado con la transferencia de genes mediante plásmidos, como es el caso de los genes qnr que codifican a la familia de las proteínas Qnr (QnrA, QnrB, QnrS, QnrC y QnrD) que se unen a la ADN girasa (gyrA y gyrB) y a la topoisomerasa IV (parC y parE) disminuyendo la acción de las quinolonas. El aumento de la resistencia a esta familia de antibióticos se puede relacionar con el aumento de CIM (concentración mínima inhibitoria) hacia diferentes quinolonas en E.coli, por lo que se habla de una “susceptibilidad disminuida”. Dado que en los últimos años se han descrito numeroso genes qnr, se ha efectuado una propuesta de nomenclatura (20) y se ha dispuesto una página web para recoger aquellos genes descritos hasta la fecha y los que surjan en un futuro (21).
La resistencia a fluoroquinolonas, específicamente a ciprofloxacino y norfloxacino, se ha relacionado con la variante cr de la acetiltransferasa 6’ (AAC (6’)-Ib-cr), que debido a la presencia de dos cambios de aminoácidos en los codones 102 y 179, es capaz de acetilar no solo aminoglúcosidos (AAC (6’)-Ib), sino también algunas fluoroquinolonas (22). La diseminación de esta acetiltranferasa ha sido descrita en cepas de E. coli aisladas de heces de pacientes hospitalizados en Uruguay (23) y en cepas de E. coli comensales aisladas de niños menores de cuatro años en Perú y Bolivia (7).
Clásicamente, el rol de las bombas de expulsión ha sido considerado como accesorio y, en general, de baja relevancia, no obstante, se ha comprobado que, para el caso de ácido nalidíxico las bombas de expulsión sí cumplen una función importante en el nivel basal de resistencia a este antimicrobiano (24). Un ejemplo de este tipo de mecanismo descrito en Escherichia coli (25) es el gen QepA, que codifica una bomba de expulsión para fluoroquinolonas hidrofílicas tales como norfloxacino, ciprofloxacino y enrofloxacino (23). Este gen ha sido descrito en cepas E. coli BLEE de México en una frecuencia de 1,7 % (26).
ANTIBIÓTICOS TETRACICLINAS
Las tetraciclinas son una familia de antibióticos cuyo mecanismo de acción es unirse a la parte 16S, de la subunidad 30S del ribosoma bacteriano, de manera que se inhibe la síntesis de proteínas al evitar la unión del aminoacil-tRNA en la posición A del ribosoma (27). La tetraciclina se ha utilizado durante décadas no solo en el tratamiento a humanos sino también en el tratamiento veterinario y como factor de crecimiento en aves (28). El mecanismo de resistencia más común hacia este antibiótico es mediante sistemas de eflujo, los que Gram negativos son codificados por los genes tetA, tetB, tetC, tetD, tetE, tetI y tetY, todos ellos previamente informados en E. coli. Sin embargo, existen otros mecanismos de resistencia como protección ribosomal y acción enzimática sobre las tetraciclinas codificados por diferentes genes (27,29), que están mostrados en la Tabla 4.
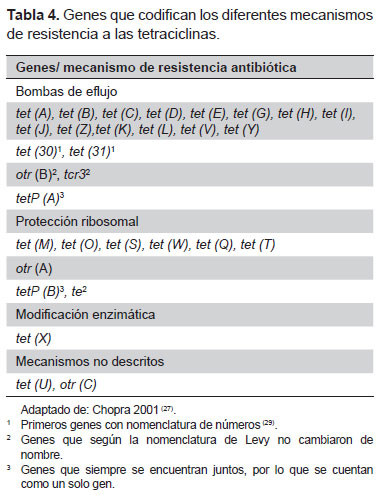
Un estudio llevado a cabo en cepas de E. coli comensales aisladas de infantes (Suecia), muestra que tetB y tetA fueron los genes de resistencia a tetraciclinas más prevalentes, 51 y 49 % respectivamente en un total de 37 cepas (28). Lo cual es importante debido a que las tetraciclinas no se utilizan en niños, sin embargo, estos genes fueron aislados de bacterias de la flora intestinal que están sirviendo de reservorio de resistencia antibiótica. En el estudio de Mosquito et al. (17), en un total de 106 cepas de E. coli peruanas tetraciclina resistentes se observó que tetA estuvo presente en el 26,4 % de las cepas, mientras que tetB estuvo presente en el 17,9 % de las cepas.
ANTIBIÓTICO CLORAMFENICOL
El cloramfenicol es utilizado en el tratamiento de humanos y animales, al ser un antimicrobiano de amplio espectro con acción sobre bacterias Gram positivas, bacterias Gram negativas, clamidias, micoplasmas y Rickettsias. Su mecanismo de acción es un fuerte y específico inhibidor de la biosíntesis de las proteínas, al prevenir la elongación de la cadena de péptidos uniéndose de manera reversible al centro de la peptidiltranferasa del ribosoma 70S (30).
Dentro de los mecanismos de resistencia a cloramfenicol, el más frecuente es el de la inactivación enzimática por acetilación mediante los diferentes tipos cloramfenicol acetiltransferasas (CAT) descritos. Estas CAT se dividen en dos tipos: tipo A y tipo B por sus diferencias en sus secuencias de aminoácidos. Además la resistencia a cloramfenicol puede estar mediada por sistemas de exportadores específicos para cloramfenicol y florfenicol (su derivado), relacionados con genes como cmlA y floR (30); descritos en la Tabla 5.
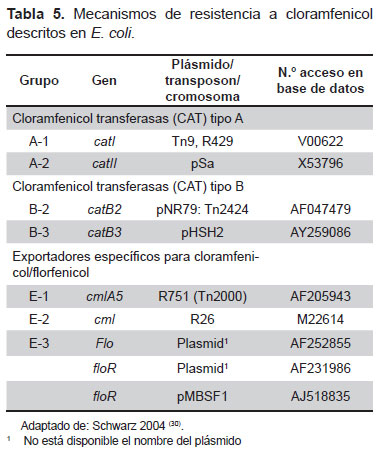
En el caso de bacterias enteropatogénicas, en un estudio realizado en Mozambique en Shigella (n= 57) y Salmonella (n= 6) se determinó que los mecanismos de resistencia a cloramfenicol se relacionaron con la presencia de CAT (89 y 67 %), cmlA (2 y 33 %) y floR (0 y 83 %) respectivamente (31). En general, el informe de los genes de resistencia a cloramfenicol relacionados con exportadores específicos cmlA y floR, es poco frecuente en relación al informe de cat. El gen floR se ha descrito en cepas de E. coli O26 provenientes de animales (32), sin embargo otros estudios donde se ha buscado este gen en STEC de origen humano y animal, no se han descrito (33). En el caso de Perú, el estudio de Mosquito et al., mostró que el gen cat es el mecanismo más frecuente en los tres grupos: cepas diarreogénicas de E coli (DEC) causantes de diarrea, DEC control y comensales. Mientras que floR (30 %) y cmlA (10 %) estuvieron mayormente presentes en el grupo comensal (17).
TRIMETROPIM – SULFAMETOXAZOL
Desde 1935, las sulfonamidas han sido extensamente usadas en medicina humana y veterinaria, lo que generó rápidamente una alta resistencia antibiótica, por ello se empezó a utilizar de manera combinada con diaminopirimidinas como es el caso del trimetropim. El mecanismo de acción de las sulfonamidas es inhibiendo la síntesis de la dihidropteroato sintasa enzima clave en la ruta del ácido fólico. Mientras que en el caso del trimetropim este actúa inhibiendo la enzima dihidrofolato reductasa (dfr), otra enzima importante en la síntesis de ácido fólico (34,35).
Los mecanismos de resistencia a sulfonamidas y a trimetropim mayormente descritos son los relacionados con la adquisición de genes mutantes mediante elementos móviles. En el caso del sulfonamidas se han descrito los genes sul1, sul2 y sul3 relacionados con integrones y que codifican formas mutantes de la enzima dihidropteroato sintasa que no pueden ser inhibidas por el antibiótico. Lo mismo sucede en el caso del trimetropim, se han descritos múltiples genes dfr que generan resistencia antibiótica (34,35,36).
En un estudio realizado en Australia, donde se aislaron E coli comensales a partir de muestras de heces de adultos sanos se determinó la presencia de los genes sul1, sul2 y sul3; así como de dfrA1, dfrA5, dfrA7, dfrA12 y dfrA17, estos genes de resistencia principalmente relacionados con integrones tipo 1 y 2 (37). En nuestro país, en un estudio con cepas de E coli aisladas de niños, las DEC-diarrea presentaron mayor resistencia a trimetropim-sulfametoxazol (SXT) (79 %) respecto a las DEC- control (65 %) y a las comensales (60 %) (p<0,05). En cuanto a mecanismos de resistencia, los genes sul1 (41 %) y dfrA1 (48 %), relacionados con trimetropim-sulfametoxazol, fueron más frecuentes en el grupo DEC-diarrea respecto a los otros grupos (p<0,05) (17).
INTEGRONES
Los integrones son piezas genéticas capaces de captar genes que codifican determinantes de resistencia antibiótica u otras funciones. Los integrones están compuestos por tres elementos necesarios para la inserción y expresión de genes exógenos: un fragmento que codifica una integrasa (intI), una secuencia attI a la que se unen los genes en casetes que codifican diferentes mecanismos de resistencia y dentro de la intI, en el extremo 3´, una secuencia promotora (Pc) a partir de la cual se transcriben los casetes de resistencia integrados (3,38), que se puede apreciar en la Figura 1.
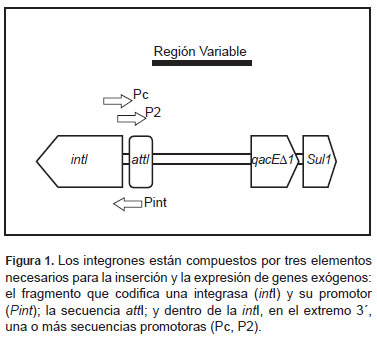
Existen dos tipos de integrones: el grupo I o también llamado “integrones móviles” relacionados con los casetes de resistencia antibiótica y el grupo II o “superintegrones” presentes, a diferencia de los primeros, a nivel cromosómico y no relacionados con la resistencia antibiótica salvo algunas excepciones. El grupo I, de nuestro interés, a su vez se divide en tres clases: integrones clase 1, clase 2 y clase 3 respectivamente, diferenciados debido a las variaciones en sus secuencias de integrasas. Entre estos tipos de integrones, el más frecuentemente descrito es el integrón I (Int1), relacionado con resistencia antibiótica en Gram negativos y que a la fecha tiene más de 100 diferentes arreglos de casetes de resistencia informados (3).
El gen de la de la integrasa (Int1) se caracteriza por tener una estructura típica constituida por: una región conservada en el extremo 5’ donde se ubican intI y attI, una sección media donde se insertan al azar los genes en casete y un extremo 3’ caracterizado por la presencia de qacED1, gen que codifica una proteína transportadora relacionada con la resistencia a sales de amonio cuaternario y por la presencia de sul1, gen anteriormente descrito relacionado con la resistencia a sulfonamidas. Sin embargo, se han descrito Int1 atípicos donde no hay sul1. En cuanto al número de genes de resistencia en la región variable, lo habitual es entre tres y cuatro, pero lo máximo encontrado han sido nueve genes de resistencia (39).
Debido a la relación directa entre la presencia de integrones y los genes de resistencia hacia diferentes familias antibióticas, durante los últimos años numerosos estudios en enterobacterias han informado su prevalencia. En el caso de E coli comensales, se describió la multidrogorresistencia en siete cepas, cinco de ellas presentaban integrón tipo 1 (37). En el Perú, la presencia de integrones clase 1 clásicos, se describió en nueve de veinte cepas SXT resistentes aisladas a partir de heces de niños sanos (34).
En el caso de E.coli diarrogénicas, un estudio realizado en Senegal donde se buscaron los tres tipos de integrones en EIEC (n=10) y EAEC (n=25), se determinó la presencia de integrón 1 en 4 EIEC y 15 EAEC, estas cepas en todos los casos menos uno fueron resistentes a cinco o más familias diferentes de antibióticos, ante esta alta resistencia antibiótica, los posibles tratamientos antibióticos se limitaban a cefalosporinas de tercera generación y a fluoroquinolonas (40). En el estudio peruano de mecanismos de resistencia en 154 cepas de E coli, se encontró que la integrasa más común fue integrasa 1 (9,1 %) respecto a integrasa 2 (6,4 %) (17).
CONCLUSIONES
Los mecanismos de resistencia antibiótica en E. coli hacia ampicilina, trimetropim-sulfametoxazol, tetraciclina, cloramfenicol y ácido nalidíxico son múltiples y son continuamente descritos. Estos mecanismos se adquieren mediante mutaciones puntuales a nivel cromosómico o transferencia horizontal de material genético entre especies relacionadas o diferentes facilitada por algunos elementos móviles tales como los integrones. Esta transferencia horizontal permite que los mecanismos se trasladen entre diferentes enteropatógenos y que se diseminen rápidamente a nivel mundial. Para cada familia de antibióticos existe más de un mecanismo de resistencia antibiótica descrito, como en el caso de la quinolonas que presentan mutaciones cromosómicas, proteínas que impiden la unión del antibiótico y las recientemente descritas bombas de eflujo específicas. En general, los tipos de mecanismos moleculares de resistencia más comunes se encuentran: inactivación enzimática, alteraciones en el sitio blanco y alteraciones de la permeabilidad.
El control molecular de la resistencia antibiótica es parte transcendental de la política de control de la vigilancia antibiótica como lo recomienda la OMS. Este mecanismo representa el futuro de la vigilancia de resistencia, debido a que si bien los mecanismos de resistencia surgen ante el uso de un antibiótico específico, generan también resistencia cruzada hacia otros antibióticos de la misma clase o con el mismo mecanismo de acción y e incluso a compuestos de familias diferentes. Por lo que son pieza clave para las políticas de control y uso de antibióticos a nivel nacional.
Contribuciones de autoría
SM, JR y TJO participaron en la concepción y diseño del trabajo, en la revisión crítica del manuscrito y en la aprobación de su versión final; además, SM realizó la redacción y TJO la obtención de financiamiento. JLB participó en la revisión crítica del manuscrito y en la aprobación de la versión final.
Conflictos de interés
Los autores declaran no tener conflictos de interés.
Fuentes de financiamiento
Este trabajo fue financiado por la Agencia Española de Cooperación Internacional (AECID), España, Proyecto: Mejora en la Infraestructura de la UPCH (Lima, Perú) para la realización de Estudios Integrales (Sociales y Moleculares) relacionados con las Enfermedades Diarreicas (D/019499/08 y D/024648/09).
REFERENCIAS BIBLIOGRÁFICAS
1. Estrategia mundial de la OMS para contener la resistencia a los antimicrobianos. Rev Panam Salud Publica. 2001;10:284-93.
2. Andersson DI, Hughes D. Antibiotic resistance and its cost: is it possible to reverse resistance?. Nat Rev Microbiol. 2010,8(4):260-71.
3. Di Conza JA, Gutkind GO. Integrons: gene collectors. Rev Argent Microbiol. 2010;42:63-78.
4. Pallecchi L, Malossi M, Mantella A, Gotuzzo E, Trigoso C, Bartoloni A, et al. Detection of CTX-M-type beta-lactamase genes in fecal Escherichia coli isolates from healthy children in Bolivia and Peru. Antimicrob Agents Chemother. 2004;48:4556-61.
5. Smith KE, Wilker PR, Reiter PL, Hedican EB, Bender JB, Hedberg CW. Antibiotic Treatment of Escherichia coli O157 Infection and the Risk of Hemolytic Uremic Syndrome, Minnesota. Pediatr Infect Dis J. 2011. [Epub ahead of print].
6. Kass B. Traveller’s diarrhoea. Aust Fam Physician. 2005;34:243-7.
7. Bartoloni A, Pallecchi L, Fiorelli C, Di Maggio T, Fernandez C, Villagran AL, et al. Increasing resistance in commensal Escherichia coli, Bolivia and Peru. Emerg Infect Dis. 2008;14:338-40.
8. Tenover FC. Mechanisms of antimicrobial resistance in bacteria. Am J Med. 2006;119:S3-10; discussion S62-70.
9. Bush K, Jacoby GA. Updated functional classification of beta-lactamases. Antimicrob Agents Chemother. 2009;54(3):969-76.
10. Lahey Clinic. ß-Lactamase Classification and Amino Acid Sequences for TEM, SHV and OXA Extended-Spectrum and Inhibitor Resistant Enzymes. Burlington, MA: Lahey Clinic Foundation; 2011.
11. Pallecchi L, Bartoloni A, Fiorelli C, Mantella A, Di Maggio T, Gamboa H, et al. Rapid dissemination and diversity of CTX-M extended-spectrum beta-lactamase genes in commensal Escherichia coli isolates from healthy children from low-resource settings in Latin America. Antimicrob Agents Chemother. 2007;51:2720-5.
12. Réjiba S, Mercuri PS, Power P, Kechrid A. Emergence and dominance of CTX-M-15 extended spectrum beta-lactamase among Escherichia coli isolates from children. Microb Drug Resist. 2011;17(2):135-40.
13. Poirel L, Carattoli A, Bernabeu S, Bruderer T, Frei R, Nordmann P. A novel IncQ plasmid type harbouring a class 3 integron from Escherichia coli. J Antimicrob Chemother. 2010;65(8):1594-8.
14. Lee SH, Jeong SH. Nomenclature of GES-type extended-spectrum beta-lactamases. Antimicrob Agents Chemother. 2005;49(5):2148.
15. Karami N, Hannoun C, Adlerberth I, Wold AE. Colonization dynamics of ampicillin-resistant Escherichia coli in the infantile colonic microbiota. J Antimicrob Chemother. 2008;62:703-8.
16. Vignoli R, Varela G, Mota MI, Cordeiro NF, Power P, Ingold E, et al. Enteropathogenic Escherichia coli strains carrying genes encoding the PER-2 and TEM-116 extended-spectrum beta-lactamases isolated from children with diarrhea in Uruguay. J Clin Microbiol. 2005;43:2940-3.
17. Mosquito SG, Pons MJ, Maves R, Saenz Y, Mercado E, Vargas M, et al. Prevalence of Extended Spectrum Beta-Lactamases in Escherichia coli strains isolated from Peruvian infants. 20th European Congress of Clinical Microbiology and Infectious Diseases (ECCMID). Vienna, Austria. April, 10-13, 2010.
18. Ruiz J. Mechanisms of resistance to quinolones: target alterations, decreased accumulation and DNA gyrase protection. J Antimicrob Chemother. 2003;51:1109-17.
19. Lautenbach E, Metlay JP, Mao X, Han X, Fishman NO, Bilker WB. The prevalence of fluoroquinolone resistance mechanisms in colonizing Escherichia coli isolates recovered from hospitalized patients. Clin Infect Dis. 2010;51:2805.
20. Jacoby G, Cattoir V, Hooper D, Martínez-Martínez L , Nordmann P, Pascual A, et al. Qnr gene nomenclature. Antimicrob Agents Chemother. 2008;52:2297-9.
21. Lahey Clinic. qnr Numbering and sequencing. Burlington, MA: Lahey Clinic Foundation; 2011.
22. Cattoir V, Nordmann P. Plasmid-mediated quinolone resistance in gram-negative bacterial species: an update. Curr Med Chem. 2009;16:1028-46.
23. Cordeiro NF, Robino L, Medina J, Seija V, Bado I, Garcia V, et al. Ciprofloxacin-resistant enterobacteria harboring the aac (6’)-Ib-cr variant isolated from feces of inpatients in an intensive care unit in Uruguay. Antimicrob Agents Chemother. 2008;52:806-7.
24. Saenz Y, Ruiz J, Zarazaga M, Teixidó M, Torres C, Vila J. Effect of the efflux pump inhibitor Phe-Arg-β-naphthylamide on the MIC values of the quinolones, tetracycline and chloramphenicol, in Escherichia coli isolates of different origin. J Antimicrob Chemother. 2004;53:544-5.
25. Yamane K, Wachino J, Suzuki S, Kimura K, Shibata N, Kato H, et al. New plasmid-mediated fluoroquinolone efflux pump, QepA, found in an Escherichia coli clinical isolate. Antimicrob Agents Chemother. 2007;51(9):3354-60.
26. Silva-Sanchez J, Barrios H, Reyna-Flores F, Bello- Diaz M, Sanchez-Perez A, Rojas T, et al. Prevalence and Characterization of Plasmid-Mediated Quinolone Resistance Genes in Extended-Spectrum β-Lactamase- Producing Enterobacteriaceae Isolates in Mexico. Microb Drug Resist. 2011. [Epub ahead of print].
27. Chopra I, Roberts M. Tetracycline antibiotics: mode of action, applications, molecular biology, and epidemiology of bacterial resistance. Microbiol Mol Biol Rev. 2001;65:232-60.
28. Karami N, Nowrouzian F, Adlerberth I, Wold AE. Tetracycline resistance in Escherichia coli and persistence in the infantile colonic microbiota. Antimicrob Agents Chemother. 2006;50:156-61.
29. Levy SB, McMurry LM, Barbosa TM, Burdett V, Courvalin P, Hillen W, et al. Nomenclature for new tetracycline resistance determinants. Antimicrob Agents Chemother. 1999;43:1523-4.
30. Schwarz S, Kehrenberg C, Doublet B, Cloeckaert A. Molecular basis of bacterial resistance to chloramphenicol and florfenicol. FEMS Microbiol Rev. 2004;28:519-42.
31. Mandomando I, Jaintilal D, Pons MJ, Vallés X, Espasa M, Mensa L, et al. Antimicrobial susceptibility and mechanisms of resistance in Shigella and Salmonella isolates from children under five years of age with diarrhea in rural Mozambique. Antimicrob Agents Chemother. 2009;53:2450-4.
32. Srinivasan V, Gillespie BE, Nguyen LT, Headrick SI, Murinda SE, Oliver SP. Characterization of antimicrobial resistance patterns and class 1 integrons in Escherichia coli O26 isolated from humans and animals. Int J Antimicrob Agents. 2007;29:254-62.
33. Srinivasan V, Nguyen LT, Headrick SI, Murinda SE, Oliver SP. Antimicrobial resistance patterns of Shiga toxin-producing Escherichia coli O157:H7 and O157:H7- from different origins. Microb Drug Resist. 2007;13:44-51.
34. Ho PL, Wong RC, Chow KH, Que TL. Distribution of integron-associated trimethoprim-sulfamethoxazole resistance determinants among Escherichia coli from humans and food-producing animals. Lett Appl Microbiol. 2009;49:627-34.
35. Infante B, Grape M, Larsson M, Kristiansson C, Pallecchi L, Rossolini GM, et al. Acquired sulphonamide resistance genes in faecal Escherichia coli from healthy children in Bolivia and Peru. Int J Antimicrob Agents. 2005;25:308-12.
36. Dahmen S, Mansour W, Boujaafar N, Arlet G, Bouallegue O. Distribution of cotrimoxazole resistance genes associated with class 1 integrons in clinical isolates of Enterobacteriaceae in a university hospital in Tunisia. Microb Drug Resist. 2010;16:43-7.
37. Bailey JK, Pinyon JL, Anantham S, Hall RM. Commensal Escherichia coli of healthy humans: a reservoir for antibiotic-resistance determinants. J Med Microbiol. 2010;59:1331-9.
38. Sabate M, Guillem P. Estructura y función de los integrones. Enfer Infecc Microbiol Clin. 2002;20:341-5.
39. Naas T, Mikami Y, Imai T, Poirel L, Nordmann P. Characterization of In53, a class 1 plasmid- and composite transposon-located integron of Escherichia coli which carries an unusual array of gene cassettes. J Bacteriol. 2001;183(1):235-49.
40. Gassama A, Aidara-Kane A, Chainier D, Denis F, Ploy MC. Integron-associated antibiotic resistance in enteroaggregative and enteroinvasive Escherichia coli. Microb Drug Resist. 2004;10:27-30.
Correspondencia:
Theresa Jean Ochoa
Dirección: Av. Honorio Delgado 430, San Martín de Porras, Lima 33, Perú
Teléfono: (511) 482 3903
Correo electrónico: theresa.J.ochoa@uth.tmc.edu; theresa. ochoa@upch.pe
Recibido: 13-07-11
Aprobado: 09-11-11