Marco Galarza
Silvia Capristano
Oscar Pellón
César Sánchez
Jorge Ballon
Heinner Guio
ARTÍCULO ORIGINAL
Expresión diferencial de micro-ARN circulantes
en pacientes con tuberculosis activa y latente
Differential expression of circulating
micro-RNAs in patients with active tuberculosis and infected
with latent tuberculosis
José Yareta ![]() 1,2, Biólogo
1,2, Biólogo
Marco Galarza ![]() 1, Biólogo
1, Biólogo
Silvia Capristano
![]() 1, tecnólogo médico con especialidad en Laboratorio
Clínico
1, tecnólogo médico con especialidad en Laboratorio
Clínico
Oscar Pellón ![]() 1, biólogo molecular, PhD en Biología Celular y
Molecular del Cáncer
1, biólogo molecular, PhD en Biología Celular y
Molecular del Cáncer
César Sánchez ![]() 1, médico cirujano; Maestría en Ciencias
1, médico cirujano; Maestría en Ciencias
Jorge Ballon
![]() 2, médico cirujano; Maestría en Ciencias
2, médico cirujano; Maestría en Ciencias
Heinner Guio ![]() 1, médico cirujano; PhD en Ciencias Médicas
1, médico cirujano; PhD en Ciencias Médicas
1 Laboratorio
de Referencia Nacional de Biotecnología y Biología Molecular, Instituto
Nacional de Salud, Lima, Perú.
2 Departamento
Académico de Biología, Universidad Nacional de San Agustín de Arequipa,
Arequipa, Perú.
RESUMEN
Objetivo: El objetivo del estudio fue analizar la expresión diferencial
de miR-21, miR-29a, miR-99b y miR-155 en muestras de suero de pacientes con
tuberculosis (TB) latente y TB activa respecto a controles sanos.
Materiales y métodos: Se utilizaron 28 muestras de suero (nueve con TB
activa, diez con TB latente y nueve controles sanos) para el análisis de
expresión génica mediante RT-qPCR con Primers y sondas TaqMan.
Se calculó la expresión diferencial por el método de Livak
utilizando un gen normalizador (RNU-48).
Resultados: Se halló una sobreexpresión de miR-155 en personas con
tuberculosis latente, respecto a los controles sanos (0,63 vs. 0,01; valor de
p=0,032).
Conclusión: El miR-155 podría ser considerado un biomarcador
para diferenciar TB latente de enfermedad activa. Se requieren estudios con
mayores tamaños muestrales para corroborar nuestros
hallazgos.
Palabras clave: Tuberculosis Latente; Tuberculosis; MicroARNs; Perú (fuente: DeCS BIREME).
ABSTRACT
Objectives: To analyze the
differential expression of
miR-21, miR-29a, miR-99b and miR-155 in serum samples from patients
with latent tuberculosis
(TB) and active TB compared to healthy
controls.
Materials and Methods: We used
28 serum samples (9 with active TB, 10 with latent TB and 9 healthy controls) for the analysis of gene expression by RT-qPCR with Primers
and TaqMan probes. The differential expression was calculated by the
Livak method using a normalizing gene
(RNU-48).
Results: Overexpression of miR-155 was found in people
with latent tuberculosis, compared to healthy controls (0.63 vs. 0.01; p value
= 0.032).
Conclusion: The miR-155 could
be considered a biomarker
to differentiate latent TB from active disease. Studies with larger
sample sizes are required to corroborate the findings.
Keywords: Latent Tuberculosis; Tuberculosis; microRNAs; Peru (source: MeSH NLM).
INTRODUCCIÓN
La tuberculosis (TB) es una de
las diez enfermedades con más muertes en el mundo y es causada por el bacilo Mycobacterium tuberculosis (Mtb). En 2017, la Organización Mundial de la Salud (OMS)
reportó que 31 120 casos de TB fueron notificados en Perú (1, 2).
Asimismo, 1,7 mil millones de personas presentan TB latente, además según Houben y Dodd del 20 % a 30 % de
la población peruana tiene TB latente (3).
La búsqueda de nuevos biomarcadores, derivados del patógeno o del hospedero, se
está enfocando en conceptos de comunicación celular, representadas por las
vesículas extracelulares llamadas exosomas. El
estudio de micro-ARN circulantes contenidos en estos exosomas
está proponiendo nuevos métodos de diagnóstico en distintas enfermedades,
incluyendo enfermedades infecciosas (4), por lo que pueden ser
detectadas fácilmente en los distintos tipos de biofluídos
del cuerpo humano.
|
MENSAJES CLAVE Motivación para realizar el estudio: La búsqueda de un biomarcador
que permita diagnosticar tuberculosis latente de manera rápida y confiable en
el Perú. Principales hallazgos: Se encontró un micro-ARN (miR-155) que estaría
sobre expresado en individuos con diagnóstico de tuberculosis latente. Implicancias: Al tener un alto porcentaje de población peruana infectada con
tuberculosis latente, un biomarcador adecuado nos
ayudará a diagnosticar de manera oportuna la infección tuberculosa antes que
se desarrolle la enfermedad activa y dirigir el tratamiento o profilaxis para
quienes realmente lo necesitan. |
El análisis de los cambios dinámicos del cargo de exosomas, a nivel de micro-ARN y proteínas en específico,
nos ofrece la posibilidad de investigar la activación de vías de señalización celular e inmunológicas en tiempo real, para
validar biomarcadores que correspondan a estadios
sintomáticos y asintomáticos (5).
En la actualidad los métodos
de diagnóstico de tuberculosis solo detectan la enfermedad activa, por lo que
no existen biomarcadores que diferencien TB latente
y TB activa para un diagnóstico más específico, es por ello que desde hace
algunos años se viene investigando la diferencia de moléculas producidas por el
hospedero (micro-ARN) en el contexto de una enfermedad infecciosa versus
personas sanas como control, este proceso se denomina «expresión diferencial» y
es evaluado por PCR en tiempo real, considerando un gen normalizador o gen de
referencia (6,
7). Estas moléculas son micro-ARN con 18 a 24 nucleótidos, no
codificantes, endógenamente expresados y altamente conservados, que regulan la
expresión postranscripcional de los genes mediante la
sub/sobre expresión de ARNs en una amplia gama de
organismos tanto en contextos fisiológicos normales como en contextos de
enfermedades.
La desregulación de micro-ARN
es variable en las enfermedades infecciosas y según población estudiada. En
este sentido, el estudio de estos micro-ARN en TB ha sido evaluado en población
europea y asiática principalmente, donde se caracterizaron diversos micro-ARN
expresados diferencialmente entre las patologías (8, 9). Los avances
tecnológicos han generado una multitud de plataformas para la creación de
perfiles de micro-ARN y una comprensión de las fortalezas y las dificultades de
los diferentes enfoques puede ayudar a su uso efectivo (10).
En el presente estudio, se
evaluó la expresión diferencial de los micro-ARN miR-21, miR-29a, miR-99b y
miR-155 a partir de muestras de suero de pacientes, con la finalidad de poder
encontrar un perfil diferente en TB latente en comparación con TB activa con
respecto a controles sanos.
MATERIALES Y MÉTODOS
Muestras clínicas y extracción
de ARN
Las muestras clínicas fueron
constituidas por 28 muestras de suero de pacientes con diagnóstico TB activa
(n=9), personas con TB latente (n=10) y controles sanos (n=9) (Tabla 1). Las
muestras estuvieron almacenadas en crioviales de 2 ml
en la congeladora a–80°C (Thermo Fisher Scientific Inc.) de la seroteca
del área de Inmunología del Laboratorio de Referencia Nacional de Biotecnología
y Biología Molecular del Centro Nacional de Salud Pública del Instituto
Nacional de Salud.
Tabla
1.
Características clínicas y de exámenes auxiliares en pacientes con TB latente,
TB activa y controles sanos.
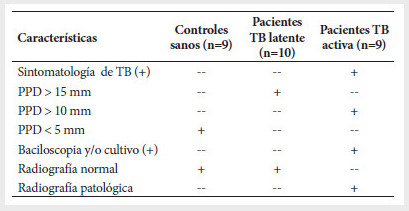
PPD: Derivado Proteico Purificado de
Mycobacterium tuberculosis; TB: Tuberculosis; mm:
milímetros; (+) resultado positivo de la prueba.
Las muestras fueron
recolectadas durante el periodo comprendido entre 2012 y 2016, y forman parte
de un estudio aprobado por el Comité de Ética en Investigación del Instituto
Nacional de Salud, «Estudio de las características inmunológicas de la
tuberculosis latente en la población peruana, año 2012-2014» (según código
OGIIT OI 01-072-10). Cada participante del estudio firmó un consentimiento
informado previamente aprobado por el Comité de Ética del Instituto Nacional de
Salud del Perú.
La extracción de ARN a partir
de los sueros seleccionados se realizó según las recomendaciones del fabricante
con el uso del kit miRNeasy (Qiagen). La concentración de ARN total fue cuantificado
usando un equipo Nanodrop 8000 (Thermo
Fisher). El ARN extraído se almacenó a -20 °C en agua ultra pura libre de ARNasas (11) y
procesado inmediatamente por retrotranscripción a
ADN complementario dentro de las 72 horas.
Selección de micro-ARN para validación por PCR en tiempo real
Se realizó una búsqueda de los micro-ARN expresados
diferencialmente en publicaciones previas que incluían diferentes poblaciones,
encontrándose principalmente trabajos en población europea y asiática (8, 9, 12-14).
Los criterios de selección de micro-ARN incluyeron: a) micro-ARN expresados en
pacientes con TB, b) micro-ARN diferencialmente expresados en suero por PCR en
tiempo real y c) micro-ARN sobreexpresados en
pacientes con TB mediante las plataformas Microarray
y Next generation
sequencing. Teniendo en cuenta estos criterios,
de un grupo de micro-ARN relacionados con TB se seleccionaron cuatro micro-ARN:
miR-21, miR-29a, miR-99b y miR-155 como posibles biomarcadores
para un diagnóstico diferencial entre TB latente y TB activa (9, 10, 13-16) en muestras de la población peruana.
PCR en tiempo real y análisis
de expresión diferencial de micro-ARN
Los primers
ya diseñado fueron los siguientes: hsa-miR-21 (MI0000077:UAGCUUAUCAGACUGAUGUUGA),
hsa-miR-29a (MI0000087: UAGCACCAUCUGAAAUCGGUUA), hsa-miR-99b (MI0000746:
UGAGGUAGGAGGUUGUAUAGUU) y hsamiR-155 (MI0000681: UUAAUGCUAAUCGUGAUAGGGGU) (Applied Biosystem) de la base de
datos miRbase (http://www.mirbase.org/)
(17).
La amplificación de los micro-ARN se realizó en dos pasos: inicialmente se hizo
una retrotranscripción para obtener el ADN
complementario, usando el kit TaqMan MicroRNA Reverse Transcription
(Applied Biosystem),
seguido de la amplificación por PCR en tiempo real usando el kit TaqMan Universal PCR Master Mix
II (Applied Biosystem).
Los volúmenes y concentraciones de muestras fueron según recomendaciones del
fabricante (18).
La retrotranscripción se realizó en el termociclador Veriti (Applied Biosystem) bajo las
siguientes condiciones: 16 °C por 30 minutos, 42 °C por 30 minutos y 85 °C por
5 minutos. La posterior amplificación se realizó en el termociclador
Rotor Gene Q (Qiagen) siguiendo las condiciones: 95
°C por 10 minutos, 45 ciclos de 95°C por 15 segundos y 60°C por 60 segundos.
Cada muestra se corrió por duplicado y
el termociclador nos consideró un Ct
promedio de ambas corridas por muestra y gen de interés evaluado. Se utilizó el
ARN nuclear RNU48 como gen de referencia (11, 19) para normalizar la expresión de los genes en
estudio.
El análisis de expresión
diferencial se calculó mediante el método de Livak,
que utiliza los valores de Ct (valor umbral para
discriminar entre negativo y positivo, máximo Ct
considerado fue de 35) (19) de las muestras enfermas y muestras sanas para
finalmente normalizarse con el gen de referencia (RNU48). La mediana de los
valores de Ct se usó para calcular la cantidad
relativa mediante el método comparativo ddCt. Los
valores de ddCt se utilizaron para hallar el nivel de
expresión diferencial o cuantificación relativa normalizada mediante el método
de Livak o 2 (-ddCt),
donde dCt muestras enfermas = (Ct micro-ARN de interés - Ct
RNU48), dCt muestras sanas = (Ct micro-ARN de interés – Ct
RNU48) y el ddCt = (dCt
muestras enfermas - dCt muestras
sanas) y la cuantificación relativa normalizada o expresión diferencial
es igual a 2-ddCt o Fold Change (FC, que es el cambio
en la expresión respecto al grupo control o muestras sanas) (7).
Análisis estadístico
Los niveles de expresión de micro-ARN se analizaron
mediante la prueba no paramétrica Kruskal-Wallis con
la cual se hicieron múltiples comparaciones entre los grupos TB latente, TB
activo y controles sanos y luego se realizó la prueba de Mann-Whitney. Se utilizó
el paquete estadístico Prism 6 (GraphPad
Software, Inc, CA, USA).
RESULTADOS
Cuantificación relativa de
micro-ARN por PCR en tiempo real
Los resultados de
cuantificación relativa por PCR en tiempo real muestran valores de Ct diferentes en cada curva de fluorescencia para miR-21,
miR-29a, miR-99b y miR-155 respecto al normalizador RNU48, los cuales nos
dieron valores de Ct menores a 35, estos Ct se usaron para hallar el ddCt
y el nivel de expresión relativa normalizado.
Al analizar los datos de Ct obtenidos por PCR en tiempo real, se encontró que los
cuatro micro-ARN seleccionados estuvieron expresados diferencialmente (pero
sin llegar a una diferencia estadísticamente significativa al comparar los
grupos mediante la prueba no paramétrica Kruskal-Wallis
y Mann-Whitney en los pacientes con TB activa y pacientes
con TB latente en comparación con los controles sanos).
Normalización y análisis de
datos
La cuantificación relativa
normalizada o expresión génica para miR-21, miR-29a, miR-99b y miR-155 en
muestras de suero, según el método de Livak, está
representado por la cantidad de veces más expresado en muestras de TB activa y
TB latente frente al grupo control. La cuantificación relativa normalizada (RQn) de estos micro-ARN entre TB latente y TB activa está
representada por la mediana relativa entre ambas patologías (Tabla 2).
Tabla
2.
Análisis de expresión diferencial normalizada de miR-21, miR-29a, miR-99b y
miR-155 entre pacientes con TB latente y pacientes con TB activa frente a
controles sanos.
L1-L10: Pacientes con diagnóstico
clínico de tuberculosis latente; A1- A9: Pacientes con diagnóstico clínico de
tuberculosis activa; ddCt*:
método de Livak o expresión (fold change); TB: Tuberculosis.
No se encontró diferencias
estadísticas en la expresión del gen miR-99b entre los pacientes con TB activa
y TB latente (Figuras 1 y 2).
Se muestra la distribución de las
expresiones desde el mínimo al máximo nivel de expresión en cada grupo y las
diferencias en las medianas para cada grupo de genes.

Figura
1.
Cuantificación relativa normalizada en diez pacientes con TB latente y nueve
pacientes con TB activa frente a los controles sanos. Análisis realizado por el
método de Livak.
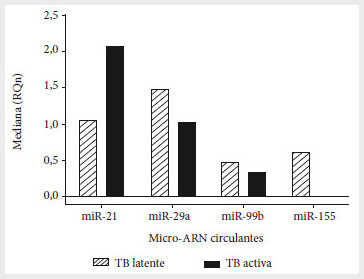
Figura
2.
Expresión diferencial (medianas) de micro-ARN entre TB latente y TB activa.
DISCUSIÓN
Desde hace algunos años, los
análisis transcriptómicos están brindando información
acerca de cómo se están expresando los genes y como estos son regulados por ARN
pequeños, dentro de este grupo los micro-ARN. Actualmente, estas pequeñas moléculas
están siendo utilizadas como potenciales componentes de vacunas, biomarcadores de diagnóstico temprano en cáncer y
predictores de enfermedades metabólicas (20, 21). Asimismo, la expresión
de otros grupos de ARNs pequeños, de los cuales no se
conocía muy claramente su función, hoy en día están
siendo considerados como biomarcadores de enfermedad (22).
Los hallazgos relacionados a
la desregulación de estos micro-ARN son variados. Wang et al. demostró
que miR-21 se expresa de modo diferencial entre pacientes con TB activa e
pacientes con TB latente, mostrando así que se pudiera utilizar este mi-cro-ARN como biomarcador para
diferenciar a las personas con TB latente de TB activa (2,99 FC) (23). En
nuestra investigación se encontró una sobreexpresión de miR-21 en muestras de
TB activa (2.07 FC), lo que es congruente con el trabajo de Wu
et al. quienes señalaron que miR-21 podría
estar involucrado en la regulación de la respuesta antimicobacterial.
Ellos reportan que miR-21 podría ser inducido después de la vacunación con BCG
por la activación de NF-kB, y que miR-21 podría suprimir la producción de IL-12
(24),
y que podría indicar que en pacientes con TB latente miR-21 esté también sobre
expresado; tal como lo reportó Kleinsteuber et al.
en una investigación donde compararon la expresión de miR-21 en donantes sanos
(PPD negativo), individuos con TB latente y pacientes con TB activa;
encontrando mayor expresión de miR-21 en pacientes con TB latente (0,0438 FC)
que en presencia de PPD (0,0393 FC) y pacientes con TB activa (0,0121 FC) (9). Sin
embargo, según nuestros resultados el perfil de expresión de miR-21 en muestras
de TB latente es mayor respecto al control, pero en menor expresión en
presencia de TB activa; por lo que miR-21 podría ser una efectiva estrategia
que usa Mtb frente a la respuesta inmune del hospedero
para evadir a las células de defensa y desarrollar la TB activa (14),
pero al comparar la expresión entre TB latente y TB activa, no hay diferencias
significativas, por lo que no sería un biomarcador
para diferenciar ambas patologías.
Por otra parte, Wu et al. reportaron que el
perfil de expresión de miR-155 (3,7 FC) en presencia de PPD (proteína derivada
de Mtb para detectar TB latente por una respuesta
inmune antígeno-anticuerpo) podría ser un potencial marcador para el
diagnóstico de TB en presencia de antígenos específicos a Mtb
(25).
Sin embargo, nuestros resultados muestran mayor expresión de miR-155 en
tuberculosis latente (0,63 FC) que en TB activa (0,01 FC) respecto a las
muestras control, miR-155 podría ser un posible biomarcador
de TB latente. Este dato no es totalmente opuesto al de J. Wu
et al., ya que ellos también encontraron una sobreexpresión de miR-155
en presencia de PPD. Esta proteína de Mtb activa al
sistema inmune temprano, expresando efectores inmunológicos contra la TB
latente tal como lo demuestra Fu et al. en un
estudio reciente donde expresa que miR-155 regula la respuesta inmune
adaptativa (13).
El sistema inmunológico al
regular la invasión de Mtb no causa sintomatología
clínica, por lo tanto la infección se vuelve latente en el hospedero, notándose
así una sobreexpresión de miR-155 en muestras de suero tal como lo predijeron Kumar et al., donde miR155 es sobreexpresado
en macrófagos infectados con Mtb como una respuesta
inmune temprana contra la infección de tuberculosis (26); lo
que ratificaría nuestros resultados de una sobreexpresión de miR-155 en
muestras de suero con TB latente (0,63 FC) al compararlos con TB activa (0,01
FC). Asimismo, Wang et al. reportaron expresión
diferencial de miR-155 entre TB activa y TB latente, ya que miR-155 está subexpresado en muestras de TB activa (0,51 FC) y sobreexpresado en muestras de TB latente (1,97 FC) (23).
Por su parte, lado Zhou et al. describen que miR-155 está subexpresado
en muestras con TB activa de niños (27); lo que permitiría usar
miR-155 como biomarcador de TB latente en las
diferentes etapas de la vida. En otro estudio, Wagh et
al. resaltó la subexpresión
de miR-155 en muestras de TB-MDR, respecto al control y pacientes con TB en
tratamiento, lo cual demuestra que la virulencia de Mtb
se puede diagnosticar por la subexpresión de miR-155 (28).
Por lo que los resultados de nuestra investigación sugieren que miR-155 podría
ser un efectivo biomarcador para el diagnóstico de TB
latente y marcar la progresión de la enfermedad en sus diferentes estadios.
El nivel de expresión de
miR-29a hallado en nuestro estudio es elevado en muestras de TB activa y TB
latente respecto al control, pero fue más sobreexpresado
en muestras de TB latente que en las de TB activa. Este resultado coincide con
los estudios de Fu et al., quienes hallaron sobreexpresión de miR-29a
(11,9 FC), que podría discriminar a pacientes con TB de controles sanos (13, 29).
Pero en nuestro estudio además rescatamos una sobreexpresión de miR-29a (1,50
FC) en TB latente respecto a la expresión en muestras de TB activa (1,05 FC),
pero sin encontrar diferencias significativas en la expresión entre TB latente
y TB activa.
Se debe mencionar que una de
las limitaciones para el presente estudio fue el número total de participantes,
esto debido a que se planteó como un estudio exploratorio, por lo que los
resultados obtenidos deben validarse con un número mayor de participantes.
Por otro lado, actualmente se
vienen realizando estudios a nivel de proteínas exosomales
que podrán ser usados en diagnóstico de rutina de algunos cánceres (30) y consideramos que el siguiente paso será con
los micro-ARN para diagnóstico de enfermedades infecciosas.
En conclusión, el análisis de
los niveles de expresión diferencial de micro-ARN en suero de pacientes con TB
activa y TB latente es interesante para el diagnóstico de esta patología.
Basándonos en los resultados de este estudio, podemos concluir que la
sobreexpresión significativa de miR-155 en muestras con TB latente podría ser
un posible biomarcador para la diferenciación entre
tuberculosis latente y activa. Sin embargo, se requieren estudios con mayor
número de participantes para corroborar nuestros hallazgos.
REFERENCIAS BIBLIOGRÁFICAS
1. GBD Tuberculosis Collaborators. Global, regional, and national
burden of tuberculosis, 1990-2016: results from the
Global Burden of Diseases,
Injuries, and Risk Factors
2016 Study. Lancet Infect Dis. 2018;18(12):1329-1349. doi:10.1016/S1473-3099(18)30625-X
2. World
Health Organization. Global
tuberculosis report 2018 [Internet]. Geneva: WHO;
2018 [citado el 14 de septiembre de 2019]. Disponible en: https://www.who.int/tb/publications/global_report/gtbr2018_main_text_28Feb2019.pdf?ua=1.
3. Houben RM, Dodd PJ. The Global Burden of Latent Tuberculosis Infection: A Re-estimation Using Mathematical Modelling. PLoS Med. 2016;13(10):e1002152. doi: 10.1371/journal.pmed.1002152.
4. Zhang W, Jiang X, Bao
J, Wang Y, Liu H, Tang L. Exosomes in pathogen infections: a bridge to deliver molecules and link functions.
Front Immunol. 2018;9:90. doi: 10.3389/fimmu.2018.00090.
5. Smith VL, Cheng
Y, Bryant BR, Schorey JS. Exosomes
function in antigen presentation during an in vivo Mycobacterium
tuberculosis infection. Sci
Rep. 2017;7:43578. doi: 10.1038/srep43578.
6. Chen C, Ridzon DA, Broomer AJ, Zhou Z, Lee DH, Nguyen JT, et al.
Real-time quantification of micro-RNAs
by stem-loop RT-PCR. Nucleic Acids Res. 2005;33(20):e179. doi:
10.1093/nar/gni178.
7. Schmittgen
TD, Livak KJ. Analyzing
real-time PCR data by the comparative C(T) method. Nat Protoc.
2008;3(6):1101-8. doi: 10.1038/nprot.2008.73.
8. Correia
CN, Nalpas NC, McLoughlin
KE, Browne JA, Gordon SV, MacHugh
DE, et al. Circulating microRNAs
as Potential Biomarkers of Infectious Disease. Front Immunol. 2017;8:118. doi: 10.3389/fimmu.2017.00118.
9. Kleinsteuber
K, Heesch K, Schattling S, Kohns M, Sander-Julch C, Walzl G, et al. Decreased expression of miR-21, miR-26a, miR-29a, and miR-142-3p in CD4(+) T cells and peripheral blood from tuberculosis patients. PloS One. 2013;8(4):e61609.
doi:
10.1371/journal.pone.0061609.
10. Pritchard
CC, Cheng HH, Tewari M. MicroRNA profiling: approaches and considerations. Nat Rev Genet.
2012;13(5):358-69. doi: 10.1038/nrg3198.
11. Yareta Yareta
JL. Validación de un perfil de microARNs para el
diagnóstico diferencial de tuberculosis latente y tuberculosis activa 2018
[Tesis]. Arequipa: Universidad Nacional San Agustín de Arequipa; 2018.
Disponible en: http://repositorio.unsa.edu.pe/bitstream/handle/UNSA/4795/BIyayajl.pdf?sequence=1&isAllowed=y.
12. Abd-El-Fattah AA, Sadik NA, Shaker OG, Aboulftouh ML. Differential microRNAs expression in serum of patients with lung
cancer, pulmonary
tuberculosis, and pneumonia. Cell
Biochem Biophys. 2013;67(3):875-84. doi:
10.1007/s12013-013-9575-y.
13. Fu Y, Yi
Z, Wu X, Li J, Xu F. Circulating microRNAs in patients with active pulmonary tuberculosis. J Clin Microbiol. 2011;49(12):4246-51. doi: 10.1128/ JCM.05459-11.
14. Harapan
H, Fitra F, Ichsan I, Mulyadi M, Miotto P, Hasan NA, et
al. The roles of microRNAs
on tuberculosis infection: meaning or myth?. Tuberculosis (Edinb). 2013;93(6):596-605. doi:
10.1016/j.tube.2013.08.004.
15. Huang
J, Jiao J, Xu W, Zhao H, Zhang C, Shi Y, et al.
MiR-155 is upregulated in patients with active tuberculosis
and inhibits apoptosis of monocytes
by targeting FOXO3. Mol Med Rep. 2015;12(5):7102-8. doi: 10.3892/mmr.2015.4250.
16. Mashima
R. Physiological roles of miR-155. Immunology. 2015;145(3):323-33. doi: 10.1111/imm.12468.
17. Kozomara
A, Griffiths-Jones S. miRBase:
annotating high confidence microRNAs using deep sequencing
data. Nucleic Acids Res.
2014;42(Database issue):D68-73. doi:
10.1093/nar/gkt1181.
18. Biosystems
A. TaqMan® Universal Master Mix
II. Protocol: Life
Technologies Corporation [Internet] thermoFisher scientific; 2010
[citado el 20 de septiembre del 2019]. Disponible en: https://assets.thermofisher.com/TFSAssets/LSG/manuals/cms_069368.pdf.
19. Miotto
P, Mwangoka G, Valente IC, Norbis
L, Sotgiu G, Bosu R, et
al. miRNA signatures in sera of patients with active pulmonary tuberculosis. PloS One. 2013;8(11):e80149. doi: 10.1371/journal.pone.0080149.
20. Chakraborty
C, George Priya Doss C, Bandyopadhyay S. miRNAs in insulin resistance and diabetes-associated pancreatic cancer: the ‘minute and miracle’ molecule moving as a monitor in the ‘genomic galaxy’. Curr Drug Targets. 2013;14(10):1110-7. doi:
10.2174/13894501113149990182.
21. Munagala
R, Aqil F, Gupta RC. Exosomal miRNAs as biomarkers of recurrent lung cancer. Tumour
biol. 2016;37(8):10703-14. doi: 10.1007/s13277-016-4939-8.
22. Qian
Z, Liu H, Li M, Shi J, Li
N, Zhang Y, et al. Potential Diagnostic Power of Blood Circular RNA Expression in
Active Pulmonary Tuberculosis. EBioMedicine.
2018;27:18-26. Doi: 10.1016/j.ebiom.2017.12.007.
23. Wang C, Yang S, Sun G, Tang X, Lu S, Neyrolles O, et al. Comparative
miRNA expression profiles in individuals with latent and active
tuberculosis. PloS One.
2011;6(10):e25832. Doi: 10.1371/journal.pone.0025832.
24. Wu
Z, Lu H, Sheng J, Li L. Inductive
microRNA-21 impairs anti-mycobacterial
responses by targeting
IL-12 and Bcl-2. FEBS letters. 2012;586(16):2459-67.
doi:
10.1016/j.febslet.2012.06.004.
25. Wu
J, Lu C, Diao N, Zhang S, Wang S, Wang F, et al.
Analysis of microRNA expression profiling identifies miR-155 and miR-155* as potential
diagnostic markers for active tuberculosis: a preliminary
study. Hum immunol. 2012;73(1):31-7. doi: 10.1016/j.humimm.2011.10.003.
26. Kumar
R, Halder P, Sahu SK, Kumar M, Kumari M, Jana K, et
al. Identification of a novel role of
ESAT-6-dependent miR-155 induction during infection of macrophages with Mycobacterium tuberculosis. Cell Microbiol. 2012;14(10):1620-31. doi:
10.1111/j.1462-5822.2012.01827.x.
27. Zhou
M, Yu G, Yang X, Zhu C,
Zhang Z, Zhan X. Circulating
microRNAs as biomarkers for the early
diagnosis of childhood tuberculosis infection. Mol Med Rep. 2016;13(6):4620-6. doi:
10.3892/mmr.2016.5097.
28. Wagh
V, Urhekar A, Modi D. Levels of microRNA miR-16 and
miR-155 are altered in serum
of patients with
tuberculosis and associate with
responses to therapy. Tuberculosis (Edinb). 2017;102:24-30. doi: 10.1016/j.tube.2016.10.007.
29. Fu Y, Yi
Z, Li J, Li R. Deregulated microRNAs
in CD4+ T cells from individuals with latent tuberculosis versus active tuberculosis. J Cell Mol Med. 2014;18(3):503-13. doi:
10.1111/jcmm.12205.
30. Zhang W, Ou X, Wu X. Proteomics
profiling of plasma exosomes
in epithelial ovarian cancer: A potential role in the coagulation cascade, diagnosis and prognosis. Int
J Oncol. 2019;54(5):1719-33.
doi: 10.3892/ijo.2019.4742.
Fuentes de financiamiento: Instituto Nacional de Salud del Perú.
Citar como: Yareta J, Galarza M, Capristano
S, Pellón O, Ballon J, Guio H. Expresión diferencial
de micro-ARN circulantes en pacientes con tuberculosis activa y latente. Rev Peru Med
Exp Salud Publica. 2020;37(1): 51-6. Doi: https://doi.org/10.17843/rpmesp.2020.371.4468.
Correspondencia: Heinner Guio Chunga; Av. Defensores del Morro 2268, Chorrillos, Lima; heinnerguio@gmail.com
Conflictos de interés: Todos los autores declaran que no tienen conflicto
de interés alguno.
Recibido:
16/04/2019
Aprobado:
29/01/2020
En línea:
23/03/2020