María V. Méndez
Ysvette Vásquez
Dayana Requena
10.17843/rpmesp.2020.372.4652
ARTÍCULO ORIGINAL
Tipificación del cassette cromosómico estafilocócico de Staphylococcus aureus resistentes al meticilino en el estado de Aragua, Venezuela
Typification of the staphylococcal chromosome cassette of methicillin-resistant Staphylococcus aureus in the state of Aragua, Venezuela
Betsi Bastidas
![]() 1, Magister en Medicina Veterinaria
1, Magister en Medicina Veterinaria
María V. Méndez
![]() 1, doctora en Ciencias
1, doctora en Ciencias
Ysvette Vásquez
![]() 2, Magister en Medicina Veterinaria
2, Magister en Medicina Veterinaria
Dayana Requena ![]() 3, magister en Ciencias Biomédicas
3, magister en Ciencias Biomédicas
1 Escuela de
Bioanálisis, Facultad de Ciencias de la Salud, Universidad de Carabobo,
Aragua, Venezuela.
2 Laboratorio de Bacteriología, Hospital de los Samanes, Aragua,
Venezuela.
3 Instituto de Investigaciones Biomédicas Dr. Francisco Triana,
Universidad de Carabobo, Aragua, Venezuela.
RESUMEN
Objetivos: Tipificar el casette SCCmec en cepas de Staphylococcus aureus resistentes a meticilino (SARM) en aislados clínicos de centros de salud del Estado Aragua-Venezuela y comparar la presencia de los genotipos SCCmec entre los centros de salud del estado y según el tipo de infección.
Materiales y métodos: Durante enero y agosto de 2015 se estudiaron 81 cepas SARM de cuatro centros de salud del estado de Aragua en Venezuela. La resistencia al meticilino se midió con el método de Kirby-Bauer con discos de oxacilina (1 µgr) y cefoxitina (30 µgr). El gen mecA y el SCCmec se analizaron por la técnica de reacción en cadena de polimerasa múltiple.
Resultados: 55 aislados (67,9%) amplificaron el gen mecA, y 24 cepas (43,6%) amplificaron el SCCmec. El SCCmec I fue el más frecuente, seguido de SCCmec IV y SCCmec III, representaron el 62,5%, 25% y 12,5%, respectivamente. El SCCmec I fue predominante en el centro de salud A (80%), mientras que el SCCmec IV se encontró en el centro de salud B (60%) y C (100%). En el centro de salud D, 50% resultó ser SCCmec I y 50% SCCmec IVd. Se encontró relación entre el SCCmec y el centro de salud con significancia estadística. En infecciones de piel y tejidos blandos y en las respiratorias predominó el SCCmec I con 63,2% y 50% respectivamente.
Conclusiones: La frecuencia de SCCmec I y IV permitirá establecer nuevas medidas en el uso y control de la resistencia a los antibióticos.
Palabras claves: Staphylococcus aureus; SCCmec; Gen mecA; Epidemiología Molecular; Staphylococcus aureus resistentes a meticilina (fuente: DeCS BIREME).
ABSTRACT
Objective: Typify the SCCmec cassette in methicillin-resistant strains of Staphylococcus aureus in clinical isolates from health centers in the State of Aragua-Venezuela and compare the presence of SCCmec genotypes among the state health centers and according to the type of infection.
Materials and methods: 81 MRSA strains from four health centers of the Aragua-Venezuela State were studied. Methicillin resistance was performed with the Kirby-Bauer method with oxacillin (1 µg) and cefoxitin (30 µg) disks. The mecA gene and SCCmec were analyzed by the multiple PCR technique.
Results: Only 55 isolates (67.9%) amplified the mecA gene, and 24 strains (43.6%) amplified SCCmec. SCCmec type I was the most frequency, followed by SCCmec IV and SCCmec III, representing 62.5%, 25% and 12.5%, respectively. SCCmec I was predominant in health center A (80%), while in B and C 60% and 100% respectively were SCCmec IV. At health center D, 50% turned out to be SCCmec I and 50% SCCmec IVd. A relationship was found between the SCCmec and the health center with statistical significance. SCCmec I predominated in skin and soft tissue and respiratory infections with 63.2% and 50%, respectively. There was no association between genotype and type of infection with a p value greater than 0.05.
Conclusions: The prevalence of SCCmec I and IV will allow establishing new measures in the use of antibiotics and epidemiological control.
Keywords: Staphylococcus aureus; SCCmec; mecA gene; Molecular Epidemiology; Staphylococcus aureus methicilin resistent (source: MeSH NLM).
INTRODUCCIÓN
El Staphylococcus aureus resistente al meticilino (SARM) es un problema de salud pública en el mundo, que causa infecciones graves en los centros hospitalarios y en la comunidad. En el 2018, la Organización Mundial de la Salud (OMS) estimó que la probabilidad de morir de los pacientes con infecciones por SARM es un 64% superior a los pacientes con infecciones no resistentes (1). Asimismo, para el 2017 la OMS incluyó al SARM en la lista de los doce patógenos más peligrosos para la salud humana por su resistencia a los antibióticos (2).
La resistencia a la meticilina se debe a que la bacteria sintetiza una proteína fijadora de penicilina conocida como PBP2a, que posee baja afinidad a la meticilina y al resto de los antibióticos betalactámicos, lo que impide el ingreso de este tipo de antibióticos al interior de la célula bacteriana para ejercer su efecto antimicrobiano. La PBP2a es codificada por el gen mecA, que se encuentra dentro de un elemento cromosómico móvil, denominado cassette cromosómico estafilocócico (SCCmec). El gen mecA se encuentra distribuido tanto en el S. aureus como en otras especies de estafilococos coagulasa negativa resistentes al meticilino (3,4).
El SCCmec puede medir entre 21 y 67 Kb, y tiene un conjunto de genes como el ccr (ccrAB y ccrC) que codifican recombinasas, además del complejo mec que contiene al gen mecA, sus genes reguladores (mecI, mecR), los determinantes genéticos adquiridos, que se producen como resultado de la integración de plásmidos y transposones, y finalmente, la secuencia de la región J (5). La importancia de conocer la constitución del SCCmec reside en que de acuerdo a los eventos de recombinación entre los genes ccr y mecA se han generado una variedad de SCCmec que permiten clasificar el SARM según el SCCmec que posee. En ese sentido, se describieron inicialmente cinco tipos de SCCmec (I-V) y un determinado número de variantes o subtipos (5,6); sin embargo, recientemente se publicaron nuevos tipos como SCCmec VI-XI (7).
Además, los SCCmec se diferencian entre sí por sus determinantes de resistencia, de tal manera que los SCCmec I, IV, V, VI y VII codifican para resistencia exclusivamente a los antibióticos betalactámicos mientras que los SCCmec II, III y VIII poseen genes adicionales de resistencia a múltiples clases de antibióticos distintos a los antibióticos betalactámicos (5,8).
Por otra parte, las cepas del SARM pueden ser adquiridas en el ambiente hospitalario (SARMAH) o en la comunidad (SARMAC). El SARMAC se caracteriza por ser sensible a múltiples antibióticos y, generalmente, solo es resistente a antibióticos betalactámicos, causa infecciones de piel y partes blandas, incluso casos graves de neumonía necrotizante, fascitis necrotizante, tromboflebitis séptica y sepsis (9,10). El SARM-AH es resistente a varios grupos de antibióticos, además de los betalactámicos, y está relacionado con pacientes que presentan factores de riesgo como el consumo elevado de antibióticos, estancias hospitalarias prolongadas, procedimientos invasivos (catéteres intravenosos, sondas urinarias, traqueotomía), úlceras de decúbito, enfermedades graves y contacto con pacientes colonizados por el SARM (9,10). Asimismo, el SARM-AC es portador de los SCCmec IV y V (5), mientras que las cepas del SARMAH tienen el SCCmec I, II o III (11).
La tipificación molecular del SCCmec se realiza por la reacción en cadena de la polimerasa (PCR, por sus siglas en inglés), empleando la técnica de PCR múltiple, que permite determinar de manera simultánea diferentes tipos de SCCmec, de gran utilidad en estudios epidemiológicos (12,13). En ese sentido, Acuña et al (14), aplicaron la técnica de PCR múltiple para tipificación de SCCmec en 21 cepas del SARM aisladas en el laboratorio de bacteriología de un hospital en Cumaná, en el estado de Sucre, donde encontraron SCCmec I y IV en pacientes ambulatorios y de la emergencia de adultos. La presencia de genotipos SCCmec IV indicó que los aislamientos eran de origen comunitario y se estaban diseminando a los servicios hospitalarios, produciendo infecciones nosocomiales. En el estado de Zulia, González et al (15), caracterizaron los SCCmec de 54 cepas SARM por PCR múltiple y evidenciaron que el 54% tenían SCCmec IV; el 40%, SCCmec I; mientras que un 4% y 2%, el SCCmec IA y el SCCmec IIIB, respectivamente.
En el estado de Aragua no se han realizado estudios previos que hayan determinado el tipo de SCCmec circulante en los centros de salud de la región, razón por la cual el objetivo de la presente investigación fue tipificar el SCCmec en cepas del SARM aisladas de centros de salud de Aragua.
|
MENSAJES CLAVE |
|
Motivación para realizar el estudio: El Staphylococcus aureus resistente al meticilino (SARM) es un problema de salud pública. La resistencia al antibiótico se debe al gen mecA ubicado en el cassette cromosómico SCCmec. El tipo SCCmec diferencia entre el SARM adquirido en el hospital o en la comunidad, y predice los posibles genes de resistencia a antibióticos diferentes a los betalactámicos. Pocos estudios se han hecho en Venezuela, y en el estado de Aragua es la primera investigación que se realiza en cuatro hospitales. Principales hallazgos: El hallazgo fue la alta frecuencia del SARM con SCCmec I de origen hospitalario.. Implicancias: La investigación contribuirá a establecer medidas para el control epidemiológico y para el uso de antibioticoterapia en cuatro centros de salud del estado de Aragua, Venezuela. |
MATERIALES Y MÉTODOS
Diseño del estudio
El estudio fue descriptivo y de corte transversal, realizado entre enero y agosto del 2015 en pacientes que asistieron a cuatro centros de salud del estado Aragua, en Venezuela, denominados A, B, C y D en la presente investigación. El centro de salud A es un hospital privado que cuenta con 72 camas, consulta ambulatoria y hospitalización. El centro de salud B es un centro de atención público para pacientes con complicaciones de pie diabético, que atiende en promedio 70 personas por día y no posee hospitalización. El centro de salud C es público, de atención preventiva para pacientes adultos y pediátricos con capacidad de atención para 2000 personas, mientras que el centro D es público y el más grande de la región, tiene capacidad de atención para 400 000 personas y 551 camas de hospitalización.
El aislamiento de las cepas de estafilococos se realizó de muestras de infecciones de piel, tejido blando, secreciones óticas, oculares, respiratorias, catéteres, entre otras; se identificó si las muestran procedían de pacientes hospitalizados o ambulatorios.
Estas muestras fueron inoculadas en placas de agar sangre e incubadas de 35±2 °C en aerobiosis durante 16-18 horas. Para la identificación bacteriana se emplearon los procedimientos estándares descritos en la literatura (16). Finalmente, de un total de 404 cultivos positivos para estafilococos, en 324 se aisló el S. aureus. Las cepas fueron preservadas a -20 °C en glicerol hasta el momento del estudio.
Prueba de susceptibilidad antimicrobiana
Se aplicó el método de difusión de disco en agar o Kirby- Bauer, siguiendo los lineamientos del Instituto para la Estandarización de Laboratorios Clínicos para la identificación de SARM (17). Se utilizó el disco de cefoxitina de 30 μg (BD) y de oxacilina de 1 μg (BD). La cepa control utilizada fue S. aureus ATCC 25923.
Extracción del ADN
Se realizó a partir de un cultivo puro del SARM en agar sangre luego de 18-24 horas de incubación. Se preparó una suspensión en un tubo Eppendorf, tomando de 1 a 5 colonias del microorganismo que se colocaron en 50 µL de agua destilada estéril; posteriormente se sometió a ebullición a 99 °C por 10 min. Finalmente, se centrifugó a 30 000 / g por 1 min y se transfirió el sobrenadante a un nuevo tubo Eppendorf. El ADN concentrado se preservó a –20 °C hasta el momento del ensayo (13).
Detección del gen mecA y genotipos SCCmec de cepas SARM
El ensayo de PCR múltiple para identificar el tipo de casette SCCmec y las condiciones para la amplificación, se realizaron según la metodología descrita previamente por Zhang et al (13), y consistió en utilizar nueve pares de cebadores, incluyendo los cebadores específicos para los tipos y subtipos SCCmec I, II, III, IVa, IVb, IVc, IVd y V y los cebadores del gen mecA. Se amplificaron ocho loci distintos, con base en las secuencias que se presentan en la Tabla 1. Para la reacción de PCR se utilizaron las siguientes condiciones: para la mezcla Máster Mix, 50 mM KCl, 20 mM Tris-HCl (pH 8,4); 2,5 mM de MgCl2, 0,2 mM de cada desoxinucleótidos trifosfato (dATP, dUTP, dGTP y dCTP). Las concentraciones de los cebadores se encuentran en la Tabla 1. Adicionalmente, se utilizó una unidad de Go Taq Flexi DNA Polymerase® (Promega Corp., EUA).
Tabla 1. Secuencias de los iniciadores que amplifican cada uno de los locis del cassette cromosómico estafilocócico (SCCmec)
Fuente: Zhang et al (13)
Para el control de calidad de las pruebas de tipificación molecular se emplearon las cepas del S. aureus ATCC 259233 (sensible al meticilino) como control negativo y del S. aureus ATCC 43300 (resistente al meticilino) como control positivo del gen mecA. El producto de la amplificación se sometió a migración electroforética en geles de agarosa al 2% a 100 v por 30 min. Se utilizó un marcador de tamaño molecular de 100 pb (New England Biolabs, Inc). Finalmente, la longitud del amplicón se comparó con los valores en el tamaño molecular registrados en la tabla 1 correspondientes al gen mecA y los tipos y subtipos de SCCmec.
Análisis estadístico
Se utilizó una base de datos en Excel 2007 de Windows XP, donde se recopiló la procedencia de las cepas del SARM. El análisis estadístico de los datos se realizó con el programa EpiInfo 3.5.1. Se realizaron análisis descriptivos con frecuencias y porcentajes. Se utilizó la prueba de Chi cuadrado con un nivel de significancia de p < 0,05 para determinar si existen diferencias según algunas características en la procedencia de las cepas.
Aspectos éticos
El proyecto fue evaluado y aprobado por el comité de Bioética de la Coordinación de Docencia e Investigación del Servicio Autónomo Hospital Central de Maracay. Adicionalmente, los pacientes que participaron en el estudio firmaron un consentimiento informado.
RESULTADOS
Durante el período de estudio se analizaron un total de 404 cultivos positivos para estafilococos provenientes de los cuatro centros de salud del estado de Aragua, 80 cepas (19,8%) eran estafilococos coagulasa negativo (SCN) y 324 (80,2%) fueron S. aureus, de las cepas analizadas 81 (25%) fueron SARM.
La detección de la presencia del gen mecA arrojó como resultado que, de las 81 cepas del SARM, solo 55 aislados (67,9%) amplificaron el gen mecA y 26 (32,1%) no lo amplificaron, con un intervalo de confianza de entre 56,6% y 77,8% y con 95% de nivel de confianza. Entre los 55 aislados que dieron un resultado positivo para el gen mecA, solo 24 (43,6 %) amplificaron algún tipo de SCCmec, mientras que en 31 aislados (56,4%) no se obtuvo amplificación con intervalos de confianza de entre 30,3% - 57,7% y 42,3%-68,7%, respectivamente (Figura 1A, B y C).
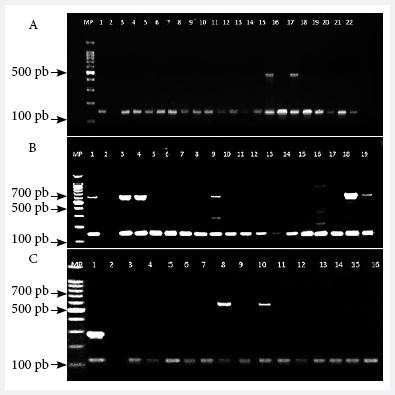
Figura 1. Electroforesis en geles de agarosa al 2% de los productos de la amplificación por PCR de genotipos SCCmec y el gen mecA. A) MP: marcador de peso molecular de 100 pb; carril 1: control positivo; carril 2: control negativo; carriles 3 al 22 aislados SAMR donde se observa banda de 147 pb correspondiente al gen mecA; carriles 15 y 17 presentan banda adicional de 493 bp correspondiente al SCCmec IVb. B) MP: marcador de peso molecular de 100 pb; carril 1: control positivo; carril 2: control negativo; carriles 3 al 19 aislados SAMR donde se observa banda de 147 pb correspondiente al gen mecA; la banda de 881 pb es SCCmec IVd, de 613 pb es SCCmec I, de 395 pb corresponde a SCCmec II, de 325 pb es SCCmec V y de 200 pb IVc; carriles 1, 3, 4, 9, 18 y 19: cepas genotipo SCCmec I; carriles del 5 al 8, del 10 al 17: no hubo amplificación con los SCCmec incluidos en el estudio. C) MP: marcador de peso molecular de 100 pb; carril 1: control positivo; carril 2: control negativo; carriles 3 al 14 aislados SAMR donde se observa banda de 147 pb correspondiente al gen mecA; carriles 8 y 10: cepas genotipos SCCmec I.
De las 24 cepas del SARM que amplificaron SCCmec se encontró que el casette más predominante entre los aislados fue el casette SCCmec tipo I, seguido de SCCmec IV (subtipos IVb y IVd) y SCCmec III representado por el 62,5%, 25% y 12,5%, respectivamente. No se encontraron los SCCmec II y SCCmec V (Tabla 2).
Tabla 2. Frecuencia de genotipos del cassette cromosómico estafilocócico y su distribución según centros de salud y tipo de muestra
|
Característica |
Tipo de cassette cromosómico estafilocócico (SCCmec) |
|
|||||
|
I |
II |
III |
IVb |
IVd |
V |
Total |
|
|
n (%) |
n (%) |
n (%) |
n (%) |
n (%) |
n (%) |
n (%) |
|
|
Número de aislamientos |
15 (62,5) |
0 (0) |
3 (12,5) |
3 (12,5) |
3 (12,5) |
0 (0) |
24 (100) |
|
Centro de salud |
|
|
|
|
|
|
|
|
A |
12 (80) |
0 (0) |
3 (20) |
0 (0) |
0 (0) |
0 (0) |
15 (100) |
|
B |
2 (40) |
0 (0) |
0 (0) |
2 (40) |
1 (20) |
0 (0) |
5 (100) |
|
C |
0 (0) |
0 (0) |
0 (0) |
1 (50) |
1 (50) |
0 (0) |
2 (100) |
|
D |
1 (50) |
0 (0) |
0 (0) |
0 (0) |
1 (50) |
0 (0) |
2 (100) |
|
Tipo de muestra |
|
|
|
|
|
|
|
|
Hemocultivo |
1 (100) |
0 (0) |
0 (0) |
0 (0) |
0 (0) |
0 (0) |
1 (100) |
|
Piel y tejidos blandos |
12 (63) |
0 (0) |
2 (10) |
3 (16) |
2 (10) |
0 (0) |
19 (100) |
|
Respiratorio |
2 (50) |
0 (0) |
1 (25) |
0 (0) |
1 (25) |
0 (0) |
4 (100) |
El mayor número de cepas del SARM en las que hubo amplificación de algún tipo de SCCmec fueron encontradas en el centro de salud A (15 cepas), donde se encontró que el SCCmec I fue predominante (80%), seguido del SCCmec III (20%). En el caso del centro de salud B, un total de 5 cepas amplificaron SCCmec, donde se encontró que tres de ellas (60%) resultaron ser SCCmec IV (subtipos IVb y IVd) y 40%, SCCmec I. En cuanto al centro de salud C, se obtuvieron dos cepas SARM que amplificaron SCCmec IV, una amplificó el subtipo IVb y la otra el subtipo IVd. En el centro salud D, solo dos aislados del SARM amplificaron SCCmec, uno de ellos resultó ser SCCmec I y la otra cepa SCCmec IVd. Se encontró relación entre el genotipo aislado y el centro de salud (p = 0,032) (Tabla 2).
De las 24 cepas analizadas, 19 se aislaron en piel y tejidos blandos, cuatro en infecciones respiratorias y 1 de hemocultivo. De los 19 aislados provenientes de piel y tejidos blandos, el SCCmec I fue el más predominante (63,2%), seguido de SCCmec IV (26,3%), donde 15,8% eran del subtipo IVb, y 10,5%, del subtipo IVd. En las muestras respiratorias se obtuvieron cuatro cepas que amplificaron SCCmec, de estas, dos (50%) portaban el SCCmec I, mientras que una resultó ser SCCmec III y una SCCmec IVd. No se encontró relación entre el genotipo y el tipo de infección (p = 0,870) (Tabla 2).
DISCUSIÓN
El 25% del SARM en el presente estudio fue similar a los datos obtenidos por Dorante et al (18), quienes encontraron que de 117 aislados de S. aureus en un hospital del estado de Aragua, 24,7% eran SARM. Asimismo, Chavez et al (19)reportaron que, en un hospital de Medellín (Colombia), de 35 aislados, 28,6% resultaron ser SARM. Sin embargo, Guillen et al (20) encontraron en su estudio en Paraguay que, de 77 cepas, el 18,7% fueron SARM, ligeramente más bajo al reportado en la presente investigación.
Por otra parte, la detección de SARM utilizando la identificación del gen mecA por la técnica de PCR, arrojó discrepancias con respecto a los resultados obtenidos con el uso de los discos de oxacilina y cefoxitina, ya que de los 81 aislados de SARM, solo 55 amplificaron el gen mecA. Otros estudios han reportado resultados similares, tal es el caso de Acuña et al (14), quienes observaron que de 21 cepas SARM, solo 19 amplificaron el gen mecA. Caso contrario, las investigaciones de Chávez et al (19) y Guillen et al (20) reportaron que todas las cepas SARM estudiadas poseían el gen mecA. En el presente estudio, a las cepas que no amplificaron el gen mecA se les corroboró la identificación y sus perfiles fenotípicos de resistencia al meticilino.
Según los resultados obtenidos, es posible que la resistencia esté relacionada a algún mecanismo diferente a la expresión de las PBP2a. Uno de ellos puede ser la hiperproducción de β-lactamasas por cepas de S. aureus, conocidas como BORSA (del inglés Borderline oxacillinresistant S.aureus) (21). Adicionalmente, sería posible inferir que la ausencia de amplificación del gen mecA pueda deberse a que la cepa sea portadora del gen mecC, no detectable por los métodos convencionales, y responsable de un 2% de infecciones por SARM en humanos. El gen mecC presenta un 70% de homología con el gen mecA y sintetiza una transpeptidasa con 60% de homología a la PBP2a (22).
En relación con la tipificación del SCCmec, 24 cepas amplificaron algún tipo de SCCmec, encontrándose como más predominante el SCCmec I, seguido de SCCmec IV (subtipos IVb y IVd) y en menor proporción el SCCmec III. La distribución de los genotipos SCCmec fue diferente en los centros de salud A, B, C y D, siendo el centro de salud A en el que se concentró el mayor número de cepas, con un claro predominio del SCCmec I y, en menor frecuencia, del tipo III, lo que confirma su origen hospitalario y demuestra que aquellos aislados que portan el tipo III, deben poseer resistencia a una gran variedad de antibióticos diferentes a los β-lactámicos (5,8). El centro B ocupó la segunda posición de acuerdo al total de aislados, con predominio de SCCmec tipo IV (subtipos IVb y IVd), seguido del tipo I. La presencia de SCCmec IV en el centro de salud B, un centro para pacientes ambulatorios con complicaciones de pie diabético, parece indicar su origen comunitario (5,8). Sin embargo, en el mismo centro, el hallazgo de SCCmec I está relacionado con las cepas SARM-AH (5,8), lo que pudiese predecir la posible diseminación de cepas adquiridas en el ambiente hospitalario en este centro de salud.
Adicionalmente, en los centros de salud C y D se encontró principalmente el SCCmec tipo IV. Sin embargo, fue bajo el número de cepas SARM asociadas a algún tipo de SCCmec, lo que pudo deberse a la escasez de recursos en los laboratorios de bacteriología que condicionan la detección e identificación de cepas SARM. La baja cantidad de cepas SARM y de SCCmec detectadas se considera una limitación para interpretar los resultados del presente estudio, por lo que serán necesarias nuevas investigaciones que permitan profundizar la distribución de SCCmec en ambos centros de salud, siendo el centro de salud D el más grande e importante de la región.
Los resultados relacionados con la alta frecuencia de SCCmec I son similares a los reportados en otras regiones de Venezuela y en otras ciudades del continente. De hecho, Acuña et al (14), encontraron en un hospital de la ciudad de Cumaná en Venezuela el predominio de SCCmec I (14 de 19 aislados SARM), seguido de SCCmec IV (3 de 19 cepas SARM). En Valdivia (Chile), se identificó la presencia de SCCmec I, seguido de SCCmec IV (23). Las investigaciones citadas coinciden además con el predominio de SCCmec I hallado en el centro de salud A. Por otra parte, la presencia de SCCmec IV en los centros de salud B y C, coincide con lo planteado por Romero et al (24) y Castellano et al (25) en hospitales del estado de Zulia, así como con lo referido por Sánchez et al (26) en hospitales de Medellín (Colombia). Un estudio realizado en un hospital de la ciudad de Cali (Colombia) (19), reportaron que 26,6% de las cepas SARM portaban el SCCmec II, a diferencia de lo observado en el presente estudio, donde no se obtuvo en los cuatro centros de salud investigados. Este último resultado podría deberse a que existan diferencias en el predominio y distribución de SCCmec entre los hospitales y el área geográfica de referencia. De hecho, en los estudios publicados hasta la fecha en otras regiones de Venezuela no se ha identificado el SCCmec II (14,15,24,25).
Identificar la presencia de SCCmec I en los centros de salud del estado de Aragua podría mejorar las opciones terapéuticas para el tratamiento de las infecciones por SARM (5,8). Sin embargo, la presencia de SCCmec IV también gana relevancia, por su resistencia exclusiva a los antibióticos betalactámicos y por estar relacionado con el SARM-AC (5,8). Los aislamientos del SARM-AC que poseen SCCmec IV, y en menor frecuencia el tipo V, también presentan los genes de la toxina Leucocidina de Panton-Valentine (LPV) (5), mientras que las cepas del SARM adquirido en el ambiente hospitalario, poseen el SCCmec II o III, y en muy pocos casos se ha aislado la LPV (5,8), es por ello que se ha propuesto que la determinación de LPV en cepas hospitalarias portadoras del SCCmec IV permite corroborar su origen y esclarecer el panorama epidemiológico (5,8).
Relacionado al tipo de infección, la mayoría de las cepas tipificadas provenían de piel y tejidos blandos, con predominio del SCCmec I, confirmando el origen hospitalario de las infecciones y proyectando el manejo de la antibioticoterapia, ya que el tipo I es portador de resistencia a antibióticos betalactámicos (5,8). Los resultados del presente estudio difieren de los encontrado por Romero et al. (24), quienes encontraron un alto porcentaje de aislamientos con SCCmec IV en muestras de piel y tejido blando.
De los 55 aislados SARM que amplificaron el gen mecA, en 56,4% no hubo amplificación del SCCmec, por lo que es posible inferir que se emplearon los cebadores para detectar los SCCmec del I al V y sus subtipos propuestos por Zhang et al. (13), lo que representó otra limitación para el presente estudio, por lo que se sugiere aplicar los cebadores específicos para los nuevos SCCmec descritos (7). La posible existencia de otros SCCmec indicaría la presencia de otros genotipos con nuevos determinantes de resistencia a antibióticos.
En conclusión, la identificación del SARM por tipificación de SCCmec, permitió evidenciar el predominio del SCCmec I y III relacionados al SARM adquiridos en el ambiente hospitalario y del SCCmec tipo IV asociado a la comunidad. Se evidenció que existe relación entre el genotipo aislado y el centro de salud. En muestras de piel y tejidos blandos predominó el SCCmec I; sin embargo, no se encontró relación entre los SCCmec y el tipo de infección.
Se recomienda llevar a cabo estudios prospectivos para la detección de SCCmec con la inclusión de los principales centros de salud de la región, el uso de métodos alternativos que verifiquen la meticilino resistencia, y la introducción de los nuevos cebadores para la identificación de la existencia de otros SCCmec. Además, se recomienda incluir la valoración de hiperproducción de betalactamasas y la determinación de las concentraciones mínimas inhibitorias de oxacilina para aquellas cepas del SARM que no amplifican el gen mecA.
REFERENCIAS BIBLIOGRÁFICAS
1. Organización Mundial de la Salud. 2018. Comunicado de Prensa. Disponible en: https://www.who.int/es/news-room/fact-sheets/detail/resistencia-a-los-antimicrobianos.
2. Organización Mundial de la Salud. 2017. Comunicado de Prensa. Disponible en: https://www.who.int/es/news-room/detail/27-02-2017-who-publishes-list-of-bacteria-for-which-new-antibiotics-are-urgently-needed.
3. Hiramatsu K, Kuroda M, Ito T. The emergence and evolution of methicillin-resistant Staphylococcus aureus. Trends. Microbiol. 2001;9(10):486-493. doi: https://doi.org/10.1016/s0966-842x(01)02175-8.
4. Cortés J, Gómez C, Cuervo S, Leal A. Implicaciones en salud Pública de Staphylococcus aureus meticilino resistente adquirido en la comunidad en Bogotá, Colombia. Rev. Salud Pública. 2007;9(3): 448–454. Disponible en: https://www.scielosp.org/pdf/rsap/2007.v9n3/448-454/es.
5. Ito T, Katayama Y, Asada K, Namiko T, Kanae T, Chuntima T et al. Structural comparison of three types of staphylococcal cassette chromosome mec integrated in the chromosome in methicillin- resistant Staphylococcus aureus. Antimicrob. Agents Chemother. 2001;45(5):1323–1336. doi: 10.1128/AAC.45.5.1323-1336.2001.
6. Oliveira D, Tomasz A, De Lancastre H. The evolution and pandemic clones of methicillin-resistant Staphylococcus aureus: Identification of two ancestral genetics brack grounds and the associated mec elements. Microb. Drug. Resist. 2001;7(4):349-361. doi: 10.1089/10766290152773365.
7. Liu J, Chen D, Peters B M, Li L, Li B, Xu Z, et al. Staphylococcal chromosomal cassettes mec (SCCmec): A mobile genetic element in methicillin-resistant Staphylococcus aureus. Microb Pathog. 2016;101:56-67. doi: https://doi.org/10.1016/j.micpath.2016.10.028.
8. Zhang K, McClure JA, Elsayed S, Conly JM. Novel staphylococcal cassette chromosome mec type, tentatively designated type VIII, harboring class A mec and type 4 ccr gene complexes in a Canadian epidemic strain of methicillin-resistant Staphylococcus aureus. Antimicrob Agents Chemother. 2009; 53(2):531–540. 10.1128/AAC.01118-08.
9. Canoa M, Domínguez M, Ezpeletac C, Padillad B, Arellano E y Martínez L. Cultivos de vigilancia epidemiológica de bacterias resistentes a los antimicrobianos de interés nosocomial. Enferm Infecc Microbiol Clin. 2008;26(4):220-229. doi: 10.1016/S0213-005X(08)72694-6.
10. Pérez N, Pavas N , Rodríguez E. Resistencia de Staphylococcus aureus a los antibióticos en un hospital de la Orinoquia colombiana. Asociación Colombiana de Infectología. Rev Infect. 2010;14(3):167-173. doi: 10.1016/S0123-9392(10)70108-9.
11. Cavalcante F, Schuenck R, Caboclo R, Ferreira D de C, Nouér S, Dos Santos K. Tetra cycline and trimethoprim/sulfamethoxazole at clinical laboratory: can they help to characterize Staphy lococcus aureus carrying different SCCmec types?. Rev Soc Bras Med Trop. 2013;46(1):100-102. doi: 10.1590/0037-868216062013.
12. Faria N, Carrico J, Oliveira D, Ramírez M, De Lencastre H. Analysis of typing methods for epidemiological surveillance of both methicillin–resistant and methicillin susceptible Staphylococcus aureus strains. J. Clin. Microbiol. 2008; 46(1):136–144. doi: 10.1128/JCM.01684-07.
13. Zhang K, McClure J, Elsayed S, Louie T. Novel Multiplex PCR Assay for Characterization and Concomiitant Subtyping of Staphylococcal Cassette Chromosome mec Types I to V in Methicillin-Resistant Staphylococcus aureus. J. of Clin. Microbiol. 2005;43(10):5026-5033. doi: 10.1128/JCM.43.10.5026-5033.2005.
14. Acuña S, Sánchez E y Patiño L. Tipificación de la meticilino resistencia en cepas de Staphylococcus spp. Hospital Universitario de Alcalá, Cumana, Estado Sucre, Venezuela. Rev. Soc. Ven. Microbiol. Caracas. 2014;34(1):1-8. Disponible en: http://ve.scielo.org/pdf/rsvm/v34n1/art03.pdf.
15. González M, Cavazza M, Perozo A. Tipo de cassette cromosómico estafilocócico en cepas clínicas de Staphylococcus aureus resistentes a meticilina. Kasmera. 2014;42(2):116-130. Disponible en: http://ve.scielo.org/pdf/km/v42n2/art04.pdf.
16. Koneman E., Allen S., Janda, W., Schreckenberg P. y Winn W. Diagnóstico Microbiológico. (5ª Ed.). Madrid: Panamericana; 2008.
17. Instituto de Estándares Clínicos y de Laboratorio. Normas de funcionamiento de los antimicrobianos, Las pruebas de susceptibilidad de disco. Undécima edición. Estados Unidos. Estados Unidos; 2012.
18. Dorante V, Hurtado E, Martínez B, Méndez MV. Frecuencia de Staphylococcus aureus meticilino resistente en pacientes que asisten al laboratorio de microbiología del hospital "Los Samanes" estado Aragua. Odous Científica. 2013;14(1):29-36. Disponible en: http://servicio.bc.uc.edu.ve/odontologia/revista/vol14-n1/art04.pdf.
19. Chávez M, Martínez A, Esparza M. Caracterización de Staphylococcus aureus obtenido del ambiente hospitalario y del personal de salud en un Hospital de la Ciudad de Cali. Rev Biosalud. 2017;16(2):22-33. doi: 10.17151/biosa.2017.16.2.3.
20. Guillén R, Carpinelli L, Rodríguez F, Castro H, Quiñónez B, Campuzano A. Staphylococcus aureus adquiridos en la comunidad: caracterización clínica, fenotípica y genotípica de aislados en niños paraguayos. Rev Chilena Infectol. 2016;33(6): 609-618. doi: 10.4067/S0716-10182016000600002.
21. Velásquez-Meza, M. Surgimiento y Diseminación de Staphylococcus aureus meticilino resistente. Salud Pública Mex. 2005;47:381-387. Disponible en: http://www.scielo.org.mx/pdf/spm/v47n5/28384.pdf.
22. Ito T, Hiramatsu K, Tomasz A, de Lencastre H, Perreten V, Holden M T, et al. Guidelines for reporting novel mecA gene homologues. Antimicrob Agents Chemother. 2012;56(10):4997-4999. doi: 10.1128/AAC.01199-12.
23. Medina G, Egea A L, Otth C, Otth L, Fernández H, Bocco J L, et al. Molecular epidemiology of hospital-onset methicillin-resistant Staphylococcus aureus infections in Southern Chile. Eur J Clin Microbiol Dis. 2013;32(12):1533-1540. doi: https://doi.org/10.1007/s10096-013-1907-8.
24. Romero A, Castellano M, Perozo M, Armindo J, Rincón G, Zabala D. Detección de cassette cromosómico en cepas de Staphylococcus aureus resistente a meticilina aisladas en un hospital universitario de la ciudad de Maracaibo. Kasmera. 2018; 46(1):40-51. Disponible en: https://produccioncientificaluz.org/index.php/kasmera/article/view/24652/pdf.
25. Castellano M, Cavazza J, Porro M, Perozo M, Armindo J. Tipo de cassette cromosómico estafilocócico en cepas clínicas de Staphylococcus aureus resistentes a meticilina. Kasmera. 2014;42(2):116-130. Disponible en: https://produccioncientificaluz.org/index.php/kasmera/article/view/19528/19492.
26. Sánchez M, Hernández O, Velásquez L, Rivas D, Marín A, González L, et al. Caracterización del gen mecA de Staphylococcus aureus resistentes a meticilina aislados de tres grupos poblacionales de la ciudad de Medellín. Infectio. 2013;17(2):66-72. doi: 10.1016/S0123-9392(13)70165-6.
Fuentes de financiamiento: El estudio fue financiado por el Centro de Desarrollo Científico y Humanístico de la Universidad de Carabobo-Venezuela (CDCHUC).
Citar como: Bastidas B, Méndez MV, Vásquez Y, Requena D. Tipificación del cassette cromosómico estafilocócico de Staphylococcus aureus resistentes al meticilino en el estado de Aragua, Venezuela. Rev Peru Med Exp Salud Publica. 2020;37(2):239-45. doi: https://doi.org/10.17843/rpmesp.2020.372.4652.
Correspondencia: María V. Méndez; mvmendezster@gmail.com
Contribuciones de autoría: BB, MVM, YV y DR han participado en la concepción del artículo, la recolección de datos, su redacción y aprobación de la versión final.
Conflictos de interés: Las autoras no presentan conflicto de interés en la publicación del artículo.
Recibido: 07/07/2019
Aprobado: 08/04/2020
En línea: 15/06/2020