
ARTÍCULO ORIGINAL
Mutaciones que confieren resistencia a fármacos antituberculosis de primera línea en Perú: una revisión sistemática de la literatura
Mutations conferring resistance to first-line anti-tuberculosis drugs in Peru: a systematic review of the literature
Aiko Vigo1,a, Lely Solari1,b, David Santos1,a, Zully M. Puyén.1,a,c
1 Instituto Nacional de Salud, Lima, Perú.
a Licenciado en Biología; b médica infectóloga, PhD
en Ciencias de la Salud; c
doctora en Microbiología.
RESUMEN
Objetivos. Sistematizar la información disponible referente a las mutaciones que confieren resistencia a los fármacos antituberculosis de primera línea. Materiales y métodos. Se realizó una revisión sistemática de la literatura científica para identificar artículos que reportaron mutaciones que confieren resistencia a fármacos antituberculosis de primera línea. Esta búsqueda hizo énfasis en la resistencia a los fármacos de isoniazida y rifampicina en cepas de M. tuberculosis de pacientes peruanos. La búsqueda fue realizada en PubMed y LILACS (Literatura Latinoamericana y del Caribe en Ciencias de la Salud). Resultados. Se incluyeron 14 artículos de los cuales tres reportaron mutaciones asociadas con resistencia a isoniazida, seis a rifampicina, ocho a pirazinamida y uno a etambutol. Todas las mutaciones a isoniazida o rifampicina fueron identificadas directa o indirectamente mediante la prueba de diagnóstico molecular GenoType MTBDRplus® v2.0. La mayor variabilidad de mutaciones fue determinada en la resistencia a pirazinamida. Conclusiones. Existe una gran variabilidad de mutaciones asociadas con resistencia a fármacos antituberculosis que han sido reportadas en Perú, y se sistematizan en el presente reporte. Estas mutaciones deben de ser tomadas en cuenta para el desarrollo de dispositivos diagnósticos o selección de pruebas diagnósticas a ser aplicadas en nuestro país.
Palabras clave: Mycobacterium tuberculosis; Resistencia a Medicamentos; Genotipo, Mutación (fuente: DeCS BIREME).
ABSTRACT
Objective. To systematize available information regarding mutations that confer resistance to first-line anti-tuberculosis drugs. Materials and Methods. A systematic review of the scientific literature was conducted to identify articles that reported mutations conferring resistance to first-line anti-tuberculosis drugs. This search emphasized resistance to isoniazid and rifampicin drugs in M. tuberculosis strains of Peruvian patients. The search was performed on PubMed and LILACS (Latin American and Caribbean Health Sciences Literature). Results. Fourteen (14) articles were included, of which three reported mutations associated with resistance to isoniazid, six to rifampicin, eight to pyrazinamide and one to ethambutol. All mutations to isoniazid or rifampicin were identified directly or indirectly by the molecular diagnostic test GenoType MTBDRplus® v2.0. The greatest variability of mutations was determined in resistance to pyrazinamide. Conclusions. There is a great variability of mutations associated with resistance to anti-tuberculosis drugs that have been reported in Peru, and they are systematized in this report. These mutations must be taken into account for the development of diagnostic devices or selection of diagnostic tests to be applied in our country.
Keywords: Mycobacterium tuberculosis; Drug Resistance; Genotype; Mutation (source: MeSH NLM).
La tuberculosis (TB) resistente a fármacos es un importante problema de salud pública a nivel global. En 2018, la Organización Mundial de la Salud (OMS) estimó que diez millones de personas desarrollaron la enfermedad. De estos casos, 500 000 tuvieron tuberculosis resistente a rifampicina, de los cuales el 78% fue tuberculosis multidrogo resistente (TB-MDR); es decir, presentaron resistencia simultánea a isoniazida y rifampicina (1). Además, se determinó que de los casos TB-MDR diagnosticados, el 7,3% fueron casos de tuberculosis extensamente drogorresistentes (TB-XDR); es decir, presentaban de manera adicional resistencia al menos a una fluoroquinolona (levofloxacino, ciprofloxacino o moxifloxacino) y a un aminoglucósido de segunda línea (capreomicina, amikacina, kanamicina). El Perú no es ajeno a las epidemias de TB-MDR y TB-XDR, encontrándose dentro de los 30 países con mayor carga de TB-MDR en el mundo (1), y reporta alrededor de 3000 casos prevalentes de TB-MDR anuales (2).
La resistencia a fármacos antituberculosis está determinada por mutaciones en el genoma de M. tuberculosis, que aparecen de manera espontánea o inducida por presión selectiva de los fármacos (3). Pacientes que toman esquemas terapéuticos inadecuados o que son poco adherentes pueden generar, en presencia de concentraciones subterapéuticas de los fármacos, proliferación de las cepas que presentan estas mutaciones genéticas de resistencia (3). Luego, estas cepas se pueden trasmitir por vía aérea a otras personas, quienes desarrollan tuberculosis resistente a fármacos sin previamente haber recibido ningún régimen terapéutico.
Actualmente, se conoce que las mutaciones presentes en el gen codificante de la «subunidad β de la RNA polimerasa» (gen rpoB) confieren resistencia a rifampicina. Asimismo, las mutaciones presentes en el gen «catalasa-peroxidasa» (gen katG) y en la región promotora del gen «enoil-ACP reductasa» (gen inhA) están asociadas con la resistencia a isoniazida (4). Por otro lado, los dispositivos de diagnóstico molecular utilizados en nuestro sistema de salud, tales como el GenoType MTBDRplus® v2.0 (Hain Lifescience) y Xpert® MTB/RIF (Cepheid, USA) detectan resistencia a los fármacos mencionados. Existen también mutaciones en otros genes que han sido identificadas como responsables de la resistencia a otros fármacos antituberculosis como pirazinamida (pncA), aminoglicósidos (rrs), fluoroquinolonas (gyrA), entre otros.
La presencia y/o ausencia de mutaciones puede variar significativamente de un país a otro, lo que podría hacer que los dispositivos diagnósticos que identifican las mutaciones más frecuentes en un país, no tengan el mismo rendimiento diagnóstico en otro (3). Por ello, el conocimiento de las mutaciones presentes en cepas de M. tuberculosis que circulan a nivel nacional, puede servir para desarrollar localmente nuevos dispositivos diagnósticos que mejoren la detección de resistencia (5).
El objetivo del presente estudio es sistematizar la información disponible referente a las mutaciones que confieren resistencia a los fármacos antituberculosis de primera línea, con énfasis en isoniazida y rifampicina, en publicaciones que describen cepas de M. tuberculosis procedentes de pacientes peruanos.

MATERIALES Y MÉTODOS
Se realizó una revisión sistemática de la literatura científica relacionada a la caracterización molecular de mutaciones presentes en cepas de M. tuberculosis con resistencia a fármacos antituberculosis de primera línea, según las recomendaciones de PRISMA (6). El estudio se enfocó en el análisis de la resistencia a isoniazida y rifampicina, y a las cepas de M. tuberculosis aisladas en pacientes peruanos.
CRITERIOS DE INCLUSIÓN Y EXCLUSIÓN
Los criterios de inclusión para las publicaciones consideradas en el estudio fueron en primer lugar, el idioma (artículos en inglés, español, francés y portugués);en segundo lugar, el diseño de estudio (reportes o series de casos, estudios observacionales o experimentales); en tercer lugar, que sean artículos con información explícita sobre la secuencia nucleotídica de la mutación que confiere la resistencia a fármacos de primera línea, principalmente isoniazida y rifampicina (independientemente de otros análisis adicionales que pudieran haber hecho, incluyendo pruebas no comerciales y comerciales); en cuarto lugar, que incluyeran cepas procedentes de pacientes peruanos, y por último, que hubiesen sido publicadas en los últimos diez años, con la finalidad de obtener un mejor reflejo de la diversidad de mutaciones que confieren resistencia en la actualidad.
Se excluyeron artículos que sólo incluyeron pruebas fenotípicas para diagnóstico de resistencia, cepas de pacientes de otros países que no fueran Perú y artículos cuyo objetivo fuera el estudio del linaje genético de la micobacteria, mas no su resistencia a fármacos antituberculosis.
ESTRATEGIA DE BÚSQUEDA BIBLIOGRÁFICA
Se realizaron búsquedas en las bases bibliográficas: PubMed, de la Biblioteca Nacional de Estados Unidos, y LILACS (Literatura Latinoamericana y del Caribe en Ciencias de la Salud), a fin de identificar los estudios a ser incluidos.
Para la búsqueda en PubMed, se utilizó la siguiente estrategia: «(polymorphism OR genotype OR variant OR mutation OR allele) AND Peru AND tuberculosis», palabras que fueron seleccionadas en base a revisiones sistemáticas previas sobre mutaciones (7,8). La búsqueda en LILACS empleó la siguiente estrategia: «mutaciones» y «tuberculosis» y «Peru».
En caso de múltiples publicaciones sobre el mismo estudio, se incluyó el estudio con la muestra más grande o el primero publicado.
SELECCIÓN DE ESTUDIOS Y RECOLECCIÓN DE DATOS
AV y LS seleccionaron de manera independiente los estudios a ser incluidos. Posteriormente, se revisó la lista de estudios incluidos y excluidos. En los que no hubo acuerdo, se discutió y llegó a un consenso. Asimismo, se revisaron las referencias y artículos relacionados a las publicaciones inicialmente identificadas.
Una vez definidos los estudios a incluir, se extrajo la información acerca de las características de la publicación (autor, revista, año), del estudio (tipo de diseño, características de los pacientes) y de las cepas (fármacos evaluadas, genes evaluados y mutaciones presentes) hacia tablas previamente diseñadas para el recojo de datos. Finalmente, se obtuvieron «tablas resumen» con la información recolectada.
No se realizó una evaluación formal de la calidad de las publicaciones, puesto que, al tratarse muchas veces de reportes puntuales de mutaciones, y al existir heterogeneidad significativa en la literatura médica para el reporte de este tipo de publicaciones (las iniciativas para homogenizar los reportes de las publicaciones de mutaciones en M. tuberculosis recién están surgiendo) se optó por no realizar evaluación de riesgo de sesgo en los estudios.
El principal aspecto a reportar fueron las secuencias nucleotídicas de las mutaciones puntuales que se encontraron asociadas con la resistencia a fármacos antituberculosis de primera línea, particularmente isoniazida y rifampicina. Finalmente, una vez extraída la información sobre las mutaciones encontradas, se graduaron en mínima, moderada y alta confianza de asociación a resistencia de acuerdo a la guía técnica sobre el «Uso de tecnologías de secuenciación de próxima generación para la detección de mutaciones asociadas con la resistencia farmacológica en el complejo de M. tuberculosis» de la OMS (9). Los resultados se reportan de acuerdo al fármaco antituberculosis analizado.
Cabe mencionar que no se procedió al registro del protocolo de esta revisión y por tratarse de un tema puramente descriptivo no se estructuró la pregunta PICO respectiva.
RESULTADOS
Se obtuvieron 70 artículos en PubMed y dos artículos en LILACS, de los cuales uno fue duplicado. Adicionalmente, se obtuvieron siete artículos más a partir de las referencias de los artículos seleccionados. Se excluyeron 64 artículos, y finalmente, 14 estudios cumplieron nuestros criterios de inclusión/exclusión, los cuales fueron admitidos para revisión. La Figura 1 muestra el flujograma de selección de los estudios incluidos. Los estudios de Moradigaravan (10) y Barletta (11) fueron excluidos por abordar fármacos de segunda línea (ácido para-amino salicílico, y quinolonas y aminoglicósidos, respectivamente).
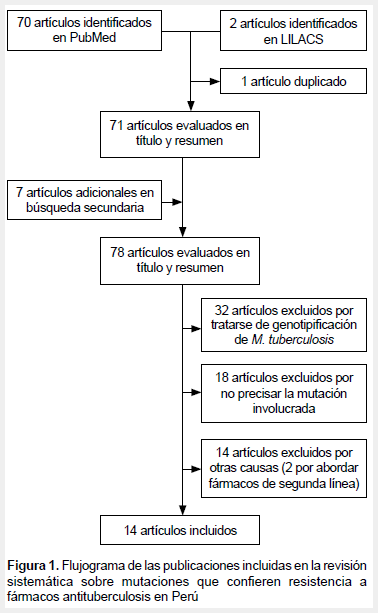
Las características principales de estos estudios se muestran en la Tabla 1. De los 14 artículos, 13 publicados en idioma inglés. Nueve artículos se enfocaron en la evaluación de mutaciones que confirieron resistencia a un solo fármaco antituberculosis y siete evaluaron más de un fármaco a la vez. Las características de tipo de población y especímenes también se describen en la Tabla 1.
Los fármacos antituberculosis de primera línea (isoniazida y rifampicina) se abordan en las Tabla 2 y 3, respectivamente. Además, la información referente a pirazinamida se encuentra presente en el material suplementario. Los estudios que evaluaron varios fármacos en paralelo fueron incluidos en todas las tablas que les correspondían.
De los tres estudios que reportaron resistencia a isoniazida, dos evaluaron los genes inhA y katG, y uno evaluó los genes katG y kasA.
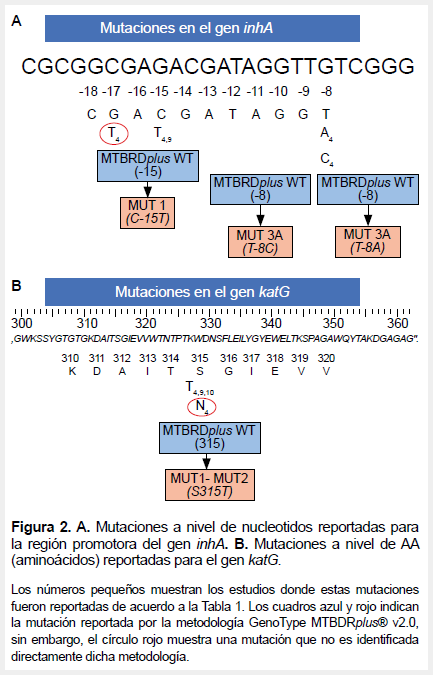
De las siete mutaciones reportadas en estos estudios, dos tuvieron una probabilidad de asociación a resistencia alta y una tuvo probabilidad moderada de asociación a resistencia (26). La mutación más frecuente se notificó en el codón 315 del gen katG. La ubicación de estas mutaciones en los genes inhA y katG, así como la región génica evaluada por la prueba comercial de diagnóstico de resistencia (GenoType MTBDRplus® v2.0) se muestra en la Figura 2.
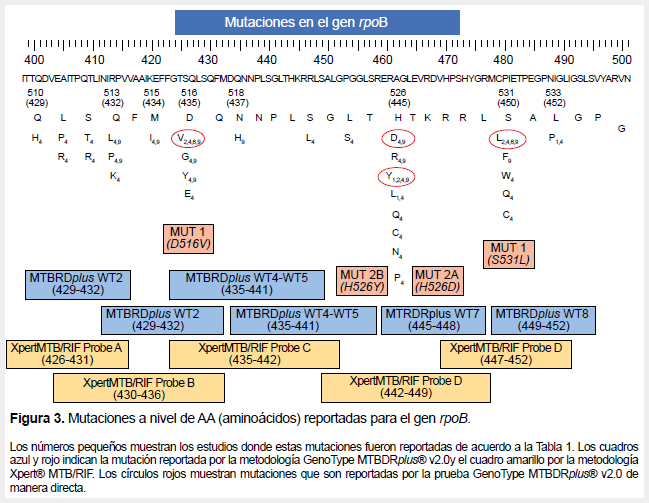
De los seis estudios que reportaron mutaciones asociadas con resistencia a rifampicina, todos evidenciaron mutaciones en el gen rpoB y se pudo observar que el estudio de Linger (15) fue el que identificó más mutaciones. Los estudios de Maha, Gygli y Galarza (12, 13, 20) presentaron en su mayoría mutaciones con alta probabilidad de asociación a resistencia. Las dos mutaciones más frecuentes, D516V y H526Y, tuvieron una alta asociación a resistencia (Tabla 3, Figura 3).
Con respecto a las mutaciones en el gen pncA asociadas con resistencia a pirazinamida, se pudo identificar ocho estudios que reportaron una alta cantidad de mutaciones. Además, se pudo apreciar que las mutaciones encontradas presentaron una alta asociación con resistencia a este fármaco. La mutación encontrada con mayor frecuencia fue la H51R, la cual se encuentra altamente asociada a resistencia. En el material suplementario se reportan las mutaciones de resistencia a pirazinamida que ya están clasificadas por la OMS y las que están pendiente de clasificación.
Con respecto a los otros fármacos antituberculosis de primera línea, se observó un estudio (12), en el que se evaluó el gen rpsL para la resistencia a estreptomicina. Este estudio utilizó Secuenciamiento de Genoma Completo y como método fenotípico el 7H10 y el MGIT BACTEC 960, encontrando la mutación K43R, la cual se encuentra fuertemente relacionada a resistencia. Asimismo, Galarza también utilizó Secuenciamiento de Genoma Completo para evaluar resistencia a etambutol a través del gen embB, encontrando la mutación Y319S, que no genera resistencia (21).
DISCUSIÓN
La presente revisión sistemática permitió identificar 14 estudios que reportan mutaciones en el genoma de M. tuberculosis, en genes vinculados con la resistencia a fármacos antituberculosis de primera línea. En total, se reportaron siete mutaciones en relación a resistencia a isoniazida (dos de ellas con alta y una con moderada confianza de asociación a resistencia). Para rifampicina, de las 31 mutaciones encontradas 21 ya han sido confirmadas como asociadas a resistencia (15 de ellas con alta, cuatro con moderada y dos con mínima confianza de asociación a resistencia). Adicionalmente, sólo se encontró una mutación asociada a etambutol, una mutación asociada a estreptomicina, y 48 a pirazinamida, fármaco en el cual ha habido una cantidad significativa de estudios de investigación. De manera adicional a los objetivos del estudio, pudimos identificar tres mutaciones asociadas con resistencia a aminoglicósidos y cinco con resistencia a quinolonas.
Esto denota un esfuerzo importante de los grupos de investigación en tuberculosis presentes en nuestro país, muchos de los cuales han sido muy activos durante las últimas décadas, produciendo investigaciones para profundizar en el conocimiento de la epidemiologia, clínica y microbiología de la tuberculosis en el Perú. En los últimos años, la rama que más ha cobrado vigencia es la de la biología molecular, que sirve para establecer las bases no solo para nuevas metodologías diagnósticas a través de la detección del material genético de la micobacteria, sino también para el desarrollo de nuevos fármacos (26).
Es importante precisar que, como parte del control de la fármaco-resistencia en tuberculosis, la prioridad debe ser la identificación de la resistencia a los dos pilares del tratamiento: isoniazida y rifampicina, por lo cual estos fármacos fueron la prioridad del estudio. Para la detección de la resistencia a la rifampicina, las pruebas comerciales disponibles y recomendadas son: el ensayo de sonda en línea GenoType MTBDRplus® v2.0 y Xpert® MTB/RIF. Ambas pruebas detectan mutaciones en la «Región Determinante de Resistencia a Rifampicina» (RDRR), mediante sondas de ADN que se unen de manera específica a dicha región. En un estudio comparativo, realizado en el 2014 entre GenoType MTBDRplus® v2.0 y Xpert® MTB/RIF, se determinó la superioridad del primero para la detección de M. tuberculosis monorresistente a rifampicina en términos de sensibilidad (27). Asimismo, la detección de la resistencia a isoniazida sólo puede realizarse mediante la prueba GenoType MTBDRplus® v2.0 tomando en consideración dos genes asociados con la resistencia como son: el gen katG y la región promotora del gen inhA. Mientras que la prueba Xpert® MTB/RIF no detecta esta resistencia.
A nivel del gen katG, el codón 315 fue la única posición que evidenció la aparición de dos mutaciones altamente asociadas con la resistencia a isoniazida: S315T Y S315N (9,15). Al evaluar las mutaciones detectadas por GenoType MTBDRplus® v2.0 (Figura 2) podemos observar que sólo la mutación S315T es identificada de manera directa, mientras que la mutación S315N (15) es identificada de manera indirecta. Del mismo modo, al contrastar la información recopilada sobre las mutaciones encontradas en el gen inhA con la prueba GenoType MTBDRplus® v2.0, la mutación presente en la posición nucleotídica g-17t es identificada de manera indirecta. Las mutaciones S315N y g-17t de los genes katG e inhA, respectivamente, no son detectadas directamente por la prueba GenoType MTBDRplus® v2.0, pero sí indirectamente mediante la ausencia de alguna banda de tipo salvaje (wild type). Por otro lado, la mutación G269S del gen kasA no es analizada por dicha prueba molecular.
Respecto a las mutaciones reportadas en el gen rpoB, en general, todas pueden ser detectadas mediante las pruebas moleculares GenoType MTBDRplus® v2.0 y Xpert® MTB/RIF. Además de las mutaciones clásicamente asociadas a resistencia, se identificaron las mutaciones L511R, D516E, N518H, Q510H, S512R, L524S, H526Q y S531C (15, 20), las cuales no son reportadas en la guía técnica de la OMS (9) . Estas mutaciones D516E, D516G, D516Y, del507, H526C, H526L, H526N, H526P, H526Q, H526R, L511P, L511R, L524S, L533P, M515I, N516H, Q510H, Q513K, Q513L, Q513P, S512T, S512R, S522L, S531C, S531F, S531Q, S531W no se encuentran definidas en las bandas de mutaciones del GenoType MTBDRplus® v2.0; sin embargo, son detectadas indirectamente por esta metodología, debido a la ausencia de las bandas wild type que abarcan los codones respectivos.
A pesar de que algunas mutaciones no son determinadas directamente a través de la prueba GenoType MTBDRplus® v2.0, tanto para isoniazida como para rifampicina, éstas serán reportadas como resistencia inferida, debido a la ausencia de bandas wild type. Algo similar ocurre con la prueba Xpert® MTB/RIF, ya que trabaja con sondas salvajes y no identifica mutaciones específicas (28). Las sondas de hibridación A, B, C, D y E de la prueba Xpert® MTB/RIF identifican de manera indirecta todas las mutaciones a rifampicina reportadas en esta revisión sistemática (29,30).
Otro hallazgo importante es la gran cantidad de mutaciones que confieren resistencia a pirazinamida (Anexo 3). La detección microbiológica de resistencia a este fármaco siempre ha sido un reto (26), y hasta el momento el método más utilizado es el Wayne (31). El hecho que exista una gran cantidad de mutaciones asociadas con resistencia a pirazinamida dificulta el desarrollo de métodos moleculares de detección de resistencia.
En el 2018, el uso del secuenciamiento de genoma completo para la detección de mutaciones asociadas a resistencia en M. tuberculosis fue recomendada por la OMS para su implementación en laboratorios de referencia nacional (9) . Debido a esto, el uso del secuenciamiento del genoma completo de M. tuberculosis, en condiciones habituales o de rutina, aportará información más completa acerca de la significancia de las mutaciones para la identificación de especies micobacterianas, detección de la resistencia a fármacos antituberculosis y comprensión de la dinámica de transmisión de los microrganismos (32, 33).
Como parte de las fortalezas de nuestro estudio, se debe tomar en cuenta que son pocas las revisiones sistemáticas sobre temas de biología molecular, y específicamente sobre resistencia a fármacos antituberculosis. Han sido publicados estudios previos como el de Vásquez-Loarte (34); pero más enfocados en la asociación de polimorfismos para evaluar el origen filogenético de M. tuberculosis. Sin embargo, nuestro estudio aborda específicamente el tema de las mutaciones presentes en las cepas que circulan en nuestro país. Esto puede resultar más útil desde el punto de vista práctico, dado que esta información reúne datos sobre las mutaciones que se encuentran presentes en las cepas peruanas a la fecha. Por otro lado, puede servir de base para el desarrollo de pruebas de detección de resistencia a fármacos antituberculosis en nuestro país, considerando tanto las mutaciones de alta prevalencia a nivel mundial, así como las que aún no están incluidas en los dispositivos diagnósticos comerciales.
Asimismo, debemos puntualizar algunos aspectos del estudio que limitan su ámbito de aplicación. En primer lugar, debido a que no todas las publicaciones registraron las frecuencias de las mutaciones, no fue posible realizar una aproximación de las mutaciones que son más prevalentes en nuestro territorio. Sólo podemos mencionar que están presentes. Por otro lado, a pesar de nuestro esfuerzo por verificar, de acuerdo a las recomendaciones de la OMS, si las mutaciones conferían mínima, moderada o alta confianza de asociación a resistencia, o no estaban asociadas a resistencia, hubo numerosas mutaciones que no pudieron ser clasificadas. Por lo tanto, las implicancias clínicas de estas últimas son inciertas por ahora, hasta que existan más estudios y se genere más evidencia sobre su impacto.
En conclusión, existe una gran variedad de mutaciones asociadas con la resistencia a fármacos antituberculosis de primeralínea en pacientes peruanos. Para los fármacos isoniazida y rifampicina, pilares del tratamiento antituberculosis, la cantidad de mutaciones asociadas a resistencia es menor en comparación a pirazinamida. Un aspecto positivo es que la gran mayoría de mutaciones tanto para isoniazida como para rifampicina son identificadas indirectamente (por ausencia de hibridación de sondas wild type) en las pruebas GenoType MTBDRplus® v2.0 y Xpert® MTB/RIF. Estas consideraciones deben de ser tomadas en cuenta para el desarrollo de dispositivos diagnósticos a ser aplicados localmente en nuestro país.
Contribuciones de los autores: AV participó en la concepción del estudio. LS participó en el diseño del estudio. AV, LS, DS y ZP participaron en el análisis e interpretación de los datos, la redacción del manuscrito y aprobaron la versión final. Todos los autores declaran tener responsabilidad sobre el estudio.
Fuentes de financiamiento: Este estudio fue financiado por INNOVATE - PERU en el marco del contrato - N°353-PNICP-PIAP-2014.
Conflictos de interés: Los autores declaran no tener conflictos de interés.
Material suplementario: Disponible en la versión electrónica de la RPMESP
REFERENCIAS BIBLIOGRÁFICAS
1. World Health Organization. WHO | Global tuberculosis report 2019 [Internet]. WHO. 2019 [citado el 31 de octubre de 2019]. Disponible en: http://www.who.int/tb/publications/global_report/en/
2. Ministerio de Salud. Direccion de Prevencion y Control de la Tuberculosis [Internet]. [citado el 29 de octubre de 2019]. Disponible en: http://www.tuberculosis.minsa.gob.pe/DashboardDPCTB/Dashboard.aspx
3. Loddenkemper R, Sagebiel D, Brendel A. Strategies against multidrug-resistant tuberculosis. European Respiratory Journal. 2002;20(36 suppl):66s-77s.
4. Dahle UR, Sandven P, Heldal E, Mannsaaker T, Caugant DA. Deciphering an Outbreak of Drug-Resistant Mycobacterium tuberculosis. J Clin Microbiol. 2003;41(1):67-72. doi: 10.1128/JCM.41.1.67-72.2003.
5. Boldú J, Cebollero P, Abu J, De Prado A. Tratamiento de la tuberculosis pulmonar. Anales del Sistema Sanitario de Navarra. 2008;30(0):87-98.
6. Moher D, Liberati A, Tetzlaff J, Altman DG, PRISMA Group. Preferred reporting items for systematic reviews and meta-analyses: the PRISMA statement. Ann Intern Med. 2009;151(4):264-9, W64. doi: 10.7326/0003-4819-151-4-200908180-00135.
7. Ramirez-Busby SM, Valafar F. Systematic review of mutations in pyrazinamidase associated with pyrazinamide resistance in Mycobacterium tuberculosis clinical isolates. Antimicrob Agents Chemother. 2015;59(9):5267-77. doi: 10.1128/AAC.00204-15.
8. Whitfield MG, Soeters HM, Warren RM, York T, Sampson SL, Streicher EM, et al. A Global Perspective on Pyrazinamide Resistance: Systematic Review and Meta-Analysis. PLoS One. 2015;10(7):e0133869. doi: 10.1371/journal.pone.0133869.
9. World Health Organization. The use of next-generation sequencing technologies for the detection of mutations associated with drug resistance in Mycobacterium tuberculosis complex: technical guide [Internet]. 2018 [citado el 29 de octubre de 2019]. Disponible en: https://apps.who.int/iris/handle/10665/274443.
10. Moradigaravand D, Grandjean L, Martinez E, Li H, Zheng J, Coronel J, et al. DFRA thyA Double Deletion in para-Aminosalicylic Acid-Resistant Mycobacterium tuberculosis Beijing Strains. Antimicrob Agents Chemother. 2016;60(6):3864-7. doi: 10.1128/AAC.00253-16.
11. Barletta F, Zamudio C, Rigouts L, Seas C. Resistance to second-line anti-tuberculosis drugs among peruvian multidrug resistant Mycobacterium tuberculosis strains. Rev Peru Med Exp Salud Publica. 2014;31(4):676-82.
12. Gygli SM, Keller PM, Ballif M, Blöchliger N, Hömke R, Reinhard M, et al. Whole-Genome Sequencing for Drug Resistance Profile Prediction in Mycobacterium tuberculosis. Antimicrob Agents Chemother. 2019;63(4):e02175-18. doi: 10.1128/AAC.02175-18
13. Farhat MR, Sixsmith J, Calderon R, Hicks ND, Fortune SM, Murray M. Rifampicin and rifabutin resistance in 1003 Mycobacterium tuberculosis clinical isolates. J Antimicrob Chemother. 2019;74(6):1477-83. doi: https://doi.org/10.1093/jac/dkz048.
14. Jaramillo L, Tarazona D, Levano KS, Galarza M, Caceres O, Becker M, et al. A rapid identification technique for drug-resistant Mycobacterium tuberculosis isolates using mismatch specific cleavage enzyme. Bioinformation. 2018;14(7):404-7. doi: 10.6026/97320630014404.
15. Linger Y, Knickerbocker C, Sipes D, Golova J, Franke M, Calderon R, et al. Genotyping Multidrug-Resistant Mycobacterium tuberculosis from Primary Sputum and Decontaminated Sediment with an Integrated Microfluidic Amplification Microarray Test. J Clin Microbiol. 2018;56:e01652-17. doi: https://doi.org/10.1128/JCM.01652-17.
16. Rueda D, Bernard C, Capton E, Boudjelloul R, Brossier F, Veziris N, et al. Estimation of pyrazinamidase activity using a cell-free In vitro synthesis of pnca and its association with pyrazinamide susceptibility in Mycobacterium tuberculosis. Int J Mycobacteriol. 2018;7(1):16-25. doi: 10.4103/ijmy. ijmy_187_17.
17. Tessema B, Nabeta P, Valli E, Albertini A, Collantes J, Lan NH, et al. FIND Tuberculosis Strain Bank: a Resource for Researchers and Developers Working on Tests To Detect Mycobacterium tuberculosis and Related Drug Resistance. J Clin Microbiol. 2017;55(4):1066-73. doi: 10.1128/ JCM.01662-16.
18. Sheen P, Requena D, Gushiken E, Gilman RH, Antiparra R, Lucero B, et al. A multiple genome analysis of Mycobacterium tuberculosis reveals specific novel genes and mutations associated with pyrazinamide resistance. BMC Genomics. 2017;18:769. doi: 10.1186/s12864-017-4146-z.
19. Dudley MZ, Sheen P, Gilman RH, Ticona E, Friedland JS, Kirwan DE, et al. Detecting Mutations in the Mycobacterium tuberculosis Pyrazinamidase Gene pncA to Improve Infection Control and Decrease Drug Resistance Rates in Human Immunodeficiency Virus Coinfection. Am J Trop Med Hyg. 2016;95(6):1239-1246. doi: 10.4269/ajtmh.15-0711.
20. Galarza M, Fasabi M, Levano KS, Castillo E, Barreda N, Rodriguez M, et al. High-resolution melting analysis for molecular detection of multidrug resistance tuberculosis in Peruvian isolates. BMC Infect Dis. 2016;16(1):260. doi: https://doi.org/10.1186/s12879-016-1615-y.
21. Galarza M, Tarazona D, Borda V, Agapito JC, Guio H. Evidence of Clonal Expansion in the Genome of a Multidrug-Resistant Mycobacterium tuberculosis Clinical Isolate from Peru. Genome Announc. 2014;2(1):e00089-14. doi: 10.1128/genomeA.00089-14.
22. Sheen P, Lozano K, Gilman RH, Valencia HJ, Loli S, Fuentes P, et al. pncA gene expression and prediction factors on pyrazinamide resistance in Mycobacterium tuberculosis. Tuberculosis (Edinb). 2013;93(5):515-22. doi: 10.1016/j.tube.2013.03.005.
23. Zimic M, Sheen P, Quiliano M, Gutierrez A, Gilman RH. Peruvian and globally reported amino acid substitutions on the Mycobacterium tuberculosis pyrazinamidase suggest a conserved pattern of mutations associated to pyrazinamide resistance. Infect Genet Evol. 2010;10(2):346-9. doi: 10.1016/j.meegid.2009.11.016.
24. Sheen P, Ferrer P, Gilman RH, López-Llano J, Fuentes P, Valencia E, et al. Effect of pyrazinamidase activity on pyrazinamide resistance in Mycobacterium tuberculosis. Tuberculosis (Edinb). 2009;89(2):109-13. doi: 10.1016/j.tube.2009.01.004.
25. Sheen P, Méndez M, Gilman RH, Peña L, Caviedes L, Zimic MJ, et al. Sputum PCR-Single-Strand Conformational Polymorphism Test for Same-Day Detection of Pyrazinamide Resistance in Tuberculosis Patients. J Clin Microbiol. 2009;47(9):2937-43. doi: 10.1128/JCM.01594-08.
26. Miotto P, Tessema B, Tagliani E, Chindelevitch L, Starks AM, Emerson C, et al. A standardised method for interpreting the association between mutations and phenotypic drug resistance in Mycobacterium tuberculosis. Eur Respir J. 2017;50(6):1701354. doi: 10.1183/13993003.01354-2017.
27. Rufai SB, Kumar P, Singh A, Prajapati S, Balooni V, Singh S. Comparison of Xpert MTB/RIF with line probe assay for detection of rifampinmonoresistant Mycobacterium tuberculosis. J Clin Microbiol. 2014;52(6):1846-52. doi: 10.1128/JCM.03005-13.
28. Hain Lifescience. GenoType MTBDRs/. Detection of resistance of MTBC complex [Internet]. 2015 [citado el 29 de octubre de 2019]. Disponible en: https://www.hain-lifescience.de/en/products/microbiology/mycobacteria/tuberculosis/genotype-mtbdrsl.html.
29. Organización Panamericana de la Salud/Organización Mundial de la Salud. Manual de Capacitación en GeneXpert [Internet]. OPS/OMS. 2017 [citado el 29 de octubre de 2019]. Disponible en: https://www.paho.org/hq/index.php?option=com_content&view=article&id=12924:manu-al-de-capacitacion-en-genexpert&Itemid=42250&lang=es.
30. Andre E, Goeminne L, Cabibbe A, Beckert P, Kabamba Mukadi B, Mathys V, et al. Consensus numbering system for the rifampicin resistanceassociated rpoB gene mutations in pathogenic mycobacteria. Clin Microbiol Infect. 2017;23(3):167-172. doi: 10.1016/j.cmi.2016.09.006.
31. Leo E, Vásquez L, Asencios L, Quispe N, Gómez L, Lecca L, et al. Determinación de la susceptibilidad de Mycobacterium tuberculosis a la pirazinamida mediante la prueba de la pirazinamidasa, Perú – 1999. Rev Peru Med Exp Salud Publica. 2003;20(2):105-6.
32. Outhred AC, Jelfs P, Suliman B, Hill-Cawthorne GA, Crawford ABH, Marais BJ, et al. Added value of whole-genome sequencing for management of highly drug-resistant TB. J Antimicrob Chemother. 2015;70(4):1198-202. doi: 10.1093/jac/dku508.
33. Quan TP, Bawa Z, Foster D, Walker T, del Ojo Elias C, Rathod P, et al. Evaluation of Whole-Genome Sequencing for Mycobacterial Species Identification and Drug Susceptibility Testing in a Clinical Setting: a Large-Scale Prospective Assessment of Performance against Line Probe Assays and Phenotyping. J Clin Microbiol. 2018;56(2):e01480-17. doi: 10.1128/JCM.01480-17.
34. Vásquez-Loarte T, Trubnykova M, Guio H. Genetic association meta-analysis: a new classification to assess ethnicity using the association of MCP1 -2518 polymorphism and tuberculosis susceptibility as a model. BMC Genet. 2015;16:128. doi: 10.1186/s12863-015-0280-2.
Correspondencia:Aiko Vigo Dirección: Defensores del Morro, Chorrillos, Lima, Perú.
Email:
aiko.vt.anvt@gmail.com
Teléfono: +51924167434
Recibido: 06/08/2019
Aprobado: 06/11/2019
En línea: 03/12/2019