María J. Pons
Lidia Ruiz-Roldán
Laura Laureano-Adame
Alfredo Corujo
Theresa J. Ochoa
Joaquim Ruiz
ORIGINAL BREVE
Resistencia a nitrofuranos en Salmonella
enterica aisladas de carne para consumo humano
Nitrofuran resistance in Salmonella enterica from human consumption meat samples
Sandra Martínez-Puchol
![]() 1, Magíster en Biología
1, Magíster en Biología
María J. Pons
![]() 2, PhD en Biología
2, PhD en Biología
Lidia Ruiz-Roldán ![]() 1, PhD en Biología
1, PhD en Biología
Laura Laureano-Adame ![]() 3, PhD en Biología
3, PhD en Biología
Alfredo
Corujo
![]() 3,
PhD en Biología
3,
PhD en Biología
Theresa J. Ochoa ![]() 4,5, medico infectólogo, magíster en
Medicina
4,5, medico infectólogo, magíster en
Medicina
Joaquim Ruiz ![]() 1,6, PhD en Biología
1,6, PhD en Biología
1 Instituto de Salud Global, Hospital Clinic - Universitat de
Barcelona, Barcelona, España.
2 Laboratorio de Microbiología Molecular y Genómica
Bacteriana, Universidad Científica del Sur, Lima, Perú.
3 Nutreco, Toledo, España.
4 Instituto de Medicina Tropical Alexander von
Humboldt, Universidad Peruana Cayetano Heredia, Lima, Perú.
5 Department of Epidemiology, School of Public Health, University of Texas Health Science Center at Houston,
Estados Unidos.
6 Universidad Continental, Lima, Perú.
RESUMEN
En el presente estudio, se
analizaron los mecanismos de resistencia a nitrofuranos
en 18 muestras cárnicas con Salmonella enterica
(15 de pollo, 2 de ternera y 1 de cerdo) de mercados de Lima (Perú).
Determinaron los serotipos de los aislamientos y la sensibilidad a furazolidona y nitrofurantoina
(con y sin el inhibidor de bombas de expulsión Phenyl-Arginine-β-Naphthylamide [PAβN]), las mutaciones en los genes snrA y cnr por PCR
y la transferabilidad de la resistencia por
conjugación. Se identificaron 15 muestras con S. infantis
(13 muestras de pollo), 2 con S. enteritidis y
1 con S. anatum. Todos los aislamientos,
excepto S. anatum, fueron resistentes a ambos nitrofuranos (concentración mínima inhibidora [CMI] a furazolidona: 32-64 µg/mL, CMI a nitrofurantoina: 128-256 µg/mL),
sin diferencias al adicionarse PAβN. Todos los aislamientos resistentes a nitrofuranos presentaron sustituciones en snrA y cnr (S. infantis: snrA STOP-151; cnr STOP-137; S. enteritidis:
snrA STOP-180; cnr
STOP-179). No se detectaron mecanismos transferibles de resistencia a nitrofuranos.
Palabras clave: Resistencia a Antibióticos; Furazolidona;
Salmonella (fuente: DeCS BIREME).
ABSTRACT
The mechanisms of resistance to nitrofurans from 18 meat samples
with Salmonella enterica
(chicken: 15; beef: 2; pork: 1) collected in Lima (Peru) were analyzed.
The isolates were serotyped and the susceptibility levels to furazolidone and nitrofurantoin [with and without the efflux
pump inhibitor Phenyl-Arginine- β-naphthylamide
(PAβN)], the presence of mutations in the snrA and cnr
genes and the transferability
of resistance by conjugation were established. Fifteen samples with S. infantis (13 from chicken samples), 2 with S. enteritidis and 1 with S. anatum were identified. All isolates except
the S. anatum were resistant to both nitrofurans showing MICs (minimum
inhibitory concentration)
of furazolidone and nitrofurantoin
of 32-64 μg/mL and 128-256 μg/mL, respectively.
The addition of PAßN had no effect
on the MIC levels. All nitrofuran-resistant
isolates showed amino acid codon alterations
at both snrA and cnr (S. infantis: snrA STOP-151; cnr
STOP-137; S. enteritidis: snrA
STOP-180; cnr STOP-179). No transferable
mechanisms of nitrofuran resistance were detected.
Keywords: Drug resistance; Furazolidone; Salmonella (source: MeSH NLM).
INTRODUCCIÓN
Los nitrofuranos
son un grupo de antimicrobianos de origen sintético activos frente a parásitos
y bacterias Gram-negativas y Gram-positivas (1). Estos compuestos han sido
ampliamente utilizados tanto en medicina humana como veterinaria, así como en
promotores de crecimiento de animales destinados al consumo humano (1).
Actualmente, su uso en veterinaria para animales de consumo humano está prohibido
en numerosos países debido al riesgo a que estos antimicrobianos o sus
metabolitos permanezcan en el alimento (1). Su uso veterinario está
prohibido en Perú desde 2013 (2).
En líneas generales, los
niveles de resistencia a nitrofurano en Enterobacteriaceae causantes de diarrea, como
por ejemplo Shigella spp.,
son bajos (3),
habiéndose reportado los mayores niveles de resistencia a estos antimicrobianos
en Salmonella spp. (4,5).
En la actualidad, los mecanismos de acción de los nitrofuranos
están poco estudiados, habiéndose descrito principalmente en mutantes obtenidos
in vitro.
Se ha descrito que los nitrofuranos precisan ser activados por nitroreductasas
para ejercer su acción; la reacción de reducción constaría de varios pasos
secuenciales, considerando que algunos de los productos generados en estos
pasos intermedios podrían poseer actividad antimicrobiana (6,7).
Las nitroreductasas implicadas en estos procesos de
activación son NfsA y NfsB (6).
Así, en Escherichia coli,
la presencia de alteraciones capaces de reducir o eliminar la funcionalidad de
estas enzimas se ha asociado con el desarrollo de resistencia a nitrofuranos, tanto en estudios con mutantes obtenidos in
vitro como en estudios desarrollados con aislamientos clínicos (8-10).
También se ha descrito el rol de bombas de expulsión en la resistencia a nitrofuranos. Además, se ha constatado que la bomba de
expulsión OqxAB, perteneciente a la familia RND (del
inglés Resistance-Nodulation-Division), es capaz de
conferir resistencia a nitrofurantoina (11).
Pese a los antecitados
mayores niveles de resistencia presentes en Salmonella spp., el número de estudios realizados enfocados al estudio
de los mecanismos de resistencia a nitrofuranos es
escaso (5,12).
Por lo tanto, el objetivo del presente estudio fue determinar los niveles y
mecanismos de resistencia a nitrofuranos en
aislamientos de Salmonella enterica
procedentes de muestras cárnicas adquiridas en mercados tradicionales de Lima.
|
MENSAJES CLAVE |
|
Motivación para realizar el estudio: Los crecientes niveles de resistencia a
antimicrobianos en Perú constituyen una sería preocupación. No obstante, hay
una escasez de datos respecto a la resistencia a nitrofuranos
en Salmonella spp. Principales hallazgos: Existen altos niveles de resistencia a nitrofuranos en Salmonella spp.
correlacionados con la presencia de mutaciones cromosomales en los genes cnr
y snrA. Implicancias: Los resultados del estudio muestran la necesidad
de investigaciones sistemáticas de los niveles de resistencia a nitrofuranos en Salmonella spp.
dirigidas no solo a niveles y mecanismos, sino
también a las causas de la selección natural de aislamientos resistentes. |
EL ESTUDIO
Se utilizaron 18 muestras con Salmonella
enterica aisladas en 2012 de un estudio previo,
que tuvo como objetivo determinar la presencia de Enterobacteriaceae
y los niveles de resistencia a antimicrobianos de E. coli
en muestras cárnicas (cerdo, pollo o ternera), adquiridas en mercados
tradicionales del norte (Comas, San Martín), centro (La Victoria, Cercado de
Lima) y sur (Villa El Salvador) de Lima (13). El estudio se realizó en el
ISGlobal, Hospital Clinic -
Universitat de Barcelona (España), Nutreco (España) y el Instituto de Medicina Tropical
Alexander von Humboldt (Perú). En todos los casos los aislamientos fueron
identificados previamente por métodos bioquímicos y confirmados mediante
amplificación del gen invA (13).
Los aislamientos de S. enterica se recuperaron
de -80 ºC y de manera previa a su uso se
reconfirmaron mediante la amplificación y secuenciación del gen 16S rRNA (14).
Se determinó los serotipos de
los aislamientos mediante microarrays (Check & Trace Salmonella kit, Check-Points B.V, Wageningen,
Holanda) siguiendo las instrucciones del fabricante. Tras ello se procedió a
determinar la sensibilidad a furazolidona (100 µg) y
nitrofurantoina (300 µg) mediante el método de
difusión en disco (BD, San Agustín del Guadalix,
España), así como los valores de concentración mínima inhibitoria (CMI) mediante
el método de dilución en agar, siguiendo las directrices de la guía CLSI (Clinical and Laboratory Standards Institute) y utilizando
la cepa E. coli ATCC 25922 como control de
calidad (15).
En la guía CLSI no existe un punto de corte específico para furazolidona,
por lo que en este caso se reportan los datos de CMI y/o diámetro de halo.
Tanto para nitrofurantoina
como para furazolidona, los valores de CMI se
determinaron en presencia de Phenyl-Arginine-ß-Naphthylamide
(PAßN, por sus siglas en inglés), un inhibidor de
bombas de expulsión de tipo RND (8). Para su uso, el PAßN
se disolvió en dimetil sulfoxido
(DMSO), motivo por lo que determinó el efecto de este solvente en el
crecimiento bacteriano.
En todos los aislamientos, se
amplificaron los genes nfsA y nfsB mediante la técnica de reacción en cadena de la
polimerasa (PCR, por sus siglas en inglés) (ciclo inicial de 94 ºC por cinco minutos, seguido por 35 ciclos de 94 ºC por 40 segundos cada uno, 60 ºC
por 30 segundos, 68 ºC por 40 segundos, y una extensión
final de 72 ºC por cinco minutos). La PCR se realizó
con los cebadores descritos por Salamanca-Pinzón et al. (16),
visualizándose en geles de agarosa al 2% teñidos con SYBR Safe (Invitrogen, Carlsbad, EE. UU.). Las bandas
obtenidas fueron recuperadas del gel y se purificaron utilizando el kit Wizard SV Gel and PCR Clean Up System (Promega, Madison, EE.
UU.). Los productos purificados fueron enviados a Macrogen
(Seul, Corea del Sur) para su secuenciación.
Por último, la presencia de
mecanismos transferibles de resistencia a nitrofuranos
se determinó por conjugación siguiendo el protocolo previamente descrito (17).
Para este fin se utilizó como cepa receptora a E. coli
J53 (resistente a azida sódica) y agar Mueller-Hinton suplementado con azida
sódica (150 µg/ml) y furazolidona (16 µg/ml) como
medio para seleccionar los transconjugantes.
HALLAZGOS
Los 18 aislamientos que se
incluyeron en el presente estudio provenían de cuatro mercados diferentes de
las tres áreas del estudio original (cono norte, centro y sur) (Tabla 1), lo
que demostró, por tanto, una amplía diseminación por toda el área de Lima.
Tabla
1.
Origen de las muestras de carne
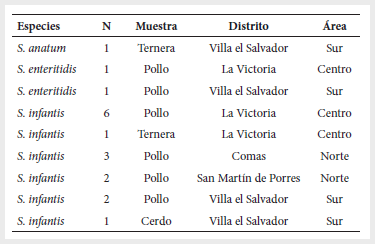
N: Número de aislamientos de S. enterica. Solo se consideró un aislamiento de Salmonella
por muestra
Los resultados del serotipado mostraron que la mayoría de los aislamientos
pertenecían al serotipo infantis (15 aislamientos,
83,3%). Las tres cepas restantes se clasificaron como enteritidis
(dos cepas, 11,1%) y anatum (una cepa, 5,6%).
Los aislamientos del serotipo infantis se
recuperaron de los tres tipos de muestras cárnicas, en especial de las muestras
de pollo. Así 13 de los 15 aislamientos recuperados procedían de muestras de
pollo, mientras que se aisló una de S. infantis
en muestras de ternera, y otra en muestras de cerdo. Por su parte, los dos
aislamientos de S. enteritidis provinieron de
muestras de pollo y el de S. anatum de ternera
(Tabla 1).
Todos los aislamientos, a
excepción de la cepa de S. anatum, presentaron
niveles de CMI de 32-64 µg/ml para furazolidona,
siendo resistentes a nitrofurantoina con CMIs de 128-256 µg/ml. El aislamiento de S. anatum presentó CMIs de 8
µg/ml para furazolidona y de 32 µg/ml para nitrofurantoina (Tabla 2).
Tabla
2.
Niveles y mecanismos de resistencia a nitrofuranos en
Salmonella spp.
n: número de
aislamientos; CMI: concentración mínima inhibitoria; wt:
ausencia de mutaciones.
a Calculado solo para
S. infantis.
Asimismo, se observó
correlación entre los valores de CMI y los halos observados en los estudios con
discos de antibiótico. Así, en el caso de los aislamientos resistentes a nitrofurantoina se observaron halos de 8- 11 mm de diámetro
para este antibiótico y de 8-13 mm para furazolidona,
mientras que el aislamiento sensible presentó halos de 20 mm a nitrofurantoina y 24 mm a furazolidona.
La adición de PAßN no afectó los valores de CMI, que en todos los casos
permanecieron inalterados, lo cual mostró la no implicación de bombas de
expulsión tipo RND en el desarrollo de resistencia a nitrofuranos
en los aislamientos estudiados. Ni PAßN ni DMSO
interfirieron con el crecimiento normal de las bacterias.
Todos los aislamientos
resistentes a nitrofuranos presentaron mutaciones en snrA y cnr. Los 15
aislamientos de S. infantis presentaron sendos
codones STOP en la posición 151 de snrA y
137 de cnr, mientras que las dos S. enteritidis los presentaron en las posiciones 180 de snrA y 179 de cnr
(Tabla 2). Por último, no se obtuvieron transconjugantes
resistentes a nitrofuranos en los estudios de
conjugación.
DISCUSIÓN
A pesar de que el uso de nitrofuranos en animales de consumo está prohibido en
numerosos países, la resistencia a estos mismos se ha descrito en enteropatógenos, como es el caso de Salmonella spp., aisladas de muestras alimentarias (4,5);
incluso, en algunos casos se han detectado restos de nitrofuranos
en productos cárnicos (18). Hay varias explicaciones posibles a
estos hechos, las cuales incluyen la estabilidad de la resistencia a nitrofuranos, el uso de estos antimicrobianos, pese a
tratarse de productos prohibidos, o la existencia de contaminación ambiental (4,8,18).
El hecho que los enteropatógenos aislados tuviesen como origen muestras de
los tres tipos de carnes (pollo, ternera y cerdo), incluidos en el estudio y
su presencia en las diferentes zonas de Lima, sugiere la amplia diseminación
geográfica en el país de S. enterica
resistentes a nitrofuranos. No obstante, se ha de
considerar que las muestras procesadas se colectaron en 2012, un año antes de
la prohibición en Perú del uso de nitrofuranos en la
cría de animales de consumo (2).
Se ha observado que la
adquisición de resistencia a nitrofuranos es un hecho
secuencial, en el que las nitrorreductasas NfsA y NfsB acumulan alteraciones
que afectan su funcionalidad y que poseen un efecto aditivo en los niveles
finales de resistencia a nitrofuranos (9). Los
resultados del presente estudio fueron concordantes, detectándose la presencia
de mutaciones conducentes a la presencia de codones STOP en los genes
equivalentes (snrA y cnr) y a la subsiguiente falta de nitroreductasas
funcionales. Hasta la fecha muy pocos estudios han analizado los mecanismos de
resistencia a nitrofuranos en S. enterica; en ellos se han encontrado escenarios
similares, con presencia de codones STOP u otras alteraciones en los genes snrA o cnr (5,12).
Si bien en el presente estudio
no se obtuvieron transconjugantes resistentes a nitrofuranos, se han descrito mecanismos transferibles de
resistencia a nitrofuranos. Entre estos, merece una
especial atención la bomba de expulsión OqxAB, la
cual ha sido recientemente implicada en el desarrollo de resistencia a nitrofurantoina (11). OqxAB
es una bomba de expulsión de tipo RND, indígena de Klebsiella
spp., la cual fue detectada y codificada por primera
vez en plásmido en aislamientos de E. coli de
origen veterinario en un estudio sobre la resistencia a olaquindox
(19).
En Perú, el uso de olaquindox en animales de consumo
se prohibió a la par que el de nitrofuranos (2). En
la actualidad, se conocen al menos 14 alelos de oqxA
y 28 de oqxB codificados en plásmidos, sin que
la actividad específica de cada uno de ellos haya sido establecida (19).
La presencia de mutaciones en acrB, emrD, yajR o macB,
genes codificantes de bombas de expulsión cromosomales,
ha sido implicada en el desarrollo de resistencia a nitrofuranos
(20).
La más estudiada de estas bombas es AcrAB que, al
igual que OqxAB, es una bomba de expulsión de tipo
RND. La bomba AcrAB, al igual que otras bombas de
tipo RND incluyendo a OqxAB, puede ser inhibida
usando substancias como PAßN (19,20).
En el presente estudio no se observó efecto de PAßN,
por lo que se descartó que la sobreexpresión de bombas tipo AcrAB
estuviese implicada en el desarrollo de resistencia a nitrofuranos
en estos aislamientos, aportando además una evidencia complementaria sobre la
ausencia de bombas plasmídicas OqxAB.
En un estudio previo en el que se desarrollaron mutantes de E. coli resistentes a furazolidona,
tampoco se detectó implicancia de bombas de expulsión inhibibles
por PAßN (8).
Debido a la metodología
utilizada la presencia de mecanismos de resistencia transferibles que solo
confiriesen modestos incrementos en los niveles de resistencia, podría haber
pasado desapercibida, lo que constituye una limitación del presente estudio.
Asimismo, los aislamientos incluidos en el estudio son del 2012, lo que resalta
la necesidad de efectuar nuevos estudios que permitan valorar la situación
actual.
El presente estudio describe
la presencia de niveles altos de resistencia a nitrofuranos
en aislamientos de Salmonella enterica procedentes
de muestras de alimentos cárnicos comercializados en el área de Lima. Estos
niveles de resistencia se relacionaron de manera directa con la presencia de
mutaciones cromosomales en los genes snrA y cnr. Es
preciso mantener un seguimiento de los niveles de resistencia a nitrofuranos en S. enterica.
REFERENCIAS BIBLIOGRÁFICAS
1. Vass
M, Hruska K, Franek M. Nitrofuran antibiotics: a review on the
application, prohibition
and residual analysis. Vet Med (Praha). 2008;53(9):469-500. doi:
10.17221/1979-VETMED.
2. Carrasco Valiente JA.
Prohíben importación y comercialización de diversos principios activos, así
como el uso de los mismos en la fabricación de productos veterinarios o
alimentos para animales destinados al consumo humano y establecen otras
disposiciones. Resolución Directoral Nº 0072-2013-MINAGRI-SENASA-DIAIA
[Internet]. Diario Oficial El Peruano; 2013 (citado el 10 de agosto de 2019):
503464-5. Disponible en: https://www.minagri.gob.pe/portal/download/pdf/marcolegal/normaslegales/resolucionesdirectorales/2013/setiembre/rd72-2013-minagri-senasa-diaia.pdf.
3. Pons MJ, Gomes C, Martínez-Puchol S, Ruiz L, Mensa L, Vila J, et al. Antimicrobial resistance in Shigella spp. causing traveller’s
diarrhoea (1995-2010): a retrospective
analysis. Travel Med Infect Dis.
2013;11(5):315-9. doi: 10.1016/j.tmaid.2013.06.010.
4. Antunes
P, Machado J, Peixe L. Illegal
use of nitrofurans in food animals: contribution to human salmonellosis?. Clin Microbiol Infect. 2006;12(11):1047-9. doi:
10.1111/j.1469-0691.2006.01539.x
5. García V, Montero I, Bances
M, Rodicio R, Rodicio MR. Incidence and genetic bases of nitrofurantoin resistance in clinical isolates of two successful multidrug-resistant clones of Salmonella enterica serovar Typhimurium: pandemic “DT 104”
and pUO-StVR2. Microb Drug Resist. 2017;23(4):405-12. doi: 10.1089/mdr.2016.0227.
6. McCalla
DR, Kaiser C, Green MHL. Genetics
of nitrofurazone resistance
in Escherichia coli.
J Bacteriol. 1978;133(1):10-6.
7. Peterson FJ, Mason RP, Hovsepian J, Holtzman JL. Oxygen-sensitive and -insensitive
nitroreduction by Escherichia coli and
rat hepatic microsomes. J Biol Chem. 1979;254:4009-14.
8. Martínez-Puchol S, Gomes C, Pons MJ, Ruiz-Roldán L, Torrents de la Peña A, Ochoa TJ, et al. Development and analysis of furazolidone-resistant Escherichia
coli mutants. APMIS.
2015;123(8):676-81. doi: 10.1111/apm.12401.
9. Whiteway
J, Koziarz P, Veall J, Sandhu N, Kumar P, Hoecher B, et al. Oxygen-insensitive
nitroreductases: analysis
of the roles of nfsA
and nfsB in development
of resistance to 5-nitrofuran derivatives
in Escherichia coli.
J Bacteriol. 1998;180(21):5529-39.
10. Shanmugam
D, Esak SB, Narayanaswamy
A. Molecular characterisation of nfsA
gene in nitrofurantoin resistant
uropathogens. J Clin Diagn Res. 2016;10(6):DC05-09. doi: 10.7860/JCDR/2016/17280.7957.
11. Ho PL, Ng
KY, Lo WU, Law PY, Lai EL,
Wang Y, et al. Plasmid-mediated OqxAB is an
important mechanism for nitrofurantoin resistance in Escherichia
coli. Antimicrob Agents Chemother. 2015;60(1):537-43. doi:
10.1128/AAC.02156-15.
12. Aviv G, Tsyba K, Steck N, Salmon-Divon M, Cornelius A, Rahav G, et
al. A unique megaplasmid
contributes to stress tolerance
and pathogenicity of an emergent Salmonella enterica
serovar Infantis strain. Environ Microbiol. 2014;16(4):977-94. doi: 10.1111/1462-2920.12351.
13. Ruiz-Roldán L, Martínez-Puchol S, Gomes C, Palma N, Riveros M, Ocampo K, et al.
Presencia de Enterobacteriaceae y Escherichia coli multirresistente a antimicrobianos en carne adquirida en
mercados tradicionales en Lima. Rev Peru Med Exp
Salud Publica. 2018;35(3):425-32.
doi:
10.17843/rpmesp.2018.353.3737.
14. Salazar de Vegas EZ,
Nieves B, Araque M, Velasco E, Ruiz J, Vila J. Outbreak
of infection with Acinetobacter strain RUH
1139 in an intensive care unit. Infect
Control Hosp Epidemiol.
2006;27(4):397-403. doi: 10.1086/503177.
15. Clinical
Laboratory Standard Institute
(CLSI). Performance standards for
antimicrobial susceptibility
testing; twenty-eight informational supplement
[Internet] CLSI document M100-S28. Wayne: CLSI; 2018
[citado el 11 de agosto de 2019]. Disponible en: https://clsi.org/media/2663/m100ed29_sample.pdf.
16. Salamanca-Pinzón SG,
Camacho-Carranza R, Hernández-Ojeda SL, Frontana-Uribe
BA, Espitia-Pinzón CI, Espinosa-Aguirre JJ. Correlation
of the genotoxic activation and kinetic properties of Salmonella enterica
serovar Typhimurium nitroreductases SnrA and cnr with the
redox potentials of nitroaromatic compounds and quinones. Mutagenesis. 2010;25(3):249-55. doi:
10.1093/mutage/geq001.
17. Pérez-Moreno MO,
Pico-Plana E, de Toro M, Grande-Armas J, Quiles-Fortuny
V, Pons MJ, et al. β-Lactamases,
transferable quinolone resistance determinants, and class 1 integron-mediated antimicrobial resistance in human
clinical Salmonella enterica
isolates of non-Typhimurium
serotypes. Int J Med Microbiol. 2013;303(1):25-31. doi:
10.1016/j.ijmm.2012.11.003.
18. McCracken RJ, Kennedy DG. Furazolidone in chicken: case study of an incident
of widespread contamination.
Br Poult Sci. 2013;54(6):704-12. doi:
10.1080/00071668.2013.850152.
19. Ruiz J. Transferable Mechanisms of quinolone resistance from 1998 onward. Clin Microbiol Rev. 2019;32(4):e00007-19. doi:
10.1128/CMR.00007-19.
20. Li XZ, Plésiat
P, Nikaido H. The challenge of efflux-mediated antibiotic resistance in Gram-negative bacteria. Clin Microbiol Rev. 2015;28(2):337-418.
doi: 10.1128/CMR.00117-14.
Fuentes de financiamiento: Este trabajo fue apoyado por la Sociedad Española
de Enfermedades Infecciosas y Microbiología Clínica 2012 (búsqueda de
antibióticos y microorganismos resistentes en animales de consumo humano y
piensos animales); JR fue apoyado por el programa I3 del Ministerio de Economía
y Competitividad, España (número de concesión: CES11/012). “ISGlobal
is a member of the CERCA Programme, Generalitat
de Catalunya”.
Citar como: Martínez-Puchol S, Pons MJ, Ruiz-Roldán L, Laureano-Adame L, Corujo A, Ochoa TJ, et al. Resistencia a nitrofuranos mediada por mutaciones en los genes cnr y snrA en Salmonella enterica procedentes de muestras cárnicas para consumo
humano. Rev Peru Med Exp Salud Publica.
2020;37(1):99-103. Doi: https://doi.org/10.17843/rpmesp.2020.371.4745.
Correspondencia: Joaquim Ruiz; Apartado 16, 08214-Badia del Valles, Barcelona,
España; joruiz.trabajo@gmail.com
Contribuciones de autoría: SMP, MJP y JR participaron en la concepción y
diseño del artículo. SMP, MJP, LRR, LLA y AC participaron en la recolección de
resultados. SMP, MJP y JR participaron en el análisis e interpretación de
datos. MJP, JR y TJO participaron en la redacción del artículo. Todos los
autores realizaron la revisión crítica del artículo, aprobaron la versión final
y asumen responsabilidad de los contenidos del manuscrito.
Conflictos de interés: Los autores declaran no tener conflicto de
interés.
Recibido:
14/08/2019
Aprobado:
22/01/2020
En línea:
23/03/2020