ARTÍCULO ORIGINAL
Identificación de especies de Leishmania en pacientes y flebotominos en áreas de transmisión en una región del Perú
Identification of Leishmania species in patients and phlebotomines in transmission areas in a region of Perú
Ofelia Córdova1,a, Franklin Vargas2,b, Yoshihisa Hashiguchi3,c, Hirotomo Kato3,c,b, Eduardo Gómez4,d
1 Universidad Privada Antenor Orrego. La Libertad, Perú.
2 Instituto de investigación de Microbiología y Parasitología Tropical, Universidad Nacional de Trujillo. La Libertad, Perú.
3 Universidad de Kochi. Kochi, Japón.
4 Universidad Católica de Guayaquil. Guayaquil, Ecuador.
a Microbióloga, Doctora en Biotecnología, Postdoctorado en Genética
b Microbiólogo y Parasitólogo, Doctor en Genética y Biotecnología
c Biólogo
d Médico Cirujano
RESUMEN
Objetivos. Identificar las especies de Leishmania presentes en las lesiones cutáneas del paciente y en las Lutzomyias que cohabitan en las áreas endémicas de la región La Libertad en el Perú. Materiales y métodos. Se usaron métodos moleculares basados en PCR y RFLP lo cual permitió obtener datos eficientes con poca muestra (pequeños especímenes), debido a su alta sensibilidad y las facilidades de aplicación en el trabajo de campo. Resultados. Los resultados del PCR de pacientes y de vectores, mostraron la presencia de Leishmania (V.) peruviana como principal agente causal de la Leishmaniosis tipo andina, transmitidas por Lutzomyia peruensis. Así mismo, se reveló la presencia de Leishmania (V.) guyanensis en Lutzomyia ayacuchensis. Conclusiones. Se mostró la presencia de L. (V.) peruviana y L. (V.) guyanensis en las áreas andinas en estudio. Hallazgos que exigen realizar una investigación más amplia sobre la distribución geográfica de L. (V.) guyanensis y las características clínicas relacionadas con la infección en áreas endémicas de Leishmaniosis cutánea.
Palabras clave: Leishmaniasis; Enfermedades Endémicas; Leishmaniasis Cutánea; Perú (fuente: DeCS BIREME).
ABSTRACT
Objectives. To identify the species of Leishmania present in the skin lesions of patients and Lutzomyias living in endemic areas of La Libertad, Peru. Materials and methods. Molecular methods based on PCR and RFLP were used, which allowed to have efficient data with small amounts of samples (small specimens), due to their high sensitivity and ease of application in the field work. Results. The results of PCR of clinical samples of patients and insect vectors showed the presence of Leishmania (V.) peruviana as a major causative agent of andean leishmaniasis transmitted by Lutzomyia peruensis. The presence of Leishmania (V.) guyanensis in Lutzomyia ayacuchensis, was found as well. Conclusions. The presence of L. (V.) peruviana and L. (V.) guyanensis in the Andean areas under study was found. These findings remark the need of a wider research about the geographical distribution of L. (V.) guyanensis and clinical features related to the infection in endemic areas of cutaneous leishmaniasis.
Key words: Leishmaniasis; Endemic Diseases; Leishmaniasis, Cutaneous; Peru (source: MeSH NLM).
INTRODUCIÓN
La Leishmaniosis es una enfermedad zoonótica producida por protozoos del género Leishmania y transmitida por la picadura de insectos hematófagos del género Phlebotomus en el viejo mundo y Lutzomyia en el nuevo mundo (1). Afecta a aproximadamente 12 millones de personas en 88 países, con una ocurrencia anual de 1,5 a 2 millones de casos de la forma cutánea y 500 000 casos de la forma visceral (2), e incidencias que han aumentado hasta 42 veces en los últimos 15 años (3).
En el Perú, la Leishmaniosis es endémica en el 74 % del territorio nacional, con un incremento sostenido del número de casos en los últimos 14 años, llegando hasta 83 casos nuevos por cada 100 000 habitantes (4).
Incidencias acumuladas que exigen realizar un análisis integral de los determinantes de riesgo del ambiente, la constitución genética del parásito o del hospedador; así como la distribución y las tasas de infección de los insectos transmisores (3).
Actualmente, se han descrito más de 800 especies de flebotominos, pero solo una parte de ellas pueden transmitir Leishmania. Asimismo, se han encontrado alrededor de veinte especies de Leishmania implicadas como agentes causales de la Leishmaniosis humana en el mundo, con características clínicas que varían con relación a las especies responsables de la infección, de la interacción parásito-hospedador y de la correlación genética de tal interacción (5). Por lo que, identificar la especie de Leishmania en las áreas endémicas, toma gran importancia en la estimación del pronóstico del paciente y la elección del tratamiento, mayor aun en las zonas en las que coexisten más de una especie patógena (6).
En la identificación de la especie de Leishmania causal de Leishmaniosis cutánea, se han usado técnicas cuya sensibilidad y especificidad se ven afectados por la habilidad o experticia que tenga la persona que toma y examina la muestra, el grado de contaminación bacteriana de la úlcera y la carga parasitaria al momento del examen, entre otras (7). Estas dificultades se pueden superar con pruebas como la reacción en cadena de la polimerasa (PCR), que es una técnica de alta sensibilidad y especificidad para el diagnóstico de la Leishmaniosis (6) y potencial herramienta de rápida detección e identificación del parásito en Lutzomyias silvestres (8).
Los métodos aplicados al estudio y caracterización de géneros y especies son diversos, por ejemplo el análisis del ADN kinetoplastídico (ADNk), la técnica de PCR seguida de la restricción del producto amplificado (PCRRFLP) y el uso de genes diana como el gen del citocromo b (cyt b), gen multicopia altamente polimórfico presente en los maxicírculos del kinetoplasto, muy útil en la identificación de especies de Leishmania (9).
Efectuar una identificación adecuada de especies o un diagnóstico positivo, reduce notablemente el riesgo de efectos adversos graves, así como la duración del tratamiento, especialmente en niños (10). Es, por tanto, muy importante identificar las especies de Leishmania empleando la técnica de PCR desde muestras clínicas o en Lutzomyias infectadas naturalmente, con resultados que contribuyan a predecir la intensidad de la infección y la tasa de riesgo de expansión de la enfermedad, en las áreas endémicas de la región La Libertad, Perú.
MATERIAL Y MÉTODOS
El presente estudio se realizó en los caseríos de La Cuesta a 1800-2000 m de altura y Nambuque a 2500 m de altura, distrito de La Cuesta, provincia de Otuzco, región La Libertad, Perú. Áreas endémicas de Leishmaniosis ubicadas a 7° 55’ 0” latitud Sur y 78° 43’ 0” latitud Oeste.
Para el examen de lesiones activas y aislamiento del parásito, se estudió 108 muestras clínicas de pacientes con antecedentes de vivir en zonas endémicas o de haberlas visitado; 48 resultaron positivas al examen clínico y parasitológico. Para el examen de las lesiones activas, se procedió a realizar raspado del margen externo de las lesiones cutáneas (11), confirmadas por exámenes dermatológicos. En los estudios parasitológicos se procedió a fijar las muestras extraídas en metanol y teñir con Giemsa para su observación microscópica (frotis). Otra parte de la muestra fue adsorbidas en tarjetas FTA® (Whatman BioScience, Newton Center, MA) para su correspondiente análisis molecular.
CAPTURA DE LUTZOMYIAS Y EXTRACCIÓN DEL ADN
Se estudiaron 190 especies de Lutzomyias, capturadas en trabajo de campo con trampa de luz Shannon, cebo humano y trampa de luz CDC (Communicable Disease Center).
En el empleo del método de cebo humano, las hembras de Lutzomyias fueron atraídas sobre colectores y aspirados con capturadores de vidrio. Para el caso de la trampa CDC, las Lutzomyias fueron atraídas por la luz y succionados por el flujo de aire generado por la hélice. Utilizando capturas diurnas y nocturnas de Lutzomyias cada dos meses, tanto en domicilio (intradomicilio), refugios naturales (huecos de árboles, rocas, madrigueras de animales, etc.) y alrededores (peridomicilio).
Las Lutzomyias fueron mantenidas vivas y transportadas en contenedores de plástico correctamente rotulados para su procesamiento en el laboratorio, donde fueron sacrificadas exponiéndolas a -21 ºC para luego ser diseccionadas bajo estereoscopio, se separó la parte terminal del abdomen a fin de completar la identificación taxonómica de acuerdo con las características morfológicas de sus espermatecas, empleando la clasificación propuesta por Young y Duncan (1994) (12). Los especímenes diseccionados e identificados fueron fijados individualmente en etanol al 70 % para su posterior análisis molecular.
La extracción del genoma de Leishmania en muestras clínicas, como en Lutzomyias colectados, se realizaron siguiendo los protocolos descritos por Marco et al. (2006) (13).
Para la extracción y purificación del ADN de Leishmania adsorbidas en las tarjetas clásicas FTA®, se siguieron las recomendaciones establecidas por la casa comercial BioScience (2002); los discos de 2 mm de diámetro de las tarjetas FTA® (Whatman BioScience) formados previamente, se lavaron por triplicado con el reactivo de purificación FTA, seguido de una elución del purificado, con 50 μL de tampón Tris-EDTA en columnas de extracción, utilizando 5 μL del eluído en la amplificación por PCR.
Para la extracción de DNA a partir de Lutzomyias, se lisaron los fragmentos de tórax y de segmentos proximales del abdomen de cada espécimen, en 50 μL de tampón de lisis (NaCl 150 mM, tris-HCl 10 mM pH 8,0, EDTA 10 mM, y SDS al 0,1 %) con 3 μL de proteinasa K (20 mg/mL), a 37 ºC y por 12 h, seguido de un centrifugado a 12 000 rpm durante 10 min. La fase acuosa obtenida fue transferida a un nuevo tubo y se le adicionó igual volumen de fenol saturado con cloroformo/ alcohol isoamílico (24:1). La fase acuosa de esta última extracción se precipitó en presencia de acetato de sodio 1:10 (vol/vol), con dos volúmenes de etanol e isopropanol para precipitar el DNA. Finalmente, se centrifugó a 12 000 rpm por 5 min y el DNA precipitado (pellet) fue digerido con RNAasa pancreática durante una hora a 37 °C, posterior lavado con etanol al 70 % durante 30 s y guardado a -20 ºC en 25 μL de agua ultra pura, hasta su amplificación.
IDENTIFICACIÓN MOLECULAR DE ESPECIES DE LEISHMANIA
En la identificación molecular del parásito, se seleccionaron previamente los oligonucleótidos cebadores que amplifican la región codificadora del gen del citocromo b (Cyt b) (AB095960) de 863 pb (9): L.cyst-AS (5’-gcggagaggaaagaaaaggc- 3’) y L.cyst-AR (5’ccactcataaatatactata- 3’) para la identificación del género Leishmania a partir de muestras de lesiones cutáneas. Y L.cyst-innerS (5’-ggtgtgggttttagtttagg-3’) con L.cyst-innerR (5’-ctacaataaacaaatcataatatg- 3’) para la determinación de Leishmania en las Lutzomyias colectadas.
Las muestras de lesiones positivas para Leishmania, fueron sometidas a una segunda PCR (PCR interna) con los oligonucleótidos cebadores: L.cyst-innerS (5’-ggtgtgggttttagtttagg- 3’) con L.cyst-innerR (5’-ctacaataaacaaatcataatatg- 3’) con amplificaciones de 886 pb (9).
Como control positivo y de especificidad se utilizaron las secuencias de referencia: L. (V.) braziliensis (MHOM/ BR/75/M2904), L. (V.) peruviana (MHOM/PE/84/LC39), Leishmania (V) panamensis (MHOM/CO/87/CL-412), L. (V.) guyanensis (MHOM/CO/85/CL-220). L. (L.) mexicana amazonensis (MHOM/ME/94/CL-856). Como control de especificidad para Leishmania se utilizó una cepa de Trypanosoma cruzi (MHOM/ CO/92/Fch).
Para la diferenciación de especies se utilizaron los oligonucleótidos cebadores que amplifican la región codificadora del gen de la isomerasa manosa fosfato (MPI) (EU 327892): MPI-S (5’-gctcttccgtcggacagcgagc-3’) y MPI-R (5’-tcactctcgaagggagttcg-3’). Una porción del amplificado del PCR fue sometida a una segunda PCR con los oligonucleótidos iniciadores: MPIinnerS (5’-gagcacacgcgtacatcag-3’) y MPI-innerR (5’-tcggacacgtgccgctcaag-3’).
AMPLIFICACIÓN POR PCR Y ANÁLISIS ELECTROFORÉTICO
Las amplificaciones fueron realizadas en mezclas de 50 μL de volumen final, con 2,5 μL de DNA y 47,5 μL de mezcla de reacción (conteniendo 2 μM de cada oligonucleótido iniciador), 2 μL de cada dNTPs, 5 μL de tampón de PCR 10X (conteniendo 50 mM MgCl2, 10 mM KCl; Tris-HCl (pH 8,3), 20 μL de agua destilada de alto grado molecular ultrapura M de dNTPs; y 10 U/ μL de Taq DNA polimerasa (Biotools B&M Labs, S.A. Madrid, España).
La amplificación de PCR fue realizada con un calentamiento inicial de placa a 95 ºC por 10 min, seguido de 35 ciclos conteniendo los siguientes parámetros: desnaturalización a 94 °C por 30 s, alineación a 45 ºC por 30 s; y extensión a 72 °C por 1 min, amplificación que fue completada con una incubación a 72 °C por 5 min hasta la completa extensión de los productos de PCR.
Una fracción de cada uno de los productos de PCR se mezcló con tampón de muestra 6X y separados por electroforesis en geles de poliacrilamida al 4 % conteniendo SYBR Green, usando TAE 1X (89 mM Tris HCL, 90 mM de ácido bórico y 20 mM de EDTA, pH = 8,3) como tampón de corrido. Los productos se visualizaron en un transiluminador de luz ultravioleta (Biorad). El tamaño del fragmento fue determinado al comparar los pares de bases del amplificado con los pares de bases de los marcadores de bases (ladder de 1 kb Bitools)
Para la diferenciación entre especie, se realizó un análisis de polimorfismo de un solo nucleótido (SNP) de los amplificados que codifica los genes MPI de L. (V.) braziliensis (MHOM/BR/75/M2904), L. (V.) peruviana (MHOM/PE/84/LC39) de las muestras en estudio, digeridos con las enzima de restricción Ava ll (Takara BioShiga, Japón). Una vez digeridos, fueron separados mediante electroforesis en geles de agarosa al 0,8 y visualizados con luz ultravioleta.
ANÁLISIS DE HOMOLOGÍA
Los productos de amplificación del PCR fueron purificados utilizando el kit de purificación (Wizard® PCR Preps DNA Purificación System Kit-Promega). Para el análisis de homología se accedió a la búsqueda de secuencias en el genbank de doce especies de Leishmania: L. (L.) donovani (AB095957), L. (L.) chagasi (AB095959), L. (L.) aethiopica (AB095962), L. (L.) major (AB095961), L. (L.) tropica (AB095960), L. (L.) amazonensis (AB095964), L. (L.) garnhami (AB095965), L (L.) mexicana (AB095963), L. (L.) panamensis (AB095968), L. (L.) braziliensis (AB095966), L. (L.) guyanensis (AB095969) y L. (L.) peruviana. El alineamiento múltiple de las secuencias se realizó con el programa Clustal W (14) y la identificación de sustituciones homólogas y no homólogas, determinando los sitios polimórficos y las distancias genéticas pareadas netas (p), así como la distribución de la variabilidad genética a lo largo del alineamiento múltiple del gen citocromo b, y la estimación de probabilidad máxima (MLE) trasladadas a aminoácidos en el programa MEGA 3.1 (15).
Todos los participantes dieron su consentimiento informado, luego de las charlas informativas que se les brindó en la zona de estudio. Los cuestionarios fueron preparados según la ocupación de cada sujeto de estudio, historia natural de la enfermedad, lesiones leishmaniásicas (localización, tipo, tamaño, tiempo de evolución) y tratamiento. El protocolo de estudio fue aprobado por el Comité de Ética del Instituto Nacional de Salud (INS).
RESULTADOS
El 85 % de la población de los caseríos viven en el área y se dedican a la agricultura (trigo y maíz como productos principales, además de papa, yuca, trigo y naranja, entre otros vegetales) también crían vacas, cerdos, aves de corral y cuyes, y conviven con perros, gatos, caballos, ovejas, cuyes, pollos, etc.
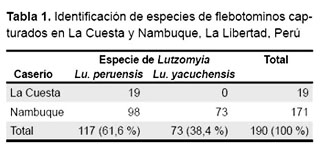
En estos caseríos, se colectaron 190 Lutzomyias, de los cuales 175 fueron machos y 15 fueron hembras. Se identificaron como Lu. peruensis (capturados con trampa CDC) y Lu. ayacuchensis (capturados por cebo humano protegido). Ambos especímenes resultaron positivos a la amplificación de las secuencias de los genes cyt b para Leishmania, como se describe en la Tabla 1.

Al realizar la amplificación de la secuencia codificadora del gen Cyt b y gen MPI de los casos sospechosos para Leishmania spp, mostraron que de las 48 muestras en estudio, 31 eran positivas para el gen Cyt b y 17 para el gen MPI, como se muestra en la Tabla 2.
El porcentaje de homología de la secuencia del gen cytb, mediante el programa Clustal y MEGA v3.1, muestran una alta identidad de las leishmanias aislada de muestras clínicas como Lu. peruensis (recolectado en La Cuesta), con L. (V.) braziliensis y L. (V.) peruviana. Mientras que el análisis molecular del gen cit b en Lu. ayacuchensis del caserío de Nambuque revelan alto porcentaje de homología con L. guyanensis, como se muestra en la Tabla 3.
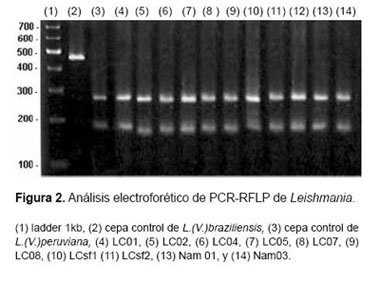
El estudio con la enzima Ava ll que solo digiere los fragmentos del gen MPI de L.(V.) peruviana de los aislados en estudio (LC 01, 02, 04, 05, 07, 08; Nam 01, Nam 02 y LC 09, 10) con L. (V.) peruviana; como se ve en la Figura 1 y Figura 2.

DISCUSIÓN
La identificación del complejo o la especie de Leishmania aislada en el presente estudio por amplificación del gen del citocromo b (cyt b) (10), ha permitido obtener resultados similares a los hallados por Marco et al. (13) en la identificación de especies de Leishmania en Argentina. De igual manera, la presencia de Leishmania (Viannia) peruviana en lutzomyias por métodos moleculares, confirma los observado previamente por Zhang et al. (2007) (16) quienes informan a L. peruviana como el agente más importante y de gran pertinencia epidemiológica de la Leishmaniosis cutánea en la región La Libertad.
Los pacientes incluidos no presentaron mal nutrición, Línea 1: marcador de tamaño de ADN-ladder 1kb, líneas 2 a 7: muestras aisladas de pacientes en La Cuesta. Líneas 8 a 12: muestras aisladas de pacientes en Nambuque. Líneas de 13 a 23: muestras aisladas de Lutzomyia colectadas en La Cuesta. Línea 24 corresponde a la muestra aislada de Lutzomyia colectada de Nambuque. Línea 25 control negativo [DNA de T. cruzi (MHOM/ CO/92/Fch)]. Línea 1: marcador de tamaño de ADN-ladder 1kb, líneas 2 a 7: muestras aisladas de pacientes en La Cuesta. Líneas 8 a 12: muestras aisladas de pacientes en Nambuque. Líneas de 13 a 23: muestras aisladas de Lutzomyia colectadas en La Cuesta. Línea 24 corresponde a la muestra aislada de Lutzomyia colectada de Nambuque. Línea 25 control negativo [DNA de T. cruzi (MHOM/ CO/92/Fch)]. infección generalizada ni hepatomegalia, esplenomegalia o adenitis, particular de una Leishmaniosis visceral, por el contrario, las lesiones se caracterizaron por ser localizadas y, en algunos casos, aparentemente metastásicas y clínicamente concordantes con lo descrito por Lambrechts et al. (5).
La obtención de resultados positivos con la técnica de PCR tiene la ventaja de necesitar poco volumen de muestra para el diagnóstico, de forma similar a lo que sucede con otros métodos utilizados en investigaciones clínicas. La identificación de especie de Leishmania (1) ladder 1kb, (2) cepa control de L.(V.)braziliensis, (3) cepa control de L.(V.)peruviana, (4) LC01, (5) LC02, (6) LC04, (7) LC05, (8) LC07, (9) LC08, (10) LCsf1 (11) LCsf2, (13) Nam 01, y (14) Nam03. directamente a partir de lesiones (Tabla 2) abre la posibilidad de aplicar la PCR directamente en muestras clínicas, evitando el paso por cultivos tediosos y con riesgo de contaminación bacteriana (7).
El análisis molecular de la secuencia génica del cyt b de los casos sospechosos (Tabla 2) muestra valores por debajo de lo esperado, sugiriendo la existencia de heterogeneidad de cebador en la PCR, por lo que de optimizarse el diseño con cebadores más específicos, permitiría su uso como gen diana de determinación de heterogeneidad de cepas (17).
La identificación de especie, mediante el secuenciamiento del gen cyt b (Tabla 3) sugiere que todos los pacientes fueron infectados con especies del subgénero Viannia, resultados corroborados a su vez con el RFLP (Polimorfismo de longitud de fragmentos de restricción) después de la PCR (Figura 1) proporcionando datos fáciles de informatizar e intercambiar entre laboratorios, construir un árbol filogenético e identificar la especie de Leishmania patógena para los humanos de las especies no patógenas para los humanos (10).
La elección de los oligonucleótidos cebadores L.cyst-AS y L.cyst-AR, utilizados con éxito en otras investigaciones (9,10), y el diseño de L.cyst-innerS con L.cyst-innerR, utilizados en una PCR anidada, dieron amplificaciones de fragmentos de 886 pb de tamaño, semejantes a los obtenidos en el control positivo de subgénero y similares a lo descrito por y Kato et al. (2007) (18) para Leishmania del subgénero Viannia. El uso de una PCR mejorada con PCR interna o nested, (realización de dos PCR consecutivas), en la que el producto amplificado de la primera reacción sirve de molde para la segunda, siempre optimiza la identificación genética.
Contar con esta técnica molecular en el laboratorio permitiría la identificación genética de especies y podría constituir una herramienta útil para la identificación de blancos terapéuticos, factores de virulencia, antígenos vacunales e individualización de especies patógenas, sobre todo en viajeros con infecciones leishmaniásicas cutáneas en el nuevo o viejo mundo.
Los fragmentos del gen MPI digeridos con las enzimas Ava II, enzimas que se comportan como buenos marcadores de diferenciación de especie, dado que digieren solo los fragmentos de L. (V.) peruviana y no de L. (V.) braziliensis (19) y evidenciados en la Figura 1, revelan una infección con L. (V.) peruviana (Figura 2); hallazgos semejantes a los encontrados en muchas investigaciones en los que se indica a L. (V.) peruviana como el principal agente causal de Leishmaniosis cutánea tipo andina en el Perú (17).
Se considera que en zonas andinas de la región La Libertad, prevalece la forma cutánea andina o uta causado por L. (V.) peruviana; además, es factible que exista más de una variedad genotípica asociada con variedades clínicas de la forma cutánea (4). En relación con la identificación de especie de Lutzomyias en los caseríos de La Cuesta y Nambuque (Tabla 1), se observa la existencia de Lu. peruensis y Lu. ayacuchensis. Los oligonucleótidos cebadores utilizados en este estudio, confirmaron su presencia en insectos transmisores de Leishmaniosis en zonas andinas de la región La Libertad. Los patrones de amplificación de secuencias del gen cyt b de Leishmania concuerdan con el subgénero Vianni; lo cual corrobora su utilidad como gen diana altamente eficaz para la identificación molecular de especies de Leishmania en los insectos vectores de Leishmaniosis cutánea en áreas endémicas (10).
Por otro lado, los resultados positivos a la amplificación de las secuencias de Leishmania (V.) guyanensis (16) y los encontrados en el análisis de homología de la especie de Leishmania aisladas de Lutzomyia, revelan un alto nivel de homología con L. (V.) peruviana presente en Lu. peruensis, y de L. (V.) guyanensis en Lu. ayacuchensis (Tabla 3), e indican que L. (V.) peruviana es la especie más frecuente en el caserío de La Cuesta y trasmitida por Lu. peruensis; mientras que, L. (V.) guyanensis es la especie más persistente en el caserío de Nambumque y que se encuentra infectando a Lu. ayacuchensis. Hallazgos que guardan coherencia con los diversos estudios realizados en el Perú, donde la Leishmaniosis cutánea andina se debe a L. (V.) peruviana, y transmitida principalmente por Lu. peruensis, Lu. verrucarum, Lu. ayacuchensis o Lu. tejadai. (16,17).
Por otro lado, existen pocos casos estudiados con L. (V.) guyanensis, pues se ha informado su presencia en 24 pacientes de 351 casos residentes en los departamentos de Amazonas, Ancash, Cuzco, San Martín, Huánuco, Ucayali y de Junín, excepto uno, que fue aislado de la selva amazónica a altitudes de entre 80 y 1000 m. Al comparar esta información con nuestro estudios, L .(V.) guyanensis se encontró en el caserío de Nambuque, región La Libertad, comunidad andina a 2500 m de altura aproximadamente, en la que antes no se había registrado; marcando una gran diferencia con la distribución conocida de esta especie de parásito (17, 19).
Encontrar Lu. ayacuchensis infectado con L. (V.) guyanensis es un hallazgo de gran interés, sobre todo porque Lu. ayacuchensis es un vector conocido por ser muy antropofílico y transmitir L. (V.) peruviana en los valles andinos, sugiriendo la posibilidad de ser transmitida a los seres humanos.
L. (V.) guyanensis es una especie poco común en pacientes peruanos, según los estudios retrospectivos sobre la distribución geográfica de los agentes causales de especies de la Leishmaniosis en el Perú (19), su identificación sugiere la existencia de un nuevo nicho ecológico para el parásito, importante factor de riesgo de brotes epidémicos en la población. Riesgo que se ve exacerbado por la presencia de lesiones metastásicas con localizaciones subcutáneas linfonodulares, frecuente en la zonas endémicas de Leishmaniosis en la región La Libertad. Características clínicas descritas por Cuba (19) al examinar cierto porcentaje de individuos parasitados con algunas especies del subgénero Viannia y en los que observó el desarrollo de lesiones características clínicas atribuidas a las formas esporotricoides causadas por L. (V).guyanensis (17).
Realizar análisis con un mayor número de enzimas de restricción permitirá la tipificación de nuevas regiones de ADN nuclear o kinetoplasto y marcadores específicos de especies, a fin de precisar la real magnitud de la variabilidad genética de la Leishmania y la presencia de nuevas especies del parásito. No obstante, estas variantes pueden estar asociadas con la L. (V.) peruviana, L. (V.) guyanensis. Continuar con estos estudios permitirá delimitar microregiones con potenciales riesgos de transmisión de la enfermedad al conocer la diversidad de las especies de Lutzomyias infectadas, favoreciendo de esta manera, la comprensión de los factores ecológicos y geográficos que influirían en la transmisión de la enfermedad, conocimientos necesarios para diseñar futuras medidas de control epidemiológico.
Una investigación más amplia sobre los parásitos en los pacientes y Lutzomyias en Nambuque, como en otras áreas andinas y endémicas de Leishmaniosis cutánea, proporcionaría conclusiones más interesantes sobre la distribución geográfica de L. (V.) guyanensis y las características clínicas relacionadas con la infección.
En conclusión, se observa la existencia de Lu. peruensis y Lu. ayacuchensis, siendo Lu. peruensis la especie más predominante. Y Leishmania peruviana, es la especie parasitante en el humano y en las Lutzomyia, y L. (V.) guyanensis, está parasitando a Lu. ayacuchensis.
AGRADECIMIENTOS
Agradecemos a todos los médicos y personal de salud de la áreas andinas en estudio de la región La Libertad y de manera especial al Dr. Yoshihisa Hashiguchi y su equipo investigación, por el apoyo logístico y científico durante la investigación.
Contribuciones de autoría
OC y FV, participaron en la concepción y diseño del trabajo, YH y EG en el análisis e interpretación de datos y en la obtención del financiamiento. OC, FV y HK en la recolección de resultados, en el análisis e interpretación de datos y en la asesoría estadística. FV y EG en el manejo de pacientes y diagnóstico de la leishmaniosis, OC, FV, YH, HK y EG participaron en la recolección de resultados, revisión crítica del manuscrito y redacción escrita. Todos los autores aprobaron la versión final del trabajo.
Fuentes de financiamiento
El presente trabajo fue posible por el apoyo del Ministerio de Educación y Cultura de Japón, y de recursos propios de la Universidad Nacional de Trujillo.
Conflictos de interés
Los autores declaran no tener conflictos de interés.
REFERENCIAS BIBLIOGRÁFICAS
1. Munstermann LE. Phlebotomine sand flies, the Psychodidae. En: Masquardt WC, Freirer J, Hagedorn H, Hemingway B, Moore C (edit.). Biology of disease vectors. 2nd ed. San Diego, CA: Elsevier; 2004. p 141-51.
2. Croft SL, Sundar S, Fairlamb AH. Drug Resistance in Leishmaniasis. Clin Microbiol. Rev. 2006;19:111-26.
3. Organización Mundial de la Salud (OMS). Control de la leishmaniasis. Informe de la Secretaría 118º Reunión. Ginebra: OMS; 2006. p. 1-7.
4. Cabrera R. Importancia del conocimiento de los determinantes de riesgo de la leishmaniosis para la vigilancia, prevención y control dirección general de epidemiología. Bol Epidemiol (Lima). 2008;17(14).
5. Lambrechts L, Fellous S, Koella JC. Coevolutionary interactions between host and parasite genotypes. Trends Parasitol. 2006;22:12-6.
6. Schönian G, Nasereddin A, Dinse N, Schweynoch C, Schallig HD, Presber W, et al. PCR diagnosis and characterization of Leishmania in local and imported clinical samples. Diag Microbiol Infect Dis. 2003;47:349-58.
7. Garcia AL , Parrado R, De Doncker S, Bermudez H, Dujardin JC. American tegumentary leishmaniasis: direct species identification of Leishmania in non-invasive clinical samples. Trans R Soc Trop Med Hyg. 2007;101(4):368-71.
8. de Pita-Pereira D, Alves CR , Souza MB, Brazil RP, Bertho AL , de Figueiredo Barbosa A, et al. Identification of naturally infected Lutzomyia intermedia and Lutzomyia migonei with Leishmania (Viannia) brasziliensis in Rio de Janeiro (Brazil) revealed by a PCR multiplex non-isotopic hybridisation assay. Trans. R. Soc. Trop. Med. Hyg. 2005;99:905-13.
9. Luyo-Acero G, Uezato H, Oshiro M, Takei K, Kariya K, Katakura K, et al. Sequence variation of the cytochrome b gene of various human infecting members of the genus Leishmania and their phylogeny. Parasitology. 2004;128:483-91.
10. Foulet F, Botterel F, Buffet P, Morizot G, Rivollet D, Deniau M, et al. Detection and identification of Leishmania species from clinical specimens by using a real-time PCR assay and sequencing of the cytochrome B gene. J Clin Microbiol. 2007;45(7):2110-5.
11. Reithinger R, Dujardin JC. Molecular diagnosis of leishmaniosis: current status and future applications. J Clinic Microbiol. 2007;45:21-5.
12. Young DG, Duncan MA. Guide to the identification and geographic distribution of Lutzomyia sand flies in Mexico, the West Indies, Central and South America (Diptera: Psychodidae). Mem Amer Ent Inst. 1994;54:1-881.
13. Marco J, Uezato H, Mimori T, Barroso P, Korenaga M, Nonaka S, et al. Are cytochrome B gene sequencing and polymorphism-specific polymerase chain reactions as reliable as multilocus enzyme electrophoresis for identifying Leishmania spp. from Argentina? Am J Trop Med Hyg. 2006;75(2):256-60
14. Thompson JD, Higgins DG, Gibson TJ. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting position- specific gap penalties and weight matrix choice. Nucleic Acids Res. 1994;22(22):4673-80.
15. Kumar S, Tamura K, Nei M. MEGA 3: Integrated software for molecular evolutionary genetics analysis and sequence alignment. Brief Bioinform. 2004;5(2):150-63.
16. Zhang WW, Miranda-Verastegui C, Arevalo J, Ndao M, Ward B, Llanos-Cuentas A, et al. Development of a genetic assay to distinguish between Leishmania viannia species on the basis of isoenzime differences. Clin Infect Dis. 2006;42(6):801-9.
17. Caceres AG , Villaseca P, Dujardin JC, Bañuls AL , Inga R, Lopez M, et al. Epidemiology of Andean cutaneous leishmaniosis: incrimination of Lutzomyia ayacuchensis (diptera: psychodidae) as a vector of leishmania in geographically isolated, upland valleys of Peru. Am J Trop Med Hyg. 2004;70(6):607-12.
18. Kato H, Uezato H, Gomez E, Terayama Y, Calvopiña M, Iwata H, et al. Establishment of a mass screning method of sand fly vectors for Lesihmania infection by molecular biological methods. Am J Trop Med Hyg. 2007;77(2):324-9.
19. Cuba C. Diagnóstico parasitológico de la leishmaniosis tegumentaria americana. Rev Peru Med Exp Salud Publica. 2000;17(1-4):39-52.
Correspondencia:
Franklin Vargas Vásquez Instituto de Investigación de Microbiología y Parasitología Tropical, Facultad de Ciencias Biológicas. Universidad Nacional de Trujillo. Av Juan Pablo II s/n. La Libertad, Perú.
Teléfono: 044 -222331.
Correo electrónico: frvargasv@yahoo.es
Recibido: 24-01-11
Aprobado: 10-08-11