César Núñez Ponce
10.17843/rpmesp.2020.372.5490
ARTÍCULO DE REVISIÓN
COVID-19: respuesta inmune y perspectivas terapéuticas
COVID-19: the novel SARS-CoV-2 pandemic
Iván Lozada-Requena
![]() 1, Doctor en Ciencias de la Vida
1, Doctor en Ciencias de la Vida
César Núñez Ponce
![]() 2, Médico Cirujano
2, Médico Cirujano
1 Facultad de
Ciencias y Filosofía, Universidad Peruana Cayetano Heredia, Lima, Perú.
2 Empresa de Investigación y Desarrollo (EMINDES) SAC, Lima,
Perú.
RESUMEN
La enfermedad causada por el nuevo coronavirus (COVID-19) se caracteriza por presentar fiebre y tos, afectar el tracto respiratorio inferior y estar asociada con la edad, comorbilidades y un sistema inmune debilitado. Típicamente se ha evidenciado linfopenias en los casos graves y una desmedida producción de citocinas inflamatorias (tormenta de citocinas), lo que explicaría el rol de la respuesta hiperinflamatoria en la patogénesis de la COVID-19. Las respuestas inflamatorias secundarias por reinfecciones del virus podrían inducir el aumento o la mejora dependiente de anticuerpos (ADE, por sus siglas en inglés), un fenómeno virémico que podría ser un mecanismo alternativo de infección celular y que se deberá tener en cuenta cuando se diseñen vacunas o inmunoterapias que involucren el estímulo de anticuerpos neutralizantes o el uso de anticuerpos monoclonales. Actualmente no existen vacunas ni tratamientos que demuestren seguridad y eficacia en pacientes con COVID-19; sin embargo, se espera la conclusión de los resultados de la aplicación de una vacuna de ácidos nucleicos ARNm (mensajero del ácido ribonucleico) y de un fármaco antiviral (remdisivir) que se encuentran en ensayos clínicos fase III. Por el momento la mejor medida para evitar la propagación de la infección es el aislamiento social exhaustivo y viene siendo adoptado por varios países según recomendación de la Organización Mundial de la Salud (OMS).
Palabras clave: Síndrome Respiratorio Agudo Grave; Neumonía Viral; Virosis; Infecciones por Coronavirus; Pandemias; Anticuerpos; Citocinas; Linfocitos; Vacunas; COVID-19 (fuente: DeCS BIREME).
ABSTRACT
Disease caused by the new coronavirus (COVID-19) is characterized by fever, cough, and affection of the lower respiratory tract. It is associated with age, comorbidities and a weakened immune system. Typically, lymphopenias have been evidenced in severe cases and an excessive production of inflammatory cytokines (cytokine storm), which would explain the role of the hyperinflammatory response in the pathogenesis of COVID-19. Secondary inflammatory responses from virus reinfections may induce antibody-dependent enhancement (ADE), a viremic phenomenon that may be an alternative mechanism of cellular infection and should be considered when designing vaccines or immunotherapies involving the stimulation of neutralizing antibodies or the use of monoclonal antibodies. Currently, no vaccines or treatments demonstrate safety and efficacy in patients with COVID-19. However, the results from phase III clinical trials which involve the application of an mRNA (messenger ribonucleic acid) nucleic acid vaccine and an antiviral drug (remdisivir), are yet to be concluded. For the time being, the best measure to prevent the spread of COVID-19 is by implementing social isolation, this measure has been adopted by several countries as recommended by the World Health Organization (WHO).
Keywords: Severe Acute Respiratory Syndrome; Viral Pneumonia; Virosis; Coronavirus infections; Pandemics; Antibodies; Cytokines; Lymphocytes; Vaccines; COVID-19 (source: MeSH NLM).
INTRODUCCIÓN
El SARS (síndrome respiratorio agudo grave) es el estadio grave de la COVID-19 producido por un daño masivo alveolar y una falla respiratoria progresiva; ocasionado por el SARS-CoV-2 (coronavirus 2 del SARS) (1). La COVID-19 se inició en diciembre de 2020 en Wuhan, provincia de Hubei (China). Al 17 de abril de 2020, el SARS-CoV-2 ha infectado en todo el mundo a 1 991 562 personas; han muerto 130 885; Europa es el continente con la mayor cantidad de casos (1 013 093), mientras que en el continente americano hay 707 121 casos confirmados y 30 245 muertes. El Perú ocupa el cuarto lugar, con 13 489 casos confirmados y 300 muertos, y muestra una tendencia al incremento (2). En Lima se ha notificado la mayor cantidad de casos (9793 confirmados), seguida por el Callao (1080 confirmados) y Lambayeque (600 confirmados) (3).
El SARS-CoV-2 pertenece a la familia Coronaviridae, subfamilia Orthocoronaviridae. Es un virus ARN de hebra simple, cuyo genoma es de aproximadamente 27-32 kb, que codifica proteínas no estructurales, como proteasas, helicasas y ARN polimerasas; y proteínas estructurales: de membrana (M), de envoltura (E), nucleocápside (N) y la proteína espiga (S). El SARS-CoV-2 utiliza la proteína espiga para infectar a las células epiteliales (células alveolares tipo II, AT2) de pulmón e intestino a través de una proteína receptora de membrana, la enzima convertidora de angiotensina 2 (ACE2, por sus siglas en inglés), de la misma forma que lo hace el virus SARS-CoV; mientras que el MERS-CoV (coronavirus del síndrome respiratorio de oriente medio) utiliza el receptor DDP4 (dipeptidil peptidasa-4) (4–6). Por tanto, no es extraño que las proteínas no estructurales y la proteína estructural S hayan sido el blanco terapéutico del SARS-CoV y MERS-CoV (7). Además, la proteína receptora ACE2 se encuentra en células del miocardio (7,5%), en células epiteliales del íleo (30%) y del esófago (>1%), en células de los túbulos proximales del riñón (4%) y en células uroteliales de la vejiga (2,4%), y son órganos de alto riesgo de infección (4).
Se cree que la zoonosis proviene de murciélagos, pero la fuente exacta y los reservorios animales aún son inciertos. Probablemente sean los gatos civetas o los pangolines (SARS) y los camellos o dromedarios (MERS) (5,6). Entre humanos la transmisión por contacto es la principal y aunque solo hay un estudio caso-control de transmisión madre-hijo, se ha demostrado que cuatro recién nacidos de madres infectadas con COVID-19 fueron negativos a las pruebas moleculares (8).
Además del SARS-CoV-2, existen más especies de coronavirus: NL63, 229E, OC43, HKU1, SARS-CoV y MERS-CoV y los más patogénicos para el ser humano son el SARS-CoV, que brotó en China (2002 y 2003), y el MERS, que brotó en Arabia Saudita (2012). Con estas especies, el SARS-CoV-2 guarda una homología de genoma del 76-79% y 50%, respectivamente (9-12). Las tres infecciones por CoV se caracterizan por presentar fiebre y tos, por comprometer el tracto respiratorio inferior y por estar asociado a la edad y comorbilidades con un sistema inmune debilitado; sin embargo, la COVID-19 está dejando mayor letalidad (10,13).
La búsqueda bibliográfica para la presente revisión se ha hecho en PubMed, incluye trabajos originales y revisiones temáticas preaprobados y aprobados para publicación y de acceso libre desde diciembre de 2019 hasta marzo de 2020 con temas de revisión obligatoria. Los términos usados han sido «COVID-19», «SARS-CoV-2» cruzados con los principales temas tratados en esta revisión.
RESPUESTA INMUNE EN LA INFECCIÓN POR SARS-COV-2
La COVID-19 se puede dividir en tres fases: asintomática con o sin virus detectable; sintomática no grave con presencia de virus y sintomática respiratoria grave con alta carga viral (14). Una pregunta no resuelta es por qué algunos desarrollan enfermedad grave y otros no. Los aspectos basados en la respuesta inmune no son suficientes para explicarlo, pero ayudarán a entender el comportamiento de este nuevo patógeno.
Los linfocitos T (LT), linfocitos B (LB) y las asesinas naturales (NK) tienen un papel importante en mantener el sistema inmune. En la infección por SARS-CoV-2, los estudios demuestran que hay una marcada linfopenia (15). En la sangre de un paciente se encontró una linfopenia de LT CD4+ y CD8+; sin embargo, ambas poblaciones presentaban un estatus hiperactivado con altas proporciones de CD4+HLA-DR+ (3,5%) y CD8+CD38+ (39,4%). Además, se encontraron proporciones elevadas de LT proinflamatorios CD4+CCR6+ y LT CD8+ con altas cantidades de gránulos citotóxicos. Estas poblaciones linfocitarias podrían explicar parcialmente el grave daño al sistema inmune (1). En otros pacientes con infección grave también se han observado linfopenias, mayor relación neutrófilos/linfocitos, menor cantidad de monocitos, eosinófilos y basófilos, en comparación con los pacientes sin síntomas de la enfermedad.
Dentro de los grupos celulares más afectados están los LT (CD4+ y CD8+), que estuvieron por debajo de los valores normales y fue más evidente en el caso de los LT CD4+ de pacientes graves. Estos resultados coinciden con los de Wang et al. en cerca del 30% de pacientes con enfermedad grave (15). Por otro lado, los LT CD4+CD45RA+ (vírgenes) se incrementan mientras que los LT CD4+CD45RO+ (memoria) disminuyen. El nivel de activación solo disminuye en los LT CD8+CD28+ junto con una disminución de LT CD4+ reguladores vírgenes (CD45RA+CD127Low+) e inducidos (CD45RA+CD127Low+). Estos datos también sugieren que el sistema inmune está desregulado durante el curso de la enfermedad por SARS-CoV-2 y es más crítica cuando el paciente tiene comorbilidades como hipertensión, diabetes, enfermedad obstructiva pulmonar crónica y complicaciones cardiovasculares; sin embargo, son necesarios más estudios con mayor cantidad de pacientes para evitar sesgos (16). Wang et al. observaron en pacientes tratados con antivirales o inmunomoduladores, que los linfocitos LT CD8+ y LB se incrementaron, probablemente por el uso de corticoides y su efecto antiinflamatorio. En este mismo estudio se observó que los LT CD8+ tienden a predecir independientemente la gravedad y la eficacia al tratamiento por COVID-19 (15).
TORMENTA DE CITOCINAS EN LA COVID-19
Un incremento exacerbado de citocinas ante la presencia de virus que atacan el sistema respiratorio se define como tormenta citocinas. Varios estudios han demostrado que elevadas cantidades de citocinas proinflamatorias en el suero se asocian a la inflamación y al extenso daño pulmonar provocado por el SARS-CoV, MERS-CoV y recientemente, en SARS-CoV-2 se están encontrando más evidencias.
Contrariamente, existen bajos niveles de interferones tipo I, que normalmente forman parte de la respuesta inmune innata. Esto trae como consecuencia la supresión de respuestas cooperadoras Th1, lo que favorece la tipo Th2 (17). El incremento de citocinas inflamatorias en pacientes con COVID-19 ha sido demostrado no solo a nivel transcripcional (18), sino también a nivel proteico en muestras de 452 pacientes donde se encontró un incremento en suero del factor de necrosis tumoral alfa (TNF-α, por sus siglas en inglés), interleuquinas (IL)-2R, IL-6, IL-8 y IL-10 en pacientes con enfermedad grave comparados a los de curso no grave, lo que sugiere un posible rol en el desarrollo de la respuesta hiperinflamatoria de COVID-19 (16).
Un estudio con 41 pacientes con SARS-CoV-2 demostró que sus plasmas presentaban mayor cantidad de TNF-α, IL-1β, IL-1Ra entre otras citocinas, lo que demostraría el posible efecto de tormenta de citocinas (19); sin embargo, al existir también elevación de algunas citocinas antiinflamatorias (p. ej., IL-4, IL-10) se requiere más estudios para demostrarlo. Es conocido que IL-17 tiene efectos proinflamatorios sobre la inducción de IL-1β, IL-6 y TNF-α, quimiocinas y metaloproteinasas de la matriz. Además, IL-17 junto con IL-22 induce péptidos antimicrobianos en mucosas con los que contribuirían a la formación de un edema potencialmente mortal enriquecido con mucinas y fibrina, visto en el SARS-CoV-2 (20). El TNF-α y la IL-1 se producen en los pulmones de pacientes con COVID-19 y son fuertes inductores de hialurano sintetasa en células epiteliales CD31+, células epiteliales alveolares EpCAM+, y fibroblastos. La hialurano sintetasa puede reducir el hialurano, lo que minimizará la absorción de agua y la formación de una gelatina que invade el pulmón y que contribuye al estrés respiratorio (14).
Otro mecanismo que se activaría por la unión del ARN viral de SARS-CoV-2 al receptor tipo Toll (TLR-9) es la producción de IL-1β que se produce a través de la activación del inflamasoma. Esta citoquina causa no solo la inflamación pulmonar sino también la fiebre y la fibrosis (21).
MEJORA DEPENDIENTE DE ANTICUERPOS (ADE)
La ADE es un fenómeno conocido en virología y ocurre cuando los anticuerpos facilitan el ingreso viral a las células huésped. Es una forma alternativa que tienen algunos virus para infectar las células. En este caso, los anticuerpos primero se unirán al virus y luego a los receptores Fc de una IgG presentes en células inmunes con lo cual mediarán el ingreso del virus a estas células (22,23). Por tanto, la ADE promueve la ingesta celular de complejos virus-anticuerpo a través del receptor FcR u otros receptores, facilitando la infección viral, lo que a su vez promueve las respuestas inflamatorias y la persistente replicación viral en los pulmones de algunos pacientes (Figura 1) (24,25).
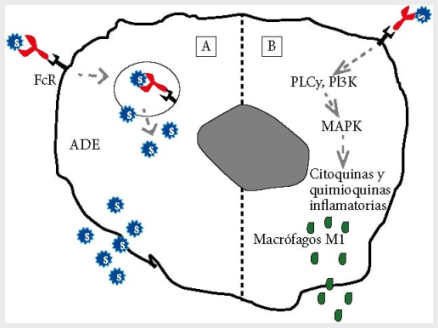
Figura 1. Mejora dependiente de anticuerpos (ADE). A. En determinados casos es posible que se produzcan anticuerpos que formen inmunocomplejos con los virus por lo general producto de respuestas secundarias. Estos anticuerpos mejorarían el ingreso viral a las células huésped que posean receptores Fc para el anticuerpo IgG. B. La ADE se produce también en células proinflamatorias como los macrófagos M1 induciendo la señalización de vías que producen la liberación de citocinas proinflamatorias. Leyenda: FcR= Receptor de Fragmento cristalizable de una inmunoglobulina IgG; PLC= Fosfolipasa C; PI3K= Fosfatidilinositol 3 kinasa; MAPK= Proteína quinasa activada por mitógeno; S= SARS-CoV-2.
Los estudios de los epítopes de la proteína S de los virus SARS-Cov y SARS-Cov-2 indican que existen regiones que no son comunes y que podrían explicar la aparición de la ADE, y que como SARS-CoV, estos anticuerpos también se generarían en pacientes recurrentemente expuestos en un lapso corto al SARS-CoV-2 (26,27). La presencia de una ADE deberá ser evaluada cuando se diseñen vacunas o se utilicen anticuerpos monoclonales contra la infección por SARS-CoV-2 (22,28). Dado que aún no existen estudios que expliquen los mecanismos sobre la respuesta inflamatoria en SARS-CoV-2, se postula que pueden ocurrir una ADE en dos estadios diferentes: uno en la respuesta primaria y el otro en la secundaria.
Luego de que ingresa el virus y antes de que aparezcan los primeros anticuerpos neutralizantes, la respuesta inflamatoria primaria es dirigida por una activa replicación viral, desregulación y respuestas antivirales del huésped, lo que puede incrementar la producción de citocinas y quimiocinas, y el daño celular por apoptosis o piroptosis. Se dice que la mayoría de los pacientes tolerarían este tipo de respuesta con una reducción de la carga viral o incluso eliminación completa del virus seguida por una atenuación de la inflamación; mientras que, en la respuesta inflamatoria secundaria, se inicia con una inmunidad adaptativa y producción de anticuerpos neutralizantes que pueden disminuir la carga viral; existiendo el problema de una ADE que puede disparar respuestas inflamatorias y causar severos daños pulmonares.
Un dato importante es que en el SARS-CoV el desarrollo de la enfermedad aguda coincide con la seroconversión de IgG antiviral en el 80% de pacientes. Otro posible mecanismo que acompaña a la infección es la ADE en los macrófagos (Figura 1) (24). En este caso, en modelos experimentales del SARS-CoV se ha demostrado que la ADE facilita la infección de macrófagos, pero el virus no logra replicarse y no altera el programa de producción de citocinas inflamatorias. En consecuencia, la hipótesis de una posible muerte masiva de células inmunes queda descartada (29,30). Es posible, en casos específicos, el uso de inmunoglobulinas intravenosas (IGIV) como estrategia terapéutica en pacientes con seroconversión temprana con el fin de inhibir la ADE mediada por receptor Fc y la producción de citocinas proinflamatorias por los macrófagos (31,32). También se aplica con la intención de neutralizar a los virus de pacientes infectados (33); sin embargo, se requieren evidencias más robustas ya que solo existen reportes combinados con antivirales sobre el efecto benéfico de esta inmunoterapia (34).
PERSPECTIVAS TERAPÉUTICAS CONTRA EL SARS-CoV-2
Para infectar una célula, el SARS-CoV-2 usa su proteína S, la cual está densamente glicosilada. La proteína S es una proteína trimérica de fusión clase I que existe en una conformación de prefusión metaestable que sufre un reordenamiento estructural sustancial para fusionar la membrana viral con la membrana de la célula huésped (35). Las investigaciones se están enfocando en la identificación de moléculas antivirales dirigidas a la proteína S y en su potencial inmunógeno.
Si bien, actualmente no existen terapias antivirales efectivas para la infección por SARS-CoV-2, es necesario que haya intervenciones de salud pública rápidas con anticuerpos monoclonales (ANM), antivirales o nuevas estrategias de vacunación. En este contexto de pandemia se debe evaluar rigurosamente la inmunoterapia basada en transferencia pasiva de suero o plasma convaleciente. Resulta claro que independientemente del tipo de tratamiento para combatir la COVID-19 se requerirán muchos meses para ensayar la eficacia in vitro e in vivo de las terapias antivirales, las que deberán ser rigurosamente diseñadas y monitoreadas. Es importante considerar que este virus tiene la ventaja de guardar relación filogenética con los virus del SARS-CoV y del MERS-CoV, lo que permitiría el uso de conocimientos previos para adaptar los modelos terapéuticos y de vacunas.
ANTICUERPOS MONOCLONALES PARA TRATAR COVID-19
Los ANM neutralizantes son potenciales herramientas terapéuticas que podrían ser dirigidos específicamente al dominio de unión al receptor (RBD, N318-V510) de la proteína S o también a la proteína receptora ACE2 de tal manera que en ambos casos se bloquearía el ingreso viral (36) (Figura 2). La estructura de los sitios de interacción entre el ectodominio de la proteína S del SARS-CoV-2 y el ANM ya se han obtenido por bioinformática (PyMOL) (5). Los ANM investigados en el SARS que pueden ser útiles para el SARS-CoV-2 son once y han sido evaluados in vitro e in vivo. De esos once anticuerpos, el ANM m396 ha demostrado una ligera interacción mientras que el CR3022 se unió potentemente con el RBD del SARS-CoV-2, lo que lo convierte en un potencial candidato terapéutico (37). Por tanto, se debe determinar la reactividad cruzada de todos los ANM producidos para el SARS-CoV con la proteína S del SARS-CoV-2, con lo que se podrían usar en ensayos clínicos. También se sugiere un cóctel de estos ANM que contrarresten los posibles cambios antigénicos en el virus (5).
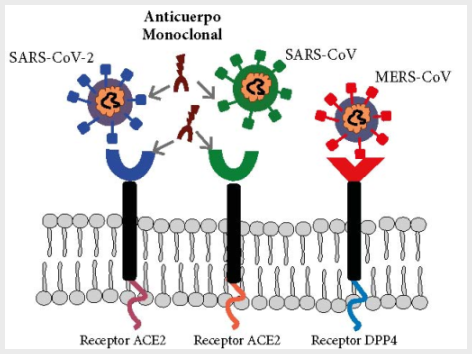
Figura 2. Anticuerpos monoclonales anti-SARS-CoV-2. Los virus SARS-CoV y SARS-CoV-2 reconocen la misma proteína receptora enzima convertidora de angiotensina 2 (ACE2, por sus siglas en inglés) sobre las células huésped que infectan; mientras que los virus MERS-CoV reconocen el receptor DPP4 (dipeptidil peptidasa-4) en las células huésped. La alta homología entre los virus SARS hace posible que se puedan utilizar los anticuerpos monoclonales en estudio para SARS-CoV en SARS-CoV-2, para intentar bloquear la proteína S y el receptor ACE2.
ANTIVIRALES CONTRA LA COVID-19
Dado que los LT Th17 pueden tener una participación en COVID-19, se postula la evaluación de varios inhibidores de su factor de transcripción RORgt (y RORα). Alternativamente, se propone el uso de inhibidores de JAK2 (por ej. fedratinib, usado en mielofibrosis) (38) para restringir la función inflamatoria de Th17. Debido a que la IL-6 y la IL-23 activan a STAT3 a través de JAK2, junto a estos inhibidores, la Administración de Alimentos y Medicamentos (FDA, por sus siglas en inglés) ha aprobado inhibidores de STAT3, que son útiles, pero podrían afectar la actividad de LB productores de IL-21. Estos inhibidores se podrían usar junto con interferones tipo I sin afectarlos, ya que estos utilizan JAK1 y TYK2 para activar STAT1 y STAT2 (20).
Dos grupos de drogas que pueden tener potencial acción contra el SARS-CoV-2 son los análogos de nucleósidos (AN) aprobados (favipiravir y ribavirin) y los experimentales (remdesivir y galidesivir). Los AN en forma de derivados de adenina o guanina se dirigen hacia la ARN polimerasa dependiente de ARN y bloquean la síntesis de ARN viral en una gran cantidad de virus, incluso los coronavirus humanos. De estos AN, remdesivir (GS5734), un profármaco fosforamidato de un derivado de adenina con potencial actividad sobre la transcriptasa reversa, tiene actividades de amplio espectro contra virus ARN como MERS y SARS, tanto en cultivos como en modelos animales. Recientemente se ha ensayado en células Vero E6 infectadas con SARS-CoV-2 y ha demostrado un EC50 (concentración media efectiva máxima) de 0,77 uM.
Un reporte del primer estadounidense de 34 años infectado evidenció mejoría clínica tras la administración de remdesivir, luego del intento fallido de otros fármacos, por lo que se concluye que este fármaco debe evaluarse clínica y rigurosamente (39,40). Tal vez sea este caso el que llevó al Instituto Nacional de Salud (NIH, por sus siglas en inglés) de los Estados Unidos a realizar dos ensayos clínicos fase III para evaluar este fármaco en pacientes con COVID-19. Se trata de estudios aleatorizados, controlados, doble ciego con 308 y 453 voluntarios para evaluar la eficacia y seguridad en pacientes con enfermedad respiratoria por COVID-19, leve o moderada (NCT042552664) y grave (NCT04257656). Los estudios se iniciaron en febrero y se deberán tener resultados en abril y mayo de 2020 (41,42). En China se realizó el ensayo LOTUS para investigar la eficacia y la seguridad de lopinavir/ritonavir (LPV/RTV, inhibidores de proteasas); sin embargo, no hubo diferencias en la mejora clínica (43).
La terapia antimalárica comenzó a mediados del siglo XVII gracias a misioneros jesuitas en el Perú, lo que llevó a obtener el alcaloide quinina por los franceses Pelletier y Caventou y luego a sintetizarse en los Estados Unidos como cloroquina (CQ) (44). Las perspectivas de fármacos como CQ e hidroxicloroquina (HCQ) son promisorias por los estudios in vitro y en animales, pero éstos fármacos requieren una sólida evidencia clínica para ser recomendados para su uso contra la COVID-19, como lo demandan varias cartas al editor sobre estos estudios (45–47). La CQ y la HCQ son aminoquinolinas, cuya diferencia radica en que la HCQ posee un grupo N-hidroxietil y la CQ, un grupo N-dietil (48).
Un estudio clínico francés con muchas deficiencias, como el tamaño de muestra, la falta de aleatorización, la deficiente selección de los grupos de estudios, la falta de descripción de efectos colaterales, entre otras, ha sido motivo de discusión en la comunidad científica. El estudio atribuye a la HCQ sulfato y además a la azitromicina la reducción de la carga viral desde el día seis en pacientes con COVID19; sin embargo, la evidencia no es lo suficientemente sólida (49). Sin bien, este estudio sirvió para que la FDA autorizara la prescripción de HCQ y CQ a pacientes con COVID-19, la comunidad científica reclama por ensayos clínicos con mejores datos y que apunten a un verdadero beneficio (50).
Los estudios in vitro indican que la CQ bloquea la infección por SARS-CoV-2 a bajas concentraciones micromolares (EC50 = 1,13 uM) y que existen registros de estudios clínicos (por ej., ChiCTR2000029939, ChiCTR2000029935, ChiCTR2000029899, entre otros) realizados en China, donde se demuestran la seguridad y la eficacia de la CQ o la HCQ en la neumonía asociada a la COVID-19. Estos estudios demostrarían que CQ fosfato lograría inhibir la exacerbación de la neumonía, mejoraría los hallazgos de las imágenes pulmonares, promovería la conversión negativa del virus y acortaría el curso de la enfermedad; además no se reportan efectos adversos graves en los pacientes recuperados (51).
Los mecanismos de acción propuestos para la CQ o la HCQ son similares, y muchos han sido evaluados in vitro y algunos modelos animales donde se ha demostrado que incrementan el pH intracelular, lo que disminuye la actividad lisosomal de las células presentadoras de antígenos con la consiguiente disminución de la activación y coestimulación de LT CD4+ y producción de IL-1, IL-6 y TNF-α. CQ y HCQ también evitan la unión de receptores TLR al RNAm con lo cual también disminuye la producción de citocinas proinflamatorias (52).
Desde el mes de marzo de 2020 se viene ejecutando un ensayo clínico fase III de la Universidad de Minesota avalado por NIH (NCT04308668) para evaluar la profilaxis posexposición y terapia preventiva de HCQ en SARS-CoV-2. Es un estudio aleatorizado, contra placebo que incorporará 3000 participantes asintomáticos y sintomáticos de los Estados Unidos y Canadá. Se espera obtener resultados en mayo del presente año (53).
Los interferones son agentes antivirales de amplio espectro y han demostrado actividad contra el SARS-CoV y el MERS-CoV. La ribavirina combinada con LPV/RTV se ha usado contra el SARS-CoV; sin embargo, contra el SARS-CoV-2 in vitro requiere una alta EC50=109,5 uM; por lo que se está realizando un estudio clínico en China (ChiCTR2000029387) de ribavirina, LPV/RTV y la combinación de estos (43). Adicionalmente se ha determinado que remdesivir e IFN-β tienen mayor actividad antiviral in vitro que LPV/RTV (54).
VACUNAS CONTRA LA COVID-19
La rápida necesidad de vacunas contra la COVID-19 ha obligado a hacer uso de la proteómica para buscar antígenos exclusivos del patógeno en la proteína S. Gracias a la bioinformática, se han podido identificar 933 pentapéptidos ausentes en el proteoma humano, de los cuales 107 péptidos se encuentran alrededor de la proteína S y de éstos 66 péptidos son más inmunógenos y se pueden usar en la elaboración de una vacuna (55). La OMS cuenta hasta con 52 alternativas de candidatos a vacunas entre plataformas basadas en proteínas, ARN, ADN, vectores no replicantes, vectores replicantes, virus inactivados, virus atenuados y partículas tipo virus. De todos estos prospectos de vacuna, solo las vacunas constituidas por ARN y por vector no replicante han iniciado estudios de seguridad en humanos (56,57).
La actual epidemia de la COVID-19 demanda rapidez, producción a gran escala y distribución de una vacuna. Se debe mirar hacia las vacunas basadas en ácidos nucleicos como las vacunas de ARNm, debido a que son confiables para aplicaciones de respuesta rápida, inducen respuestas inmunes ampliamente protectoras y tienen procesos de manufactura rápidos y flexibles. Las vacunas de ARNm mimetizan una infección viral al expresar antígenos de la vacuna in situ, lo que resulta en la inducción de respuestas inmune humoral y de LT CD8+. Adicionalmente, este tipo de vacunas pueden estimular la inmunidad innata, ya que pueden ser reconocidas por receptores, como TLR, que permiten la maduración de células presentadoras de antígenos encargadas de mejorar la inmunidad adaptativa. Una ventaja de las vacunas de ARNm es que no generarán partículas infecciosas ni se integrarán al genoma de las células huésped. Una vez que se identifica el antígeno más inmunogénico del patógeno, se secuencia el gen, se sintetiza y se clona en un plásmido de ADN. Luego el ARNm es transcrito in vitro y se vacuna al paciente utilizando como vehículo, por ejemplo, a nanopartículas lipídicas (NPL) (Figura 3) (58).

Figura 3. Candidato vacunal de ARNm contra el SARS-CoV-2. Las NPL contienen el ARNm con la secuencia de un fragmento de la proteína S son inoculadas IM (A). Las NPL son reconocidas y endocitadas por las CPA (B). El ARNm se transcribe a proteínas (C). Las proteínas siguen la vía de presentación de antígenos endógena (MHC-I) (D) o la exógena (MHC-II) (E). Las vías endógena y exógena activan a los linfocitos T CD8+ (citotóxicos) y CD4+ (cooperadores), lo que estimula los mecanismos de respuesta celular (F) y humoral (G), respectivamente. Adicionalmente, el material genético de la vacuna podría ser reconocido por los receptores tipo Toll (TLR) endógenos y estimular la síntesis de citocinas (H). Leyenda: NPL = nanopartículas lipídicas; ARNm = ácido ribonucleico mensajero; IM = intramuscularmente; CPA= células presentadoras de antígeno; MHC I y II = complejo mayor de histocompatibilidad.
En el mes de marzo de 2020, el NIH, con el apoyo del Instituto Nacional de Alergias y Enfermedades Infecciosas (NIAID, por sus siglas en inglés) de los Estados Unidos, inició el ensayo clínico NCT04283461 sobre la seguridad y la inmunogenicidad de la vacuna mARN-1273 para profilaxis de la infección por SARS CoV-2. Se trata de un ensayo de fase I, abierto, de rangos de dosis en 45 hombres y mujeres no embarazadas entre 18 y 55 años sanos. La vacuna mARN-1273 ha sido fabricada por Moderna TX, Inc. Se trata de una vacuna constituida por ARNm encapsulado en nanopartículas lípidicas (NPL) que codifica la proteína de fusión S del SARS-CoV-2. La vacuna será administrada intramuscularmente en los días 1 y 29 en tres cohortes (25 ug, 100 ug y 250 ug). El seguimiento será por 12 meses, luego del segundo refuerzo y se evaluará la inducción de anticuerpos IgG mediante ELISA en el día 57 (59).
En marzo de este año también se inició el ensayo de la vacuna CanSino por el Instituto de Biotecnología Militar de China usando adenovirus-5 (Ad5) en versión no replicante, es un vector portador del gen de la proteína S de SARS-CoV-2. Existe la preocupación de la comunidad científica debido a que algunas personas pueden tener inmunidad a Ad5, lo que limitaría la distribución del gen de la proteína S a células humanas, como ha sucedido con vacunas para HIV basadas en Ad5 (56,60).
CONCLUSIONES
La pandemia por SARS-CoV-2 se diseminó a través de la trasmisión humano-humano, y, aunque el virus identifica los receptores ACE2 en células epiteliales de varios órganos, es posible que existan aumentos de infecciones dependientes de anticuerpos (ADE). Este aspecto deberá ser considerado cuando se evalúen vacunas o se utilicen anticuerpos monoclonales.
La respuesta inmune es uno de los factores clave que condiciona la capacidad de respuesta de los infectados por SARS-CoV-2 y es claro que, en fases graves o con comorbilidades, esta respuesta suele disminuir, como lo evidencia la marcada linfopenia y la hiperinflamación. También ha sido evidente que la edad cumple un rol importante relacionado a la inmunosenescencia; sin embargo, entre los jóvenes las reinfecciones podrían agravar su enfermedad.
Actualmente no existen antivirales ni vacunas contra la COVID-19; sin embargo, existen ensayos clínicos de seguridad y eficacia para fármacos y vacunas que son promisorios para tratar la COVID-19. La medida más efectiva para evitar la trasmisión del SARS-CoV-2 ha sido el aislamiento social exhaustivo. En el Perú, el Gobierno ha hecho cumplir la medida priorizando la vida del ciudadano, limitando el crecimiento exponencial del número de infectados, en el afán de reducir significativamente la pérdida de vidas. Los tratamientos antivirales y las medidas preventivas serán determinantes para retornar a la vida normal, aunque manteniendo las medidas de bioseguridad requeridas.
REFERENCIAS BIBLIOGRÁFICAS
1. Xu Z, Shi L, Wang Y, Zhang J, Huang L, Zhang C, et al. Pathological findings of COVID-19 associated with acute respiratory distress syndrome. Lancet Respir Med. 2020;2600(20):19–21. doi: 10.1016/S2213-2600(20)30076-X.
2. WHO. World Health Organization [Internet]. Situation reports 87. 2020 [citado el 16 de abril de 2020]. p. 1. Disponible en: https://www.who.int/emergencies/diseases/novel-coronavirus-2019/situation-reports/.
3. Centro Nacional de Epidemiología Prevención y Control de Enfermedades - MINSA. Sala situacional COVID-19 Perú [Internet]. 2020 [citado el 17 de abril de 2020]. p. 1. Disponible en: https://covid19.minsa.gob.pe/sala_situacional.asp.
4. Zou X, Chen K, Zou J, Han P, Hao J, Han Z. Single-cell RNA-seq data analysis on the receptor ACE2 expression reveals the potential risk of different human organs vulnerable to 2019-nCoV infection. Front Med [Internet]. 2020;1–8. doi: 10.1007/s11684-020-0754-0.
5. Shanmugaraj B, Siriwattananon K, Wangkanont K, Phoolcharoen W. Perspectives on monoclonal antibody therapy as potential therapeutic intervention for Coronavirus disease-19 (COVID-19). Asian Pac J Allergy Immunol. 2020;10–8. doi: 10.12932/AP-200220-0773.
6. Gao QY, Chen YX, Fang JY. 2019 Novel coronavirus infection and gastrointestinal tract. J Dig Dis. 2020;21:125–6.
7. Li G, De Clercq E. Therapeutic options for the 2019 novel coronavirus (2019-nCoV). Nat Rev Drug Discov [Internet]. 2020;19(3):149–50. doi: 10.1038/d41573-020-00016-0.
8. Chen Y, Peng H, Wang L, Zhao Y, Zeng L, Gao H, et al. Infants Born to Mothers With a New Coronavirus (COVID-19). Front Pediatr [Internet]. 2020;8:104. doi: 10.3389/fped.2020.00104.
9. Guo L, Ren L, Yang S, Xiao M, Chang D, Yang F, et al. Profiling Early Humoral Response to Diagnose Novel Coronavirus Disease (COVID-19). Clin Infect Dis. 2020; pii: ciaa310. doi: 10.1093/cid/ciaa310. [Epub ahead of print].
10. Wu Z, McGoogan JM. Characteristics of and Important Lessons from the Coronavirus Disease 2019 (COVID-19) Outbreak in China: Summary of a Report of 72314 Cases from the Chinese Center for Disease Control and Prevention. JAMA. 2020; doi: 10.1001/jama.2020.2648. [Epub ahead of print].
11. Liu Z, Xiao X, Wei X, Li J, Yang J, Tan H, et al. Composition and divergence of coronavirus spike proteins and host ACE2 receptors predict potential intermediate hosts of SARS‐CoV‐2. J Med Virol. 2020; doi: 10.1002/jmv.25726. [Epub ahead print].
12. Ou X, Liu Y, Lei X, Li P, Mi D, Ren L, et al. Characterization of spike glycoprotein of SARS-CoV-2 on virus entry and its immune cross-reactivity with SARS-CoV. Nat Commun [Internet]. 2020;11(1):1620. doi: 10.1038/s41467-020-15562-9.
13. Meo SA, Alhowikan AM, Khlaiwi TAL, Meo IM, Halepoto DM, Iqbal M, et al. Novel coronavirus 2019-nCoV: Prevalence, biological and clinical characteristics comparison with SARS-CoV and MERS-CoV. Eur Rev Med Pharmacol Sci. 2020;24(4):2012–9. doi: 10.26355/eurrev_202002_20379.
14. Shi Y, Wang Y, Shao C, Huang J, Gan J, Huang X, et al. COVID-19 infection: the perspectives on immune responses. Cell Death Differ. 2020; doi: 10.1038/s41418-020-0530-3. [Epub ahead of print].
15. Wang F, Nie J, Wang H, Qiu Z, Yong X, Liping D, et al. Characteristics of peripheral lymphocyte subset alteration in COVID-19 pneumonia. J Infect Dis. 2020; doi: 10.1093/infdis/jiaa150. [Epub ahead of print].
16. Qin C, Zhou L, Hu Z, Zhang S, Yang S, Tao Y, et al. Dysregulation of immune response in patients with COVID-19 in Wuhan, China. J Chem Inf Model. 2020; doi: 10.1093/cid/ciaa248. [Epub ahead of print].
17. Sarzi-Puttini P, Giorgi V, Sirotti S, Marotto D, Ardizzone S, Rizzardini G, et al. COVID-19, cytokines and immunosuppression: what can we learn from severe acute respiratory syndrome? Clin Exp Rheumatol. 2020;38(2):337–42.
18. Xiong Y, Liu Y, Cao L, Wang D, Guo M, Guo D, et al. Transcriptomic Characteristics of Bronchoalveolar Lavage Fluid and Peripheral Blood Mononuclear Cells in COVID-19 Patients. Emerg Microbes Infect. 2020;9(1):761–70. doi: 10.1080/22221751.2020.1747363.
19. Huang C, Wang Y, Li X, Ren L, Zhao J, Hu Y, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. Lancet. 2020;395(10223):497–506. doi: 10.1016/S0140-6736(20)30183-5.
20. Wu D, Yang XO. TH17 Responses in Cytokine Storm of COVID-19: An Emerging Target of JAK2 Inhibitor Fedratinib. J Microbiol Immunol Infect. 2020; doi: 10.1016/j.jmii.2020.03.005. [Epub ahead of print].
21. Conti P, Ronconi G, Caraffa A, Gallenga C, Ross R, Frydas I, et al. Induction of pro-inflammatory cytokines (IL-1 and IL-6) and lung inflammation by Coronavirus-19 (COVI-19 or SARS-CoV-2): anti-inflammatory strategies. J biol Regul Homeost Agents. 2020;32(2). doi: 10.23812/CONTI-E. [Epub ahead print].
22. Wan Y, Shang J, Sun S, Tai W, Chen J, Geng Q, et al. Molecular Mechanism for Antibody-Dependent Enhancement of Coronavirus Entry. J Virol. 2019;94(5):1–15. doi: 10.1128/JVI.02015-19.
23. Peeples L. News Feature: Avoiding pitfalls in the pursuit of a COVID-19 vaccine. Proc Natl Acad Sci U S A. 2020;117(15):8218-8221. doi: 10.1073/pnas.2005456117.
24. Fu Y, Cheng Y, Wu Y. Understanding SARS-CoV-2-Mediated Inflammatory Responses: From Mechanisms to Potential Therapeutic Tools. Virol Sin. 2020; doi: 10.1007/s12250-020-00207-4. [Epub ahead of print].
25. Jin Y, Yang H, Ji W, Wu W, Chen S, Zhang W. Virology, Epidemiology, Pathogenesis, and Control of COVID-19. Viruses. 2020;12(4):1–17. doi: 10.3390/v12040372.
26. Tetro JA. Is COVID-19 Receiving ADE From Other Coronavirus? Microbes Infect. 2020;22(2):72–3. doi: 10.1016/j.micinf.2020.02.006.
27. Tilocca B, Soggiu A, Musella V, Britti D, Sanguinetti M, Urbani A, et al. Molecular basis of COVID-19 relationships in different species: a one health perspective. Microbes Infect [Internet]. 2020; doi: 10.1016/j.micinf.2020.03.002. [Epub ahead of print].
28. Ahn D-G, Shin H-J, Kim M-H, Lee S, Kim H-S, Myoung J, et al. Current Status of Epidemiology, Diagnosis, Therapeutics, and Vaccines for Novel Coronavirus Disease 2019 (COVID-19). J Microbiol Biotechnol. 2020;30(3):313–24. doi: 10.4014/jmb.2003.03011.
29. Yip M, Leung H, Li P, Cheung C, Dutry I, Li D, et al. Antibody-dependent enhancement of SARS coronavirus infection and its role in the pathogenesis of SARS. Hong Kong Med J. 2016;3(4):25–31.
30. Ricke DO, Malone RW. Medical Countermeasures Analysis of 2019-nCoV and Vaccine Risks for Antibody- dependent Enhancement (ADE) [Internet]. Preprint. 2009 [citado el 17 de abril de 2020]. p. 1–18. Disponible en: https://www.preprints.org/manuscript/202003.0138/v1.
31. Ferro F, Elefante E, Baldini C, Bartoloni E, Puxeddu I, Talarico R, et al. COVID-19: the new challenge for rheumatologists. Clin Exp Rheumatol. 2020;38(2):175–80.
32. Rismanbaf A. Potential Treatments for COVID-19; a Narrative Literature Review. Arch Acad Emerg Med. 2020;8(1):e29.
33. Jawhara S. Could Intravenous Immunoglobulin Collected from Recovered Coronavirus Patients Protect against COVID-19 and Strengthen the Immune System of New Patients? Int J Mol Sci. 2020;21(7):2272. doi: 10.3390/ijms21072272.
34. Shen C, Wang Z, Zhao F, Yang Y, Li J, Yuan J, et al. Treatment of 5 Critically Ill Patients With COVID-19 With Convalescent Plasma. JAMA. 2020; doi: 10.1001/jama.2020.4783. [Epub ahead of print].
35. Wrapp D, Wang N, Corbett KS, Goldsmith JA, Hsieh C-L, Abiona O, et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science. 2020;367(6483):1260–3. doi: 10.1126/science.abb2507.
36. Zhou G, Zhao Q. Perspectives on therapeutic neutralizing antibodies against the Novel Coronavirus SARS-CoV-2. Int J Biol Sci. 2020;16(10):1718–23. doi: 10.7150/ijbs.45123.
37. Tian X, Li C, Huang A, Xia S, Lu S, Shi Z, et al. Potent binding of 2019 novel coronavirus spike protein by a SARS coronavirus-specific human monoclonal antibody. Emerg Microbes Infect. 2020;9(1):382–5. doi: 10.1080/22221751.2020.1729069.
38. Blair HA. Fedratinib: First Approval. Drugs. 2019;79(15):1719–25. doi: 10.1007/s40265-019-01205-x.
39. Holshue ML, DeBolt C, Lindquist S, Lofy KH, Wiesman J, Bruce H, et al. First case of 2019 novel coronavirus in the United States. N Engl J Med. 2020;382(10):929–36. doi: 10.1056/NEJMoa2001191.
40. Guo Y-R, Cao Q-D, Hong Z-S, Tan Y-Y, Chen S-D, Jin H-J, et al. The origin, transmission and clinical therapies on coronavirus disease 2019 (COVID-19) outbreak – an update on the status. Mil Med Res. 2020;7(1):1–10. doi: 10.1186/s40779-020-00240-0.
41. NIH NCT04252664. ClinicalTrials.gov [Internet]. NIH US National Library of Medicine. 2020 [citado el 16 de abril 2020]. p. 1. Disponible en: https://clinicaltrials.gov/ct2/show/NCT04252664.
42. NIH N. ClinicalTrials.gov [Internet]. NIH US National Library of Medicine. 2020 [citado el 31 de marzo de 2020]. p. 1. Disponible en: https://clinicaltrials.gov/ct2/show/NCT04257656.
43. Lu C-C, Chen M-Y, Chang Y-L. Potential therapeutic agents against COVID-19: What we know so far. J Chinese Med Assoc [Internet]. 2020; doi: 10.1097/JCMA.0000000000000318. [Epub ahead of print].
44. Scuccimarri R, Sutton E, Fitzcharles M. Hydroxychloroquine: a potential ethical dilemma for rheumatologists during the COVID-19 pandemic. J Rheumatol [Internet]. 2020; doi: 10.3899/jrheum.200369. [Epub ahead of print].
45. Yao X, Ye F, Zhang M, Cui C, Huang B, Niu P, et al. In Vitro Antiviral Activity and Projection of Optimized Dosing Design of Hydroxychloroquine for the Treatment of Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2). Clin Infect Dis. 2020; doi: 10.1093/cid/ciaa237. [Epub ahead print].
46. Guastalegname M, Vallone A. Could chloroquine /hydroxychloroquine be harmful in Coronavirus Disease 2019 (COVID-19) treatment? Clin Infect Dis. 2020; doi: 10.1093/cid/ciaa321. [Epub ahead print].
47. Mégarbane B. Chloroquine and hydroxychloroquine to treat COVID-19: between hope and caution. Clin Toxicol (Phila) [Internet]. 2020; doi: 10.1080/15563650.2020.1748194. [Epub ahead of print].
48. Sahraei Z, Shabani M, Shokouhi S, Saffaei A. Aminoquinolines Against Coronavirus Disease 2019 ( COVID-19 ): Chloroquine or Hydroxychloroquine. Int J Antimicrob Agents [Internet]. 2020; doi: 10.1016/j.ijantimicag.2020.105945. [Epub ahead of print].
49. Gautret P, Lagier J-C, Philippe P, Hoang VT, Meddeb L, Mailhe M, et al. Hydroxychloroquine and azithromycin as a treatment of COVID-19: results of an open-label non-randomized clinical trial. Int J Antimicrob Agents [Internet]. 2020; doi: 10.1016/j.ijantimicag.2020.105949. [Epub ahead print Mar 20, 2020].
50. Lenzer J. Covid-19 : US gives emergency approval to hydroxychloroquine despite lack of evidence. BMJ [Internet]. 2020; 369:m1335. doi: 10.1136/bmj.m1335.
51. Gao J, Tian Z, Yang X. Breakthrough : Chloroquine phosphate has shown apparent efficacy in treatment of COVID-19 associated pneumonia in clinical studies. Biosci Trends [Internet]. 2020; doi: 10.5582/bst.2020.01047. [Epub ahead print Mar 16, 2020].
52. Zhou D. COVID-19: a recommendation to examine the effect of hydroxychloroquine in preventing infection and progression. J Antimicrob Chemother [Internet]. 2020; doi: 10.1093/jac/dkaa114. [Epub ahead print].
53. NIH N. Post-exposure Prophylaxis / Preemptive Therapy for SARS-Coronavirus-2 (COVID-19 PEP) [Internet]. NIH US National Library of Medicine. 2020. Disponible en: https://clinicaltrials.gov/ct2/show/NCT04308668.
54. Zhai P, Ding Y, Wu X, Long J, Zhong Y, Li Y. The epidemiology, diagnosis and treatment of COVID-19. Int J Antimicrob Agents [Internet]. 2020; 105955. doi: 10.1016/j.ijantimicag.2020.105955. [Epub ahead of print].
55. Lucchese G. Epitopes for a 2019-nCoV vaccine. Cell Mol Immunol [Internet]. 2020; doi: 10.1038/s41423-020-0377-z. [Epub ahead print].
56. Cohen J. Vaccine designers take first shots at COVID-19. Science [Internet]. 2020;368(6486):14–16. doi: 10.1126/science.368.6486.14.
57. Lurie N, Saville M, Hatchett R, Halton J. Developing Covid-19 Vaccines at Pandemic Speed. N Engl J Med [Internet]. 2020; doi: 10.1056/NEJMp2005630. [Epub ahead of print].
58. Maruggi G, Zhang C, Li J, Ulmer JB, Yu D. mRNA as a Transformative Technology for Vaccine Development to Control Infectious Diseases. Mol Ther. 27(4):757-772. doi: 10.1016/j.ymthe.2019.01.020.
59. NIH NCT04283461. ClinicalTriasl.gov [Internet]. NIH US National Library of Medicine. 2020 [citado 1 de abril 2020]. p. 1. Disponible en: https://clinicaltrials.gov/ct2/show/study/NCT04283461.
60. Agrawal S, Goel AD, Gupta N. Emerging prophylaxis strategies against COVID-19. Monaldi Arch Chest Dis [Internet]. 2020;90(1). doi: 10.4081/monaldi.2020.1289.
Financiamiento: Autofinanciado.
Citar como: Lozada-Requena I, Núñez Ponce C. COVID-19: respuesta inmune y perspectivas de intervenciones terapéuticas. Rev Peru Med Exp Salud Publica. 2020;37(2):312-9. doi: https://doi.org/10.17843/rpmesp.2020.372.5490
Correspondencia: Iván Lozada-Requena; ivan.lozada@upch.pe
Contribuciones de autoría: ILR participó en la concepción de la idea, el análisis, la redacción y la revisión del artículo. CNP participó en la redacción y revisión del artículo.
Conflicto de interés: Los autores declaran que no existen conflictos de interés en la publicación del artículo.
Recibido: 05/04/2020
Aprobado: 22/04/2020
En línea: 01/05/2020