Milagros Zavaleta Apestegui
Carlos Raúl Sevilla-Andrade
Julia Piscoya Sara
Claudia Villoslado Espinoza
William Vicente Taboada
Edgar Gonzales-Escalante
10.17843/rpmesp.2020.374.5832
ORIGINAL BREVE
Enterobacterales productores de betalactamasas de espectro extendido portadores del gen mcr-1 en Lima, Perú
Extended-spectrum beta-lactamase-producing enterobacterales carrying the mcr-1 gene in Lima, Peru
Katherine Yauri-Condor ![]() 1, Tecnóloga/o médica/o
1, Tecnóloga/o médica/o
Milagros Zavaleta Apestegui ![]() 1, bióloga molecular
1, bióloga molecular
Carlos Raúl Sevilla-Andrade ![]() 1,2, Tecnóloga/o médica/o
1,2, Tecnóloga/o médica/o
Julia Piscoya Sara ![]() 2, médica/o cirujana/o; magíster en Epidemiología
2, médica/o cirujana/o; magíster en Epidemiología
Claudia Villoslado Espinoza ![]() 3, médica/o cirujana/o; especialista en Patología Clínica
3, médica/o cirujana/o; especialista en Patología Clínica
William Vicente Taboada ![]() 3,4, médica/o cirujana/o; especialista en Patología Clínica
3,4, médica/o cirujana/o; especialista en Patología Clínica
Edgar Gonzales-Escalante ![]() 1,5, Tecnóloga/o médica/o; magíster en Microbiología
1,5, Tecnóloga/o médica/o; magíster en Microbiología
1 Centro de
Investigaciones Tecnológicas, Biomédicas y Medioambientales - CITBM,
Universidad Nacional Mayor de San Marcos, Lima, Perú.
2 Instituto de Medicina Tropical «Daniel A. Carrión»,
Departamento Académico de Microbiología Médica, Facultad de Medicina,
Universidad Nacional Mayor de San Marcos, Lima, Perú.
3 Instituto Nacional de Enfermedades Neoplásicas, Lima, Perú.
4 Laboratorios Unilabs, Lima, Perú.
5 Laboratorio de Resistencia Bacteriana, Instituto de
Investigaciones en Bacteriología y Virología Molecular (IBaViM), Facultad de
Farmacia y Bioquímica, Universidad de Buenos Aires, Buenos Aires, Argentina.
RESUMEN
Se analizó la presencia del gen mcr-1 en 165 enterobacterales productores de betalactamasas de espectro extendido (EP-BLEE) recuperados en 2017 de sangre (40), orina (57), secreciones respiratorias bajas (12) e hisopados rectales (56) de pacientes hospitalizados en el Instituto Nacional de Enfermedades Neoplásicas (Perú). La identificación y la susceptibilidad antimicrobiana se determinaron por el sistema automatizado Phoenix M50; la resistencia a colistina por Colistin Agar-Spot (CAS); la detección de mrc-1 por el método fenotípico de predifusión de colistina e inhibición con EDTA (CPD-E) y por reacción en cadena de la polimerasa (PCR, por sus siglas en inglés). De los 165 EP-BLEE 25 fueron positivos para mcr-1 por el método CPD-E y se confirmó por PCR. Por el método CAS, 20/165 fueron resistentes a colistina. Además, mostraron resistencia a las fluoroquinolonas y a la gentamicina, y permanecieron sensibles a la amikacina; dos aislamientos presentaron metalocarbapenemasas. La obtención de datos sobre la resistencia a antimicrobianos considerados de última línea (colistina) es crucial para establecer medidas para su control.
Palabras clave: Enterobacteriaceae; beta-Lactamasas; Colistina; Farmacorresistencia Microbiana. (Fuente: DeCS BIREME).
ABSTRACT
We analyzed the presence of the mcr-1 gene in 165 extended-spectrum beta-lactamase-producing enterobacterales (ESBL-PE) obtained during 2017, from blood (40), urine (57), lower respiratory secretions (12) and rectal swabs (56) of patients hospitalized in the Instituto Nacional de Enfermedades Neoplásicas (Peru). Antimicrobial identification and susceptibility were determined by the Phoenix M50 automated system; colistin resistance by Colistin Agar-Spot (CAS); mrc-1 detection by colistin pre-diffusion and inhibition with EDTA test (CPD-E) and by polymerase chain reaction (PCR). We found that from the 165 ESBL-PE, 25 were positive for mcr-1 by the CPD-E method and confirmed by PCR. Colistin resistance was found in 20/165 by using the CAS method. Additionally, they showed resistance to fluoroquinolones and gentamicin, while remaining sensitive to amikacin; two isolates presented metallo-carbapenemases. Obtaining data on resistance to last-line antimicrobials (colistin) is crucial to establish measures for its control.
Keywords: Enterobacteriaceae; beta-Lactamases; Colistin; Drug Resistance, Microbial. (Source: MeSH NLM)
INTRODUCCIÓN
La diseminación mundial de bacterias gramnegativas multidrogorresistentes (MDR) y extremadamente resistentes a los antimicrobianos (XDR), incluidos los enterobacterales productores de carbapenemasas (EPC), ha llevado a la reinserción de colistina como terapia de último recurso; este antibiótico interactúa directamente con el lipopolisacárido de la membrana externa (1). Los principales mecanismos de resistencia implican la modificación del lípido A, mediados por mutaciones en genes del sistema regulador PhoPQ-PmrAB, incluso durante el tratamiento en microorganismos clínicamente relevantes como Klebsiella pneumoniae (2,3). En 2015, se describió un mecanismo de resistencia a la colistina mediada por plásmidos (PMCR, por sus siglas en inglés), relacionado con el gen mcr-1 (Mobile Colistin Resistance) entre enterobacterales aislados de animales y humanos en China (4), este codifica una fosfoetanolamina transferasa que modifica el blanco de colistina, mediante la adición de fosfoetanolamina, lo que reduce la afinidad por la colistina (5).
Diversos estudios han demostrado una distribución mundial de mcr1, especialmente en Escherichia coli, y una presencia ocasional en otras especies bacterianas (6). Al igual que con otros genes de resistencia, se han detectado diferentes variantes alélicas de mcr-1 (mcr-2, mcr-3, mcr-4, mcr-5, mcr-6, mcr-7, mcr-8, mcr-9 y mcr-10) (5, 6). En América del Sur, se ha reportado la presencia de genes mcr-1 en varios países, a partir de aislamientos de humanos, animales y alimentos (7-9). En 2016, la Organización Mundial de la Salud (OMS) recomendó implementar y fortalecer la vigilancia e investigación epidemiológica de la PMCR (10). En el Perú, hasta el momento se ha informado la presencia de mcr-1 en aislamientos de E. coli de origen clínico (11,12).
En ese sentido, como parte del proyecto «Vigilancia epidemiológica de bacterias resistentes en infecciones asociadas a la atención en salud», se plantea este estudio con el objetivo de conocer los enterobacterales productores de betalactamasa de espectro extendido (EP-BLEE) portadores del gen mcr-1 en el Instituto Nacional de Enfermedades Neoplásicas (INEN) del Perú.
|
MENSAJES CLAVE |
|
Motivación para realizar el estudio: La resistencia a los fármacos considerados de última línea frente a bacterias multidrogorresistentes (MDR) es motivo de preocupación. Es necesario investigar la resistencia a la colistina en enterobacterales productores de betalactamasa de espectro extendido (EP-BLEE) para conocer la realidad de este tipo de microrganismos en nuestro medio. Principales hallazgos: De los aislamientos de EP-BLEE, el 15,2% fueron positivos para el gen mcr-1, presentaron resistencia acompañante a fluoroquinolonas y gentamicina, y permanecieron sensibles a la amikacina; dos de estos aislamientos eran productores de metalocarbapenemasas. Implicancias: Los resultados de este estudio muestran la importancia del relevamiento de datos sobre la resistencia a la colistina mediada por plásmidos en MDR, lo cual respalda la necesidad de la implementación de estudios de epidemiología molecular para su control y se evite su diseminación en el ámbito hospitalario. |
EL ESTUDIO
Se realizó un estudio descriptivo. Entre enero y diciembre de 2017, se recolectaron 165 aislamientos consecutivos únicos de EP-BLEE, entre Escherichia coli (112), Klebsiella pneumoniae (41), Enterobacter cloacae (5), Proteus mirabilis (4), Klebsiella oxytoca (2) y Klebsiella ozaneae (1), recuperados de muestras de sangre (40), orina (57), secreciones respiratorias bajas (12) e hisopados rectales (56) de pacientes hospitalizados en el INEN.
La identificación y susceptibilidad antimicrobiana se realizó con el sistema automatizado Phoenix M50 y la confirmación de BLEE con el sistema BD-Expert (BD Diagnostics, Sparks, MD), la interpretación se realizó siguiendo las recomendaciones del Clinical and Laboratory Standards Institute (CLSI) (13). La detección de la resistencia a la colistina se realizó por el método de tamizaje Colistin Agar-Spot (CAS) (Agar Mueller-Hinton [Merck, Alemania]; sulfato de colistina sulfato [Sigma-Aldrich, Alemania]), desarrollado por el Servicio de Antimicrobianos, INEI ANLIS «Dr. Carlos G. Malbrán» (14). Además, se realizó el método fenotípico de predifusión de colistina e inhibición con el ácido etilendiaminotetraacético (EDTA) (CPD-E) (Agar Mueller-Hinton [Merck, Alemania]; discos de colistina 10 µg [Oxoid, Inglaterra]; discos de EDTA/SMA 372/900 µg [Britania, Argentina]), descrito por Yauri et al. (15), para la detección del gen mcr.
Se usó ADN bacteriano total como molde para la detección molecular, y esta se llevó a cabo en el laboratorio de Epidemiología Molecular y Genética del Instituto de Medicina Tropical «Daniel A. Carrión» del Centro de Investigaciones Tecnológicas, Biomédicas y Medioambientales - CITBM, de la Universidad Nacional Mayor de San Marcos. Para la identificación de la presencia de genes de resistencia (mcr-1, blaCTX-M, blaNDM, y blaKPC) se utilizó la reacción en cadena de la polimerasa (PCR, por sus siglas en inglés) como se ha descrito anteriormente (4,16, 17) (Figura 1).
*CPD-E: predifusión de colistina e inhibición con EDTA.
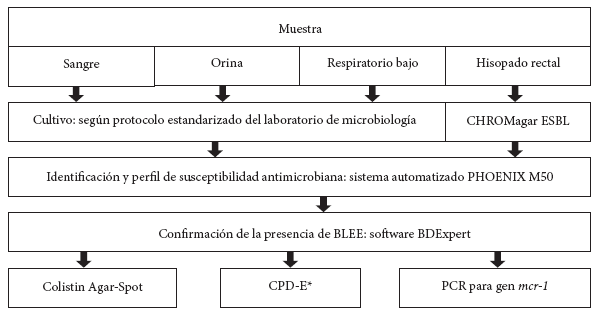
Figura 1. Flujograma de la detección de enterobacterales productoras de betalactamasas de espectro extendido portadoras del gen mcr-1.
El presente reporte se ha desarrollado como parte del estudio de los Círculos de Investigación en Ciencia y Tecnología del Fondo Nacional de Desarrollo Científico, Tecnológico y de Innovación Tecnológica (FONDECYT), aprobado por el Departamento de Investigación del INEN. El estudio sigue los lineamientos de las buenas prácticas y la ética en investigación biomédica. Se reportan frecuencias absolutas y relativas de las variables de interés. Se utilizó el programa Microsoft Excel para el análisis descriptivo.
HALLAZGOS
De los 165 aislamientos de EP-BLEE, 25 (15,2%) dieron positivo por el gen mcr-1; 20 (12,1%) fueron resistentes a la colistina por el método de tamizaje CAS. La detección fenotípica del mcr con el método CPD-E tuvo 100% de correlación con el método genotípico (25 positivos). La presencia del gen mcr-1, según especie, se dio de la manera siguiente: E. coli (18), K. pneumoniae (4), E. cloacae (2) y K. oxytoca (1). Según el tipo de muestra, el mcr-1 se detectó más en los aislamientos provenientes de hisopados rectales (11/25) y en la sangre (9/25) (Tabla 1).
Tabla 1. Distribución de aislamientos de enterobacterales productores de betalactamasas de espectro extendido portadoras del gen mcr-1 por tipo de muestra y procedencia.
|
Procedencia |
Tipo de muestra |
||||
|
Sangre |
Orina |
Secreciones respiratorias bajas |
Hisopado rectal |
Total |
|
|
Abdomen |
1 |
1 |
1 |
- |
3 |
|
Ginecología |
- |
1 |
- |
- |
1 |
|
Medicina oncológica |
3 |
1 |
- |
10 |
14 |
|
Pediatría oncológica |
4 |
1 |
- |
- |
5 |
|
Tórax y mama |
- |
- |
- |
1 |
1 |
|
Urología |
1 |
- |
- |
- |
1 |
Además, el perfil de susceptibilidad de las EP-BLEE portadoras del gen mcr-1 mostraron resistencia a las fluoroquinolonas y a la gentamicina, y permanecieron sensibles a la amikacina. Cabe resaltar que dos aislamientos de K. pneumoniae presentaron resistencia a los carbapenémicos por la presencia de metalocarbapenemasas (Nueva Delhi Metalo beta-lactamasa [NDM, por sus siglas en inglés]) (Figura 2).
AMK: amikacina; GEN: gentamicina; IPM: imipenem; MEM: meropenem; CAZ:
ceftazidima; CTX: cefotaxima; CIP: ciprofloxacino.
* 22 aislamientos fueron portadores de genes blaCTX-M, y 2
aislamientos fueron además portadores de genes blaNDM.
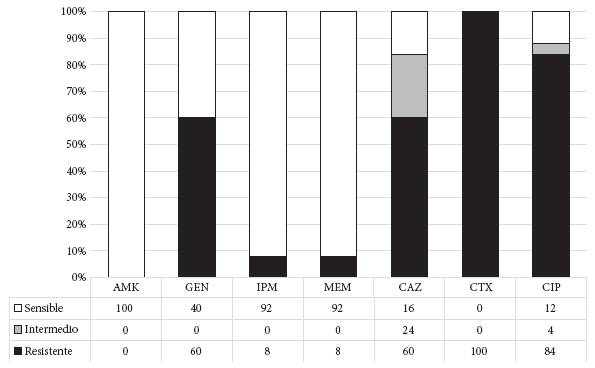
Figura 2. Perfil de susceptibilidad a los antimicrobianos de las enterobacterales productores de betalactamasas de espectro extendido portadoras del gen mcr-1 (n = 25)*.
DISCUSIÓN
La presencia de PMCR a causa del gen mcr-1 está ampliamente distribuida en todo el mundo y en algunos casos se encuentra relacionado con otros marcadores de resistencia, como las betalactamasas (BLEE o carbapenemasas) (2,7,11,18-20). En nuestro estudio, la presencia del gen mcr-1 en EP-BLEE fue del 15,2% de aislamientos recuperados de diferentes tipos de muestra de infecciones y colonizaciones (portadores) en pacientes hospitalizados. En 2017, Ugarte et al. (12) realizaron el primer reporte en el Perú sobre mcr-1 en siete aislamientos de E. coli recuperados a partir de cultivos de orina de pacientes comunitarios. En 2019, Deshpande et al. (11) informaron tres aislamientos de E. coli productores de mcr-1 a partir de infecciones de sangre y piel recuperados en 2016 en nuestro país, uno de los aislamientos era productor de BLEE (blaCTX-M-55).
La presencia de enterobacterales productores de betalactamasas portadores del gen mcr-1 se encuentran en aumento en la región (18-20), no solo coproductores de BLEE, sino también de carbapenemasas, como se observó en dos de nuestros aislamientos de K. pneumoniae MDR, con resistencias que van desde los betalactámicos (CTX-M, NDM), fluoroquinolonas, gentamicina e incluso colistina, que es considerada un antibiótico de último recurso frente a bacterias MDR, quedando solo la amikacina como alternativa terapéutica.
El método de tamizaje CAS presenta una concordancia del 99,5% comparada con el método microdilución en caldo, considerado como el método de referencia para la determinación de resistencia a la colistina. En nuestro estudio, se pudo detectar resistencia a la colistina en 17/25 aislamientos productores de mcr-1, probablemente debido a que estos aislamientos presentaban una concentración mínima inhibitoria (CMI) <3 µg/mL, que es la concentración de colistina que utiliza el método de tamizaje CAS (14).
Un aislamiento de K. pneumoniae catalogado como resistente a la colistina no portaba el gen mcr-1; esta resistencia a la colistina podría estar relacionada con mutaciones en genes del sistema regulador PhoPQ-PmrAB (2,3); o a la presencia de variantes alélicas del gen mcr no investigadas en este estudio (5,6). Un dato que nos parece importante mencionar es que desde 2020, el CLSI cambió los puntos de corte para la interpretación de colistina en enterobacterales, Pseudomonas aeruginosa y Acinetobacter spp. quedando solo las categorías de intermedio (≤2 µg/mL) y resistente (≥4 µg/mL); y dejando como metodologías aceptables la Microdilución en clado para colistina (BMD), prueba de colistina en agar (CAT, por sus siglas en inglés) y elución de disco de colistina en caldo (CBDE, por sus siglas en inglés) (13).
El método CPD-E (15), basado en la característica de la metaloproteína dependiente de zinc de la enzima fosfoetanolamina transferasa codificada por el gen mcr-1, que le permite ser inhibida por quelantes como el EDTA, tuvo una excelente correlación con los aislamientos portadores del gen mcr-1, demostrando ser una buena alternativa fenotípica para la detección de productores de MCR.
La presencia del gen mcr-1 en aislamientos clínicos MDR causantes de infecciones es preocupante, ya se ha demostrado la presencia gastrointestinal de bacterias con este gen en el ambiente hospitalario o comunitario (10). En nuestro estudio, la frecuencia de colonizados con EP-BLEE portadores del gen mcr-1 fue del 44%; esta colonización es muy peligrosa, ya que existe el riesgo de que pueda diseminarse, a través de plásmidos, a otras cepas virulentas o clones epidémicos (10).
Este estudio presenta algunas limitaciones que debemos mencionar. Los resultados obtenidos corresponden a una colección de aislamientos MDR (productores de BLEE) de una institución de salud especializada; no se pueden extrapolar los resultados a otras instituciones. Es necesario evaluar la presencia del gen mcr-1 con un número mayor de aislamientos no MDR de diferentes centros hospitalarios para conocer el impacto real de este marcador de resistencia en nuestro país. Además, no se analizaron variantes alélicas distintas del gen mcr-1 y otras causas de resistencia a colistina, como mutaciones en los genes del sistema regulador del lípido A de la membrana bacteriana.
En conclusión, la recolección de datos sobre resistencia bacteriana a los antimicrobianos considerados de última línea es crucial para establecer medidas acordes a la realidad local y compararlas a nivel regional y mundial. A pesar de que nuestros resultados muestran el panorama de un solo centro, la aparición de aislamientos MDR con resistencia a la colistina, respalda la necesidad de implementar estudios de epidemiología molecular para prevenir el establecimiento de infecciones asociadas a la atención en salud por este tipo de microorganismos.
Contribuciones de autoría: KYC y EGE concibieron el artículo, recolectaron los datos y el material de estudio, y redactaron. MZA, CRSA y JPS participaron en la idea de la investigación, la redacción y la asesoría técnica y administrativa. CBE y WVT participaron en la recolección de datos y material de estudio y redacción del artículo. Todos los autores realizaron la revisión crítica del artículo, aprobaron la versión final y asumen responsabilidad de los contenidos del manuscrito.
Conflictos de interés: Los autores no tienen ningún conflicto de interés que declarar.
Agradecimientos: Al personal del Laboratorio de Microbiología del Instituto Nacional de Enfermedades Neoplásicas por su colaboración en el proceso de recolección de los aislamientos; al equipo de investigación de Biotecnología y Salud del Centro de Investigaciones Tecnológicas, Biomédicas y Medioambientales por su contribución en biología molecular; y al personal del Laboratorio de Epidemiología Molecular y Genética de la Universidad Nacional Mayor de San Marcos por su continuo apoyo.
Financiamiento: Estudio financiado por FONDECYT para los Círculos de Investigación en Ciencia y Tecnología (RD N.° 142-2015-FONDECYT-DE) y por el Centro de Investigaciones Tecnológicas, Biomédicas y Medioambientales - CITBM.
REFERENCIAS BIBLIOGRÁFICAS
1. Falagas ME, Kasiakou SK. Colistin: the revival of polymyxins for the management of multidrug-resistant gram-negative bacterial infections. Clin Infect Dis. 2005;40(9):1333-41. doi: 10.1086/429323.
2. Gao R, Hu Y, Li Z, Sun J, Wang Q, Lin J, et al. Dissemination and Mechanism for the MCR-1 Colistin Resistance. PLoS Pathog. 2016;12(11):e1005957. doi: 10.1371/journal.ppat.1005957.
3. Blair JM, Webber MA, Baylay AJ, Aqbolu DO, Piddock LJ. Molecular mechanisms of antibiotic resistance. Nat Rev Microbiol. 2015;13(1):42-51. doi: 10.1038/nrmicro3380.
4. Liu YY, Wang Y, Walsh TR, Yi LX, Zhang R, Spencer J, et al. Emergence of plasmid-mediated colistin resistance mechanism MCR-1 in animals and human beings in China: a microbiological and molecular biological study. Lancet Infect Dis. 2016;16(2):161–168. doi: 10.1016/S1473-3099(15)00424-7.
5. Poirel L, Jayol A, Nordmann P. Polymyxins: antibacterial activity, susceptibility testing, and resistance mechanisms encoded by plasmids or chromosomes. Clin Microbiol Rev. 2017;30(2):557-96. doi: 10.1128/CMR.00064-16.
6. Giamarellou H. Epidemiology of infections caused by polymyxin-resistant pathogens. Int J Antimicrob Agents. 2016;48(6):614-21. doi: 10.1016/j.ijantimicag.2016.09.025.
7. Quiroga C, Nastro M, Di Conza J. Current scenario of plasmid-mediated colistin resistance in Latin America. Rev Argent Microbiol. 2018;51(1):93-100. doi: 10.1016/j.ram.2018.05.001.
8. Fernandes MR, Moura Q, Sartori L, Silva KC, Cunha MP, Esposito F, et al. Silent dissemination of colistin-resistant Escherichia coli in South America could contribute to the global spread of the mcr-1 gene. Euro Surveill. 2016;21(17):30214. doi: 10.2807/1560-7917.ES.2016.21.17.30214.
9. Monte DF, Mem A, Fernandes MR, Cerdeira L, Esposito F, Galvao JA, et al. Chicken meat as a reservoir of colistin resistant Escherichia coli strains carrying mcr-1 genes in South America. Antimicrob Agents Chemother. 2017;61(5):e02718-16. doi: 10.1128/AAC.02718-16.
10. Organización Panamericana de la Salud/Organización Mundial de La Salud [internet]. Alerta Epidemiológica: Enterobacterias con resistencia transferible a colistín, implicaciones para la salud pública en las Américas [Internet]. Washington D.C.: OPS/OMS; 2016. [citado el 25 de abril de 2020]. Disponible en: https://www.paho.org/hq/dmdocuments/2016/2016-jun-10-alerta-epi-enterob-resist.pdf.
11. Deshpande LM, Hubler C, Davis AP, Castanheira M. Updated prevalence of mcr-like genes among Escherichia coli and Klebsiella pneumoniae in the SENTRY program and characterization of mcr-1.11 variant. Antimicrob Agents Chemother. 2019;63:e02450-18. doi: 10.1128/AAC.02450-18.
12. Ugarte R, Olivo J, Corso A, Pasteran F, Albornoz E, Sahuanay ZP. Resistencia a colistín mediado por el gen mcr-1 identificado en cepas de Escherichia coli y Klebsiella pneumoniae. Primeros reportes en el Perú. An Fac med. 2018;79(3):213-7. doi: 10.15381/anales. v79i3.15313.
13. Clinical and Laboratory Standards Institute (CLSI). 2020. Performance standards for antimicrobial susceptibility testing: 30th informational supplement M100-S30 [Internet]. CLSI, Wayne, PA [citado el 25 de abril de 2020]. Disponible en: https://clsi.org/standards/products/microbiology/documents/m100/.
14. Servicio Antimicrobianos, Laboratorio Nacional de Referencia en Antimicrobianos, INEI-ANLIS "Dr Carlos G. Malbrán" [Internet]. Métodos de Screening "colistín Agar-Spot". [citado el 25 de abril de 2020]. Disponible en: http://antimicrobianos.com.ar/ATB/wp-content/uploads/2017/09/Protocolo-Agar-spot-COL-2017-version2-Agosto2017.pdf.
15. Yauri K, Gonzales E, Di Conza J, Gutkind G. Detection of plasmid-mediated colistin resistance by colistin pre-diffusion and inhibition with EDTA test (CPD-E) in Enterobactereaceae. J Microbiol Methods. 2019;167:105759. doi: 10.1016/j.mimet.2019.105759.
16. Servicio Antimicrobianos, Laboratorio Nacional de Referencia en Antimicrobianos, INEI-ANLIS "Dr Carlos G. Malbrán" [Internet]. Protocolo de PCR-Multiplex para la detección de carbapenemasas. [citado el 25 de abril de 2020]. Disponible en: http://antimicrobianos.com.ar/2019/10/protocolo-de-pcr-multiplex-para-la-deteccion-de-carbapenemasas/.
17. Ghasemi Y, Archin T, Kargar M, Mohkam M. A simple multiplex PCR for assessing prevalence of extended-spectrum β-lactamases producing Klebsiella pneumoniae in Intensive Care Units of a referral hospital in Shiraz, Iran. Asian Pac J Trop Med. 2013;6(9):703-708. doi: 10.1016/S1995-7645(13)60122-4.
18. Sellera FP, Fernandes MR, Sartori L, Carvalho MPN, Esposito F, Nascimento CL, et al. Escherichia coli carrying IncX4 plasmid mediated mcr-1 and blaCTX-M genes in infected migratory Magellanic penguins (Spheniscus magellanicus). J Antimicrob Chemother. 2017;72(4):1255-6. doi: 10.1093/jac/dkw543.
19. Delgado-Blas JF, Ovejero CM, Abadia-Patino L, Gonzalez-Zorn B. Coexistence of mcr-1 and blaNDM-1 in Escherichia coli from Venezuela. Antimicrob Agents Chemother. 2016;60(10):6356-8. doi: 10.1128/AAC.01319-16.
20. Dalmolin TV, Martins AF, Zavascki AP, de Lima-Morales D, Barth AL. Acquisition of the mcr-1 gene by a high-risk clone of KPC-2-producing Klebsiella pneumoniae ST437/CC258, Brazil. Diagn Microbiol Infect Dis. 2018;90(2):132-3. doi: 10.1016/j.diagmicrobio.2017.09.016.
Citar como: Yauri-Condor K, Zavaleta Apestegui M, Sevilla-Andrade CR, Piscoya Sara J, Villoslado Espinoza C, Vicente Taboada W, et al. Enterobacterales productores de betalactamasa de espectro extendido portadores del gen mcr-1 en Lima, Perú. Rev Peru Med Exp Salud Publica. 2020;37(4):711-5. doi: https://doi.org/10.17843/rpmesp.2020.374.5832.
Correspondencia: Edgar Gonzales Escalante; Calle José Santos Chocano 199, Ciudad Universitaria, Bellavista, Callao; egones_5@hotmail.com
Recibido: 19/05/2020
Aprobado:09/09/2020
En línea: 05/11/2020