Juan Ramírez-Illescas
Miluska Delgado-Flores
Alicia Núñez-Llanos
Maritza Mayta-Barrios
Carta al editor
Primer reporte de carbapenemasa tipo KPC en Pseudomonas aeruginosa en un hospital del Perú
First report of KPC-type carbapenemase in Pseudomonas aeruginosa at a peruvian hospital
Gina Salvador-Luján ![]() 1,2,
Bióloga con mención en Microbiología y Parasitología, magíster en
Microbiología
1,2,
Bióloga con mención en Microbiología y Parasitología, magíster en
Microbiología
Juan Ramírez-Illescas ![]() 3, biólogo
3, biólogo
Miluska Delgado-Flores ![]() 4, médica de enfermedades infecciosas
y tropicales
4, médica de enfermedades infecciosas
y tropicales
Alicia Núñez-Llanos ![]() 3, bióloga genetista
biotecnóloga
3, bióloga genetista
biotecnóloga
Maritza Mayta-Barrios ![]() 3, bióloga, maestra en Ciencias
3, bióloga, maestra en Ciencias
1 Laboratorio de Microbiología, Hospital Militar Central, Lima, Perú.
2 Facultad de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos, Lima, Perú.
3 Centro Nacional de Salud Pública, Instituto Nacional de Salud, Lima, Perú.
4 Servicio de Medicina Interna-Infectología, Hospital Militar Central, Lima, Perú.
Sr. Editor: En los últimos años se ha registrado el incremento de bacterias productoras de carbapenemasas; estas enzimashidrolizan a los carbapenémicos, antibióticos betalactámicos de amplio espectro utilizados en el tratamiento de infecciones por bacterias multirresistentes. Según la OrganizaciónMundial de la Salud, P. aeruginosa productora de carbapenemasas es uno de los microorganismos prioritarios en el estudio de la resistencia a los antibióticos a nivel mundial y una amenaza a la salud pública por producir infecciones graves con alta letalidad (1). Las enzimas Klebsiella pneumoniae carbapenemasa (KPC, por sus siglas en inglés) presentangran actividad de hidrólisis frente a los betalactámicos y confieren resistencia variable a los carbapenémicos; además, están asociadas a otros mecanismos de resistencia, así, las cepas portadoras se presentan como multirresistentes a los betalactámicos y a otras familias de antibióticos.
KPC en P. aeruginosa es codificada por el gen blaKPC localizado en el transposón Tn 4401 con capacidad de insertarse en los plásmidos de bacterias gramnegativas y de diseminación geográfica, y entre especies (2).
En América Latina se reportó el primer aislamiento de P. aeruginosa productora de KPC en Colombia en el 2007, posteriormente se reportaron en Puerto Rico, Trinidad y Tobago, Argentina y Brasil (3,4). En el Perú existen reportes de aislamientos de P. aeruginosa productoras de metalobetalactamasas (MBL) (5); sin embargo, no existen reportes de KPC.
Como parte de la vigilancia epidemiológica de las infecciones asociadas a la atención en salud establecida en el Hospital Militar Central, reportamos por primera vez el aislamiento de P. aeruginosa productora de KPC en la orina de un varón de 56 años, hospitalizado en el servicio de medicina interna. El paciente ingresó en abril del 2019 con diagnóstico de pie diabético grado 2 según la escala de WIFI (Wound, Ischemia and foot Infection), secundario a diabetes mellitus tipo 2 desde hace 26 años; además, el paciente tenía neuropatía diabética, hipertensión arterial, enfermedad renal crónica estadio III, colon irritable e infecciones urinarias a repetición; posteriormente se le detectó hidroureteronefrosis moderada bilateral a predominio derecho, colocándosele una sonda Foley.
Durante su estancia prolongada tuvo infecciones urinarias recurrentes que produjeron sepsis. Recibió tratamiento con antibióticos de amplio espectro: quinolonas, cefalosporina de cuarta generación, antipseudomónicos y carbapenémicos. En los urocultivos se aislaron Escherichia coli con resistencia a quinolonas y Proteus sp. con resistencia a aminoglucósidos y quinolonas (29 de julio del 2020); Acinetobacter sp. resistente a cefalosporinas, quinolonas, carbapenémicos y sensible a sulfametoxazol (5 de agosto del 2020). El 14 de septiembre del 2020 el laboratorio de microbiología reportó el aislamiento de bacilos gramnegativos no fermentadores de lactosa con resistencia a cefalosporinas, aminoglucósidos, quinolonas, carbapenémicos, monobactams y a betalactámicos asociados a inhibidores de betalactamasas. Este hallazgo se comunicó al servicio de epidemiología y al comité de infecciones intrahospitalarias. La cepa aislada se envió al Laboratorio de Referencia Nacional de Infecciones Intrahospitalarias del Instituto Nacional de Salud para la confirmación de la especie bacteriana, determinar la sensibilidad a los antimicrobianos, y detectar carbapenemasas y la susceptibilidad a la colistina.
Se identificó P. aeruginosa y en el antibiograma se confirmó la resistencia de contacto (6 mm) a meropenem, imipenem, ceftazidime, ceftalozano/tazobactam, amikacina, gentamicina, ciprofloxacina, levofloxacina, cefepime y piperacilina/tazobactam, utilizando los puntos de corte del Clinical and Laboratory Standards Institute (6). Se realizó la detección fenotípica de carbapenemasas por método de sinergia utilizando discos de ácido etilendiaminotetraacético/ mercaptoacetato de sodio (EDTA/SMA), imipinem y meropenem, con resultado negativo para MBL; y la prueba de Blue CARBA, con resultado positivo para carbapenemasas. Se realizó la pruebade inmunocromatografía (CORIS, RESIST-4 O.K.N.V.) y se obtuvo un resultado positivo con dos bandas de color rojo-púrpura en la línea de control y línea de KPC, lo que indica la presencia de KPC. La sensibilidad a colistina se determinó con el agar spot colistin, con resultado negativo.
La caracterización molecular se realizó previa extracción del ADN por lisis celular a partir de colonias bacterianas frescas de un cultivo en agar tripticasa soya. Los cebadores usados fueron KPC-F (5’- AAC AAG GAA TAT CGT TGA TG -3’) y KPC-R (5’- AGA TGA TTT TCA GAG CCT TA -3’). Para la amplificación del gen blaKPC se utilizó el protocolo descrito por el laboratorio Malbran de Argentina. Finalmente, mediante reacción de cadena de polimerasa se confirmó la presencia del gen blaKPC (Figura 1).
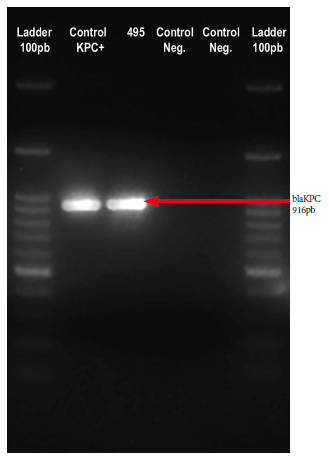
Figura 1. Productos de amplificación del gen blaKPC de 916 pares de bases (pb) del aislamiento de Pseudomonas aeruginosa (495). La amplificación se realizó por reacción en cadena de polimerasa convencional y la electroforesis se hizo en agarosa 1,5%. Marcador de tamaño molecular: 100 pb DNA Ladder GeneDirex®. KPC+: control positivo, Neg: control negativo.
La detección de KPC en P. aeruginosa tiene un gran impacto en la salud pública por conferir resistencia a todos los betalactámicos y diseminarse en elementos móviles genéticos a otras bacterias intrahospitalarias, lo cual aumenta la morbimortalidad de los pacientes. Se destaca la falta de opciones terapéuticas frente a P. aeruginosa productora de KPC y la necesidad de disponer de métodos de diagnóstico rápidos y oportunos para evitar su diseminación en el ambiente hospitalario. Este hallazgo nos conduce a generar la crítica del uso indiscriminado de antibióticos, promover su uso racional y evidencia la necesidad de implementar, como rutina, la detección rápida de carbapenemasas en los laboratorios de microbiología, para evitar el retraso de las decisiones terapéuticas y el control de las infecciones intrahospitalarias por bacterias multirresistentes.
Agradecimiento: Al Dr. Aleksandar Cvetković Vega por su colaboración en la revisión final del manuscrito.
Contribución de los autores: GSL, JRI, MMB, ANLL y MDF han participado en la concepción, investigación, realización de los procesos, diseño, análisis, interpretación, redacción y revisión crítica de la carta. Todos los autores aprobaron la versión final del manuscrito.
Financiamiento: Autofinanciado.
Criterios éticos: El presente reporte no requirió la aprobación de un Comité de Ética Institucional ni consentimiento informado del paciente hospitalizado, por ser parte de la vigilancia epidemiológica de las infecciones asociadas a la atención de salud establecidas en el Hospital Militar Central. Los datos de identificación del paciente fueron tratados confidencialmente.
REFERENCIAS BIBLIOGRÁFICAS
1.World Health Organization. Prioritization of pathogens to guide discovery, research and development of new antibiotics for drug-resistant bacterialinfection, including tuberculosis. [Consultado abril de 2019]. Disponible en: https://apps.who.int/iris/handle/10665/311820.
2. Naas T, Cuzon G, Villegas MV, Lartigue MF, Quinn JP, Nordmann P. Genetic structures at the origin of acquisition of the beta-lactamase bla KPC gene. Antimicrob Agents Chemother. 2008;52(4):1257-63. doi: 10.1128/AAC.01451-07.
3. Akpaka PE, Swanston WH, Ihemere HN, Correa A, Torres JA, Tafur JD, et al. Emergence of KPC-producing Pseudomonas aeruginosa in Trinidad and Tobago. J Clin Microbiol. 2009;47(8):2670-2671. doi: 10.1128/JCM.00362-09.
4. Walkty A, Alexander D, Karlowsky J, Nichol K, Embil J. Report of a KPC-producing Pseudomonas aeruginosa isolate in Canada. J Antimicrob Chemother. 2019;74(6):1748-1749. doi: 10.1093/jac/dkz064.
5. Salvador-Luján G, García-de-la-Guarda R, Gonzales-Escalante E. Caracterización de metalo-β-lactamasas en aislados clínicos de Pseudomonas aeruginosa recuperados de pacientes hospitalizados en el Hospital Militar Central. Rev Perú Med Exp Salud Publica.2018; 35(4): 363-641. doi: 10.17843/rpmesp.2018.354.3755.
6. Performance Standards for Antimicrobial Susceptibility Testing. Clinical Laboratory Stand; 2020. Disponible en: https://clsi.org/standards/products/microbiology/documents/m100/.
Citar como: Salvador-Luján G, Ramírez-Illescas J, Delgado-Flores M, Núñez-Llanos A, MaytaBarrios M. Primer reporte de carbapenemasa tipo KPC en Pseudomonas aeruginosa en un hospital del Perú. Rev Peru Med Exp Salud Publica. 2021;38(3):474-5. doi: https://doi.org/10.17843/rpmesp.2021.383.7044.
Correspondencia: Gina Salvador-Luján; Jr. Hipólito Unanue 159, Las Leyendas. San Miguel, Lima, Perú; gsalvador3@hotmail.com.
Recibido: 07/01/2021
Aprobado: 30/06/2021
En línea: 11/08/2021