Jesús Medina-Ranilla
Víctor Chau-Rodríguez
Renato Li-Soldevilla
Álvaro Vera-Albújar
Patricia J. García
Articulo revisión
Variantes del SARS-CoV-2: epidemiología, fisiopatología y la importancia de las vacunas
SARS-CoV-2 variants: epidemiology, pathophysiology and the importance of vaccines
Marcelo Bedoya-Sommerkamp ![]() 1,
Médico cirujano
1,
Médico cirujano
Jesús Medina-Ranilla ![]() 1, Médico cirujano
1, Médico cirujano
Víctor Chau-Rodríguez ![]() 1, Médico cirujano
1, Médico cirujano
Renato Li-Soldevilla ![]() 1, Médico cirujano
1, Médico cirujano
Álvaro Vera-Albújar ![]() 1, Médico cirujano
1, Médico cirujano
Patricia J. García ![]() 2, médico cirujano, doctor en medicina
2, médico cirujano, doctor en medicina
1 Facultad de Medicina, Universidad Peruana Cayetano Heredia, Lima, Perú.
2 Facultad de Salud Pública y Administración, Universidad Peruana Cayetano Heredia. Lima, Perú.
RESUMEN
El SARS-CoV-2 es un virus ARN monocatenario de la familia de los coronavirus, causante de la COVID-19 (Coronavirus Disease 2019). Este virus es responsable de la pandemia actual que, desde su aparición a finales de 2019, ha provocado la muerte de millones de personas y ha tenido un impacto global no solo a nivel sanitario sino también económico y social. Por ello, el presente artículo tiene como objetivo revisar la información más actualizada sobre el SARS-CoV-2, empezando por describir los mecanismos de transmisión del virus, su fisiopatología y filogenética. Asimismo, presentará a las variantes emergentes del SARS-CoV-2, su relevancia para la salud pública local y global, su epidemiología en Perú, y finalmente, el rol y la importancia de las vacunas en este contexto.
Palabras clave: SARS-CoV-2, COVID-19, epidemiología, fisiopatología, vacunas. (Fuente: DeCS BIREME).
ABSTRACT
SARS-CoV-2 is a single-stranded RNA virus of the coronavirus family, which causes COVID-19 (Coronavirus Disease 2019). This virus is responsible for the current pandemic, which, since its emergence in late 2019, has caused millions of deaths and has had a global impact not only on public health but also on social and economic areas. Therefore, this article aims to review the most up-to-date information on SARS-CoV-2, beginning with the description of the pathophysiology and phylogenetics of the virus. Also, we will present the emerging SARS-CoV-2 variants, their relevance for local and global public health, their epidemiology in Peru, and finally, the role and importance of vaccines in this context.
Keywords: SARS-CoV-2, COVID-19, epidemiology, pathophysiology, vaccines. (Source: MeSH NLM).
INTRODUCCIÓN
El SARS-CoV-2 es un virus ARN monocatenario de la familia de los coronavirus, causante de la COVID-19 (Coronavirus Disease 2019). Esta enfermedad fue reconocida por la Organización Mundial de la Salud (OMS) como pandemia en marzo de 2020 y, al momento, ha sido reportada en más de 180 países, con más de 218 millones de infectados y más de 4,5 millones de muertes (1).
Durante este tiempo hemos sido testigos de la evolución del virus, con la aparición de nuevas variantes que poseen mayor capacidad de transmisión, mayor habilidad de escapar de la inmunidad y mayor letalidad (2–4). Estas características hacen que el reto de controlar la pandemia sea mucho mayor, aun con varias vacunas efectivas ya aplicándose en diversos países y otras más en desarrollo (5).
El presente trabajo tiene como objetivo revisar la información más actualizada sobre el SARS-CoV-2, empezando por describir su fisiopatología y filogenética, a fin de entender mejor las variantes de este virus, su relevancia para la salud pública global y local, su epidemiología en el Perú, y, finalmente, el rol y la importancia de las vacunas en este contexto.
BÚSQUEDA Y SELECCIÓN DE EVIDENCIA
Se realizó una búsqueda en PubMed y medRxiv. Se incluyeron todos los artículos publicados hasta el 2 de septiembre de 2021, y se limitó la búsqueda a artículos en español e inglés, de acuerdo con los siguientes filtros: "Clinical Trial", "Meta-Analysis", "Review", "Systematic Review". Para la búsqueda se utilizaron las palabras clave: "SARS-CoV-2", "Variants", "Peru", "Epidemiology, "Pathophysiology" y "Vaccines". Para los ensayos clínicos, se incluyeron solamente estudios en humanos.
Se encontraron 5871 artículos. Se eliminaron los manuscritos duplicados y, revisando los títulos, se eliminaron aquellos no considerados relevantes para el presente estudio. Posteriormente, se leyeron los resúmenes de los artículos completos (1752) para seleccionar los que se incluirían en el estudio. Finalmente, se incluyeron 64 artículos.
Por otro lado, se revisaron las páginas web del Ministerio de Salud (https://www.gob.pe/minsa), Instituto Nacional de Salud (INS) (https://web.ins.gob.pe/es), OMS (https://www.who.int/emergencies/diseases/novel-coronavirus-2019), Centro de Control de Enfermedades de Estados Unidos (CDC) (https://cdc.gov/coronavirus/2019-ncov/index.html), Administración de Alimentos y Medicamentos de los Estados Unidos (FDA) (https://www.fda.gov/about-fda/fda-en-espanol/enfermedad-del-coronavirus-covid-19) y del Global Initiative on Sharing All Influenza Data (GISAID) (https://www.gisaid.org/). Para la búsqueda en estas plataformas digitales se utilizaron los términos "Coronavirus", "COVID-19", "Variantes/Variants", "Vacunas/Vaccines", con lo que se encontraron normas técnicas, datos de coberturas, campañas y noticias acerca del tema. Se incluyeron 16 artículos.
FISIOPATOLOGÍA DE LA INFECCIÓN POR EL SARS-CoV-2
Tropismo y mecanismo de ingreso del virus a las células hospederas
Para su ingreso a la célula hospedera, el SARS-CoV-2 requiere la unión de su espícula (proteína S) a la enzima convertidora de angiotensina 2 (ECA-2) en la membrana plasmática de la célula hospedera. Sin embargo, primero dicha proteína debe ser escindida por la proteasa transmembrana de serina 2 (Transmembrane Protease Serine - TMPRSS2) de la membrana de la célula hospedera en dos subunidades: S1, que contiene el dominio de unión de región (Region Binding Domain - RBD) a la ECA-2 y S2, que facilita la fusión viral al acercar las membranas viral y celular (6). No obstante, esta escisión también puede darse por la catepsina-L en los endosomas, lo que permite infectar células sin TMPRSS2, pero es un proceso más lento (7).
Asimismo, aunque el SARS-CoV-2 posee una homología genómica del 80% con el SARS-CoV, su proteína S difiere en que 1) su RBD tiene 10 a 20 veces mayor afinidad por la ECA-2, y 2) su sitio de escisión S1/S2 puede ser procesado previamente por la proteína intra y extracelular furina, la cual se expresa en todo el organismo, pero a nivel respiratorio casi exclusivamente en el tracto respiratorio superior (8,9). Dicho procesamiento previo permite una escisión más rápida por TMPRSS2, evitando la vía lenta endosomal y, de esta manera, extiende su tropismo celular hacia el tracto respiratorio superior, lo que brinda al virus mayor rapidez y capacidad de transmisión (7,10). Por último, otro factor que incrementa la probabilidad de unión a la ECA-2, es el número y flexibilidad de su proteína S. Cada virión posee 24-40 moléculas de proteínas S, las cuales son capaces de realizar movimientos de rotación y balanceo (11).
Interacciones con el sistema inmune
La proteína S del SARS-CoV-2 evade el reconocimiento del sistema inmune al estar cubierta por varios glucanos no inmunogénicos y al ocultar su RBD en sus regiones internas, mostrándola únicamente ante la proximidad de ECA-2, y estabilizando dicha conformación con 2 moléculas de glucanos debajo de la RBD (12).
Asimismo, el SARS-Cov-2 prefiere utilizar la proteína TMPRSS2 para su ingreso a la célula hospedera porque, además de su mayor rapidez, le permite evitar las proteínas antivirales endosomales (13). Ensayos clínicos realizados con cloroquina, fármaco que altera el pH endosomal y dificulta la entrada de virus por esta vía, no han tenido resultados positivos, a pesar de que evitan la replicación viral in vitro. Esto se debe a que la cloroquina actúa en células cultivadas con SARS-Cov-2 que expresan catepsina-L, mas no la proteína TMPRSS2 (14).
Al replicarse, el SARS-CoV-2 evade el sistema inmune mediante diversos mecanismos. Uno de ellos implica la conversión del retículo endoplasmático liso en vesículas que recubren al ARN viral, evitando que sea reconocido por los receptores reconocedores de patrones (Pattern Recognition Receptors - PRR) de las células del sistema inmune innato. Otros mecanismos involucran sus proteínas virales no estructurales (Non-Structural Protein - NSP), como el poro formado por NSP3 en la vesícula antes descrita que expulsa el ARN viral hacia el citosol para su traducción. Cabe resaltar a la NSP1, que inhibe la respuesta de interferón a varios niveles: 1) utiliza las proteasas de la célula hospedera para degradar cualquier ARN mensajero (ARNm) no viral, 2) ocluye la entrada del canal ribosomal, inhibiendo así la traducción del ARN no viral, y 3) se une a los canales de salida del núcleo celular, evitando la salida del ARNm al citoplasma (15-18). Aunque falta confirmar el rol del resto de estas proteínas, hay evidencia de que serían más potentes que sus contrapartes en otros coronavirus, incluyendo al SARS-CoV y al MERS-CoV (7,19).
Asimismo, algunas proteínas S, en lugar de anclarse a nuevos viriones, viajan a la membrana celular de la célula infectada, en donde activan canales iónicos y facilitan la formación de una cubierta lipídica que induce la fusión de varias células. De esta manera, se forman sincitios que permiten la expulsión de millones de viriones y la evasión del sistema inmune al fusionarse incluso con células de dicho sistema, como linfocitos. Esto podría explicar en parte la linfopenia observada en casos graves de COVID-19 (18,20).
Las partículas virales, en general, al salir de la célula hospedera, forman su cubierta con parte de la membrana plasmática. Sin embargo, los coronavirus obtienen dicha estructura de las membranas del aparato de Golgi y son expulsados a través de vesículas de dicha organela. Además, el SARS-CoV-2 sale de la célula hospedera a través de lisosomas. Tanto el aparato de Golgi como los lisosomas son ricos en furina, y se postula que esa es la razón por la cual el virus sigue esa línea de tráfico intracelular (21).
Por otro lado, se intenta encontrar una explicación fisiopatológica de los factores de riesgo conocidos para enfermedad severa. Un artículo reciente postula que la presencia de autoanticuerpos contra las proteínas de la respuesta interferón tipo 1 (IFN-1), que incrementan con la edad, podrían estar implicadas en este riesgo (22). Además, se postula que el desarrollo de casos severos en pacientes mayores (inmunosenescencia) o con factores de riesgo (obesidad, diabetes mellitus, falla renal, enfermedad pulmonar obstructiva crónica, tratamiento inmunosupresor, etc.) puede deberse a una dificultad para montar una respuesta inmune adaptativa de tipo Th1 (contra patógenos intracelulares) adecuada. En estos casos, puede generarse o por un estado proinflamatorio basal, con predominio de interleuquina 1 (IL-1), interleuquina 6 (IL-6) y factor de necrosis tumoral alfa (TNF-α) o por una inmunosupresión celular (23,24). Ello, junto con lo detallado anteriormente, facilita una gran replicación viral descontrolada.
El incremento agudo de estas IL dirigirá una respuesta de tipo Th2 (contra patógenos extracelulares y en alergias), incapaz de erradicar la infección con la consecuente sobreestimulación y desregulación del sistema inmune (25,26). Adicionalmente, se promueve la secreción de anticuerpos no neutralizantes por los linfocitos B (que podrían facilitar la infección de los fagocitos), y la activación excesiva de respuesta Th17, que promovería una reparación tisular anómala y fibrosis (23,25,27). Esta situación, independientemente de la replicación viral, desencadena una neumonía intersticial, que puede evolucionar al síndrome de dificultad respiratoria aguda (SDRA) (23,24).
Por último, en fases avanzadas existe disrupción de la barrera alveolo-capilar, que permite la infección de las células endoteliales pulmonares y sistémicas. Esto facilita un estado procoagulante, vasoconstrictor local y dilatador sistémico, lo que genera micro trombos, embolismos, hipotensión, sepsis y fallo multiorgánico (23,24,28-30). Aquellos que sobreviven pueden presentar pérdida de función tisular normal debido a inflamación prolongada y/o fibrosis (31). Cabe recordar que la ECA-2 se encuentra en múltiples tejidos como la lengua, esófago, íleon, colon, vías biliares, corazón, riñón, vejiga, glía, endotelio vascular, vías respiratorias altas y bajas, etc.
VARIANTES DEL SARS-COV-2
Una mutación es un cambio específico en la secuencia de nucleótidos en el material genético de un organismo, y aquel que contenga una o más de estas mutaciones respecto a su secuencia genética original es denominado "variante" (32). Nos referimos a variantes clínica y epidemiológicamente relevantes (33) a aquellas que, gracias a una o más mutaciones, son capaces de escapar de la inmunidad natural (provocar reinfecciones), escapar de la inmunidad generada por vacuna (infecciones posvacunales), escapar de la efectividad del tratamiento (fármacos antivirales, anticuerpos monoclonales, plasma convaleciente, etc.), afectar la severidad (mayor virulencia) o dinámica de transmisión (mayor infectividad) y afectar el rendimiento de las pruebas diagnósticas.
A pesar de que la tasa de mutación de los virus en general es alta (generadas al azar durante su replicación) el SARS-CoV-2 posee una ARN polimerasa dependiente de ARN (RdRp) capaz de corregir errores, lo que explicaría su menor tasa de mutaciones respecto a otros virus ARN. Sin embargo, las mutaciones que sí logran presentarse se enfrentan a un proceso de selección natural: solo aquellas que le confieran al virus una mayor transmisibilidad o escape del sistema inmune persisten en la población (32). Es el secuenciamiento genético del virus a nivel epidemiológico el que nos permite observar esta evolución (34).
Potenciales mecanismos fisiopatológicos de las principales mutaciones
Además de su identificación, el secuenciamiento genético permite explorar los mecanismos mediante los cuales mutaciones puntuales pueden dar una ventaja evolutiva respecto a su contraparte salvaje. Las seis mutaciones puntuales que a la fecha parecen tener mayor importancia son la D614G, N501Y, E484K, P681H, L452R y T478K (20,27,35-45). Sus características se resumen en la Tabla 1.
Tabla 1. Mutaciones importantes de las variantes emergentes del SARS-CoV-2.
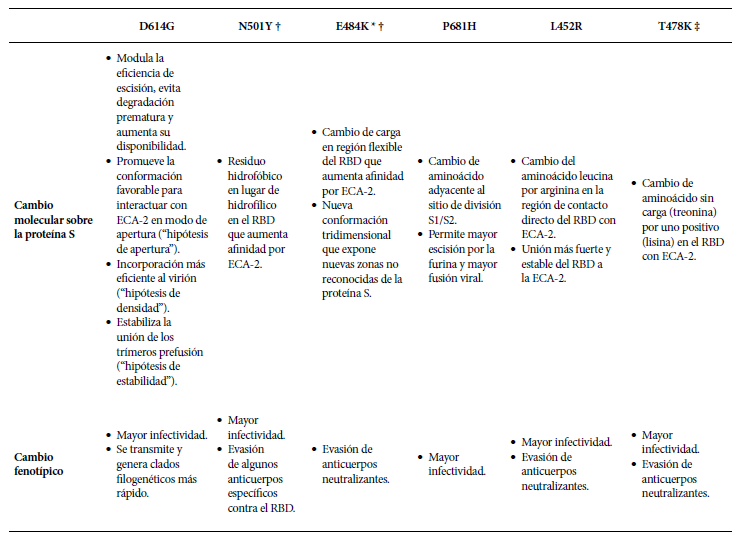
* Apareció de manera espontánea en SARS-CoV-2 cultivado en células de pulmón humanas in vitro en un medio rico en plasma convaleciente (1).
‡ Apareció en SARS-CoV-2 cultivado en medio rico en anticuerpos neutralizantes débiles (2).
† La presencia simultánea de N501Y y E484K en la misma variante produce un cambio conformacional del RBD mayor que con cada mutación por separado (efecto sinérgico), lo que otorga mayor escape del sistema inmune al poder evadir mayor variedad de anticuerpos (3).
Si bien la información más difundida sobre el posible mecanismo mediante el cual las mutaciones de las nuevas variantes generarían cambios en el fenotipo del virus es sobre las mutaciones en la proteína S, se está estudiando también los efectos de las mutaciones en las NSP. Por ejemplo, un estudio reciente con la variante delta, asocia la mutación 323(P->L) en la NSP12, que forma parte del complejo RdRp, a una mayor severidad y mortalidad por SARS-CoV-2; el mecanismo planteado sería una mayor estabilidad de este complejo y, por tanto, una mayor capacidad de replicación viral (46).
Las nuevas variantes y su nomenclatura
La transmisión y diseminación del SARS-CoV-2 por más de un año y medio ha permitido la generación de múltiples mutaciones y variantes. Estas han tratado de ser organizadas y nombradas según diferentes grupos de investigación (47). Rambaut et al las clasificaron en linajes (PANGO lineages), según sus características epidemiológicas y su relevancia biológica. Dicha nomenclatura ha sido la más utilizada recientemente (48,49).
Por su parte, el Instituto de Salud Pública del Reino Unido utiliza una nomenclatura propia y denomina "variantes de cuidado" (Variants of Concern - VOC) a aquellas con un impacto epidemiológico importante demostrado, y "variantes en estudio" (Variants Under Investigation - VUI), a aquellas con el potencial de volverse VOC (50). La CDC de los Estados Unidos y la OMS han adoptado también la definición de "variantes de cuidado" y han acuñado el término "variantes de interés" (Variants of Interest - VOI), con interpretación equivalente a las VUI y recomiendan su uso a nivel mundial (33,34,51). Recientemente, la OMS ha propuesto una nueva nomenclatura de las VOC y VOI usando el alfabeto griego, para facilitar su pronunciación y evitar la estigmatización de los países en donde se originaron (52). Actualmente, se cuenta con más de 1350 linajes identificados y por lo menos nueve variantes emergentes con relevancia epidemiológica demostrada, clasificadas como VOC o VOI por la OMS (49,60) (Tabla 2).
Tabla 2. Variantes emergentes de SARS-CoV-2 epidemiologicamente relevantes segun la Organizacion Mundial de la Salud.
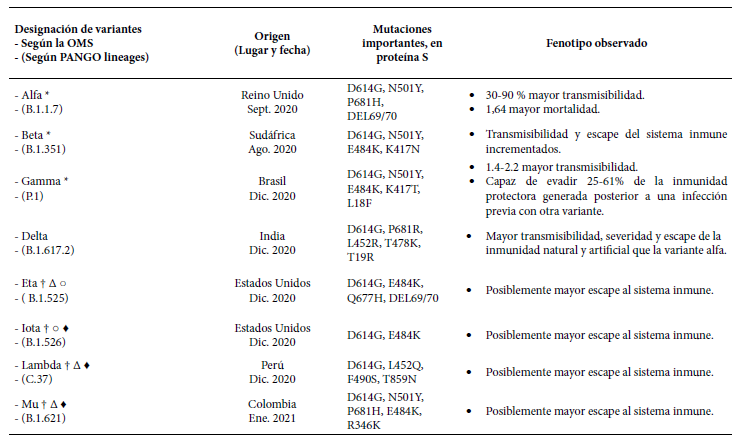
* Variantes de cuidado según la CDC, OMS y PHE. El INS no considera actualmente a la variante beta una VOC.
† Variantes de interés según la OMS. Δ Variantes de interés según la Instituto de Salud Pública del Reino Unido (PHE). ○ Variantes de interés según la CDC. ♦ Variantes de interés según el INS.
Como se puede observar, si bien hay un relativo consenso sobre qué variantes son de cuidado (VOC) o de interés (VOI) a nivel global, las clasificaciones no son exactamente iguales. Esto se debe a que cada institución asigna dichas categorías a las variantes según su relevancia dentro de cada uno de sus respectivos países. Por ejemplo, la variante lambda se considera VOI en el Reino Unido y en el Perú, pero en los Estados Unidos su prevalencia es tan baja que no se la considera importante (32,50,51).
Por otro lado, también se encuentran las variantes consideradas "provisionalmente extintas"; por ejemplo, las variantes A.23.1+E484K, B.1.1.7+E484K y B.1.324.1+E484K. Estas fueron, en su momento, variantes de cuidado o interés para el Reino Unido, pero al no encontrarse en los registros internacionales en las últimas 12 semanas, se han dejado de considerar para el reporte (50). De esto se puede concluir que la categorización de una variante como VOC o VOI en el futuro seguirá siendo un proceso dinámico, que no solo dependerá de las mutaciones que tenga cada variante, sino de su interacción multifactorial con las poblaciones y su "competencia biológica" contra las otras variantes circulantes a lo largo del tiempo.
Orígenes propuestos de las nuevas variantes
Se encuentran dos teorías, no mutuamente excluyentes, sobre el origen de las VOI. La primera postula que habrían aparecido a partir de infecciones en pacientes inmunosuprimidos, en su mayoría con una respuesta de células B deficiente. En ellos, se ha observado ciertas características clave que favorecen su aparición: mayor carga viral, mayor duración de enfermedad (hasta 134 días) y, lo más resaltante, la aparición de mutaciones capaces de evadir el tratamiento con plasma convaleciente (por tal motivo, algunos autores recomiendan tener precaución con dicho tratamiento) (61–63).
No obstante, una segunda teoría afirma que, teniendo en cuenta que la cantidad de pacientes inmunocomprometidos con COVID-19 que han recibido plasma convaleciente es muy pequeña en comparación con el total de infectados, es más probable que las variantes emerjan espontáneamente por la gran cantidad de virus en replicación, transmitiéndose entre cientos de millones de humanos y que, la más aptas para sobrevivir, son seleccionadas por la respuesta inmune (61). En efecto, se puede ver que las características clave a nivel individual presentes en los reportes mencionados de pacientes inmunocomprometidos podrían extrapolarse a nivel poblacional:
Mayor cantidad de virus: A nivel poblacional se presenta como más personas infectadas. Los países en los que se identificaron las principales VOC (Reino Unido [alfa], Sudáfrica [beta], Brasil [gamma] e India [delta]) estaban pasando por picos de incidencia de casos (1).
Tiempo: La fecha de aparición de las VOC se inicia luego de un año de la identificación del SARS-CoV-2, y, mientras más tiempo pasa, más variantes aparecen (Tabla 2).
Presión selectiva: Mientras más personas se infectan y se recuperan, la cantidad de anticuerpos contra el virus es mayor. Se ha descrito que la "inmunidad de rebaño" no es duradera, tanto en estudios in vitro como en pacientes tratados con plasma convaleciente (61,64,65). Además, se ha descrito la aparición de variantes que evaden la inmunidad natural adquirida, generando rebrotes importantes, como el reportado a principios del 2021 en Brasil por la mutación E484K (56). Al momento, queda pendiente estudiar el efecto de la inmunidad inducida artificialmente por las vacunas, mientras que poco a poco mayor porcentaje de la población mundial vaya siendo vacunada (66).
Por otro lado, al hacer el análisis filogenético de las mutaciones compartidas por las nuevas variantes (N501Y, E484K, P681H, L452R, T478K) se ve que han ocurrido de manera independiente en tiempo y espacio (evolución convergente), lo que apunta nuevamente a una presión selectiva poblacional que lleva a una mejor adaptación del SARS-CoV-2 al ser humano (3,58,67).
Finalmente, cabe mencionar la aparición de una nueva variante que podría ser resultado de ambas teorías de origen. Esta variante recibe el nombre de C.1.2 y se ha detectado por primera vez en Sudáfrica, durante su tercera ola en el mes de mayo de 2021. Si bien su prevalencia actual es baja, llama la atención tres aspectos: 1) su secuencia se encuentra a 44-59 mutaciones de la variante original de Wuhan (una de las más diferentes hasta el momento); 2) su tasa de mutación calculada es 1,8 veces más rápida que la tasa de evolución previamente esperada del SARS-CoV-2, es decir, ha acumulado esta gran cantidad de mutaciones en una menor cantidad de tiempo, con respecto a sus linajes contemporáneos (argumento que hace pensar que se originó en un solo hospedero); y, apuntando a una evolución convergente, 3) comparte varias mutaciones con otras VOC y VOI, que incrementan la infectividad del virus y lo ayudan a evadir el sistema inmune, en especial las respuestas contra las variantes alfa y beta (43). La relevancia de esta nueva variante estará sujeta a la vigilancia epidemiológica de su comportamiento. Actualmente, no es reconocida como una VOC o VOI por la OMS (60).
IMPACTO EPIDEMIOLÓGICO DE LAS NUEVAS VARIANTES
El impacto de las variantes y sus mutaciones se aprecia en el tiempo. Por ejemplo, la mutación D614G se originó en enero 2020 y, en marzo de ese año, llamó la atención por presentar mayor infectividad (32,68). Para junio, había llegado a ser la variante predominante en el mundo y, al momento, el 100% de las variantes activas del SARS-CoV-2 contienen dicha mutación (47).
Sin embargo, el impacto epidemiológico más evidente de las nuevas variantes es la generación de nuevas olas de contagios luego de su aparición inicial en sus respectivos países de origen. Es posible que las medidas clásicas de restricción sean menos efectivas contra las variantes con mayor infectividad, y/o que la inmunidad de rebaño alcanzada con una variante previa ya no resulte efectiva (1,4,54-56,58). No obstante, es importante mencionar que, aunque algunas variantes como la alfa o delta, han demostrado estar asociadas a mayor mortalidad, dicho efecto a nivel epidemiológico no se da solamente de manera directa por las mutaciones que poseen. También se genera de forma indirecta, por su capacidad de generar una gran cantidad de contagios en corto tiempo, lo cual sobrecarga los sistemas de salud (2,4,54,69).
En la actualidad, la variante más prevalente en el mundo es la delta (47). En países en los que hace unos meses la variante alfa (más transmisible y mortal que la variante original de Wuhan) era la más prevalente, la variante delta la ha desplazado casi por completo (47,50). Es importante mencionar que dichos países (Inglaterra, Estados Unidos, Alemania, Israel, España, entre otros) presentan un considerable porcentaje de su población vacunada, por lo menos más del 50% con dos dosis (66).
La variante delta ha demostrado ser más transmisible y conllevar a un mayor riesgo de hospitalización que la variante alfa en personas no vacunadas (4,69). Además, posee la capacidad de evadir la respuesta inmune ya sea natural o inducida por vacunas generando así reinfecciones (4,50,70).
NUEVAS VARIANTES EN EL PERÚ
En Latinoamérica, se ve un panorama relativamente distinto al descrito en la sección anterior. La variante delta aún no es la más prevalente, hay otras variantes circulantes con una mayor presencia, como la gamma, lambda y la recientemente descrita variante mu (47). Este patrón se puede observar en países con alto porcentaje de población vacunada, como Chile, y en países aún en camino de completar su campaña de vacunación como Perú (47,51).
Sin embargo, lo que sucede es dinámico en el tiempo y según regiones. Un reporte del INS, publicado el 25 de marzo de 2021, analizó muestras de pacientes diagnosticados con COVID-19 por PCR, provenientes de Lima Metropolitana (n = 199) y Loreto (n = 79). En Lima Metropolitana se encontró que el 39,7% de las muestras correspondían a la variante gamma; el 2,5% a la variante alfa y el 39,2% a otros linajes, mientras que en Loreto se encontró que el 8,1% correspondía a la variante gamma y el 70,1% a otros linajes. No se identificó a la variante alfa (71).
En abril del mismo año, se publicó un estudio, que describe a un nuevo linaje proveniente de la variante B.1.1.1, llamado C.37, al que posteriormente se le nombró con la letra griega lambda. Su origen ha sido identificado en el Perú y en Chile (72).
El INS implementó una estrategia de secuenciamiento genómico con resultados positivos que son presentados en el portal web institucional según la semana epidemiológica (51). Estos permiten ver cómo, progresivamente, la variante lambda (C.37), se convirtió en la variante más detectada, llegando a constituir hasta un 88,7% hacia fines de abril del 2021 (34). Posteriormente, y acompañando a la disminución del número de casos de COVID-19 en el país, la variante Lambda ha ido disminuyendo en importancia porcentual. Para finales de agosto 2021, la variante delta representa un 40% de las muestras; la variante gamma, 37%; la variante lambda, 17%, y la variante mu, 5% (51).
LAS VACUNAS Y NUEVAS VARIANTES
El desarrollo de vacunas debe ir de la mano con la vigilancia genómica. La posibilidad de escape inmunológico a las vacunas ha sido apoyada por reportes de disminución de la eficacia para desarrollar una enfermedad sintomática por la variante beta. En Sudáfrica, donde la variante beta con la mutación E484K era predominante, los resultados preliminares de la fase 3 de Johnson & Johnson (vacuna de vector viral), mostraron una eficacia contra el desarrollo de enfermedad sintomática del 57%, menor al 72% reportado en los Estados Unidos (55,73). Asimismo, los resultados preliminares de la fase 3 de Novavax (vacuna de subunidad proteica) en población sudafricana reportaron una eficacia contra el desarrollo de enfermedad sintomática de 49,4% en comparación al 89,3% reportado en el Reino Unido (74). Asimismo, otros estudios in vitro con las vacunas de Pfizer y AstraZeneca mostraron una disminución de los títulos de anticuerpos frente a la variante delta" (75-77). La vacuna desarrollada por Sinopharm (vacuna de virus inactivado), la cual viene siendo utilizada en el Perú, ha mostrado una eficacia contra el desarrollo de enfermedad sintomática de 78,1% por la cepa original y de 59% por la variante delta (70), mientras que un estudio en trabajadores de salud en el Perú durante una segunda ola con predominancia de variantes lambda y gamma reportó una eficacia de 94% para prevenir la muerte (80). Sin embargo, aún no se han definido los estándares in vitro (títulos de anticuerpos o marcadores de inmunidad celular) que se asocian a protección de la vacuna contra infecciones (76,77). Hasta el momento, solo la vacuna de Pfizer ha recibido la aprobación total por la FDA para su administración a personas mayores de 16 años (78). En la Tabla 3 se muestra la eficacia de las principales vacunas en fase 3 contra el desarrollo de enfermedad severa sobre las variantes del SARS-CoV-2 (60,75,77,79,80).
Tabla 3. Eficacia de las vacunas en fase 3 contra el desarrollo de enfermedad sintomática sobre las variantes del SARS-CoV-2.
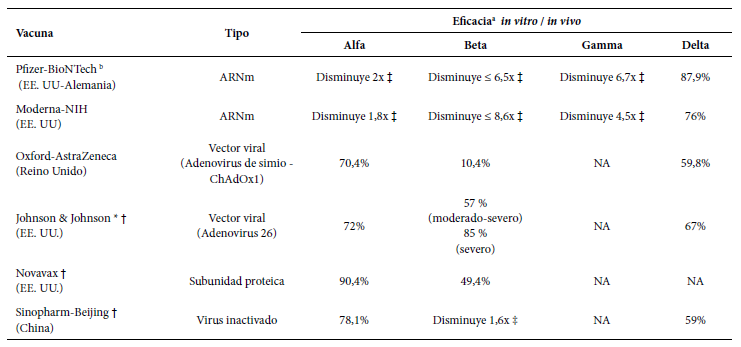
a Eficacia para evitar el desarrollo de enfermedad sintomática. b Vacuna aprobada por la FDA. * Vacunas con una sola dosis. † Vacunas con reportes de fase 3 interinos, no publicados en revistas científicas. ‡ Disminución de la producción de anticuerpos neutralizantes in vitro.
NA = no se disponen de datos.
CONCLUSIONES
La aparición de variantes del SARS-CoV-2 con características clínicas y epidemiológicas relevantes seguirá dándose mientras persista el contagio masivo con el virus. La falta de adherencia a las medidas de salud pública, como la no utilización de mascarillas, el no respetar el distanciamiento social y la demora en la administración de las vacunas favorecen este hecho.
Además, debido al potencial de las nuevas variantes de ser más transmisibles y de escapar de la inmunidad inducida natural y artificial, es una prioridad el desarrollo de vacunas de próxima generación que provoquen una actividad neutralizante de amplio espectro. Con la evidencia actual, creemos necesaria la creación de una normativa global para enfrentar este desafío. En primer lugar, es importante el secuenciamiento genómico exhaustivo para vigilar la aparición de nuevas variantes, a través de la creación de un sistema de alerta temprana genómica. Luego, se necesita evaluar la efectividad de las vacunas existentes contra las nuevas variantes y, con esa información, reformularlas periódicamente. Además, las vacunas deberán desarrollarse de acuerdo con las necesidades regionales, para responder a los brotes que surjan en los diferentes países.
Sin embargo, lo más importante sigue siendo suprimir la replicación viral a nivel comunitario. Esto solo se logrará con medidas efectivas de salud pública, la distribución temprana y equitativa de las vacunas disponibles y el alcance de una alta cobertura de vacunación. Además, aunque la población se encuentre vacunada, debe seguir acatando las medidas de salud pública de distanciamiento físico, uso de mascarillas y ventilación de ambientes. Finalmente, estas medidas deben ser coordinadas de manera global, promoviendo el intercambio de información científica, experiencias y regulaciones.
Contribuciones: MBS, VCR, JMR, RLS y AVA participaron en la concepción y diseño del artículo, la recolección y análisis de datos, la redacción del manuscrito y la aprobación de la versión final. PJG participó en la revisión crítica del artículo y aprobación de la versión final.
Conflictos de interés: Los autores declaran no tener conflicto de intereses.
Financiamiento: El presente trabajo ha sido autofinanciado por los autores.
REFERENCIAS BIBLIOGRÁFICAS
1. Jhons Hopkins University of Medicine. [Internet]. 2021. COVID-19 Map - Johns Hopkins Coronavirus Resource Center [citado el 6 septiembre 2021]. Disponible: https://coronavirus.jhu.edu/map.html.
2. Challen R, Brooks-Pollock E, Read JM, Dyson L, Tsaneva-Atanasova K, Danon L. Risk of mortality in patients infected with SARS-CoV-2 variant of concern 202012/1: matched cohort study. BMJ. 2021;579. doi: 10.1136/bmj.n579.
3. Faria NR, Mellan TA, Whittaker C, Claro IM, Candido D da S, Mishra S, et al. Genomics and epidemiology of a novel SARS-CoV-2 lineage in Manaus, Brazil. Science. 372(3544):815-21. doi: 10.1101/2021.02.26.21252554.
4. Mlcochova P, Kemp S, Dhar MS, Papa G, Meng B, Mishra S, et al. SARS-CoV-2 B.1.617.2 Delta variant emergence and vaccine breakthrough. Research Square 2021. p. 721.
5. Zimmer C, Corum J, Wee S-L. Coronavirus Vaccine Tracker. The New York Times [Internet]. 2021 [citado el 18 marzo 2021]. Disponible: https://www.nytimes.com/interactive/2020/science/coronavirus-vaccine-tracker.html.
6. Xia S, Zhu Y, Liu M, Lan Q, Xu W, Wu Y, et al. Fusion mechanism of 2019-nCoV and fusion inhibitors targeting HR1 domain in spike protein. Cell Mol Immunol. 2020;17(7):765-7. doi: 10.1038/s41423-020-0374-2.
7. Harrison AG, Lin T, Wang P. Mechanisms of SARS-CoV-2 Transmission and Pathogenesis. Trends Immunol. Elsevier; 2020;41(12):1100-15. doi: 10.1016/j.it.2020.10.004.
8. Wrapp D, Wang N, Corbett KS, Goldsmith JA, Hsieh C-L, Abiona O, et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science. 2020;367(6483):1260-3. doi: 10.1126/science.abb2507.
9. Wolff G, Limpens RWAL, Zevenhoven-Dobbe JC, Laugks U, Zheng S, de Jong AWM, et al. A molecular pore spans the double membrane of the coronavirus replication organelle. Science. 2020;369(6509):1395-8. doi: 10.1126/science.abd3629.
10. The Human Protein Atlas. [Internet]. 2021. Tissue expression of Furin [citado el 15 mayo 2021]. Disponible: https://www.proteinatlas.org/ENSG00000140564-FURIN/tissue.
11. Turoňová B, Sikora M, Schürmann C, Hagen WJH, Welsch S, Blanc FEC, et al. In situ structural analysis of SARS-CoV-2 spike reveals flexibility mediated by three hinges. Science. 2020;370(6513):203-8. doi: 10.1126/science.abd5223.
12. Casalino L, Gaieb Z, Goldsmith JA, Hjorth CK, Dommer AC, Harbison AM, et al. Beyond Shielding: The Roles of Glycans in the SARS-CoV-2 Spike Protein. ACS Cent Sci. 2020;6(10):1722-34. doi: 10.1021/acscentsci.0c01056.
13. Peacock TP, Goldhill DH, Zhou J, Baillon L, Frise R, Swann OC, et al. The furin cleavage site in the SARS-CoV-2 spike protein is required for transmission in ferrets. Nat Microbiol. 2021;6(7):899-909. doi: 10.1038/s41564-021-00908-w.
14. Wang M, Cao R, Zhang L, Yang X, Liu J, Xu M, et al. Remdesivir and chloroquine effectively inhibit the recently emerged novel coronavirus (2019-nCoV) in vitro. Cell Res. 2020;30(3):269-71. doi: 10.1038/s41422-020-0282-0.
15. Thoms M, Buschauer R, Ameismeier M, Koepke L, Denk T, Hirschenberger M, et al. Structural basis for translational shutdown and immune evasion by the Nsp1 protein of SARS-CoV-2. Science. 2020;369(6508):1249-55. doi: 10.1126/science.abc8665.
16. Finkel Y, Gluck A, Nachshon A, Winkler R, Fisher T, Rozman B, et al. SARS-CoV-2 uses a multipronged strategy to impede host protein synthesis. Nature. 2021;594(7862):240-5. doi: 10.1038/s41586-021-03610-3.
17. Schubert K, Karousis ED, Jomaa A, Scaiola A, Echeverria B, Gurzeler L-A, et al. SARS-CoV-2 Nsp1 binds the ribosomal mRNA channel to inhibit translation. Nat Struct Mol Biol. 2020;27(10):959-66. doi: 10.1038/s41594-020-0511-8.
18. Zhang Y, Zeng G, Pan H, Li C, Hu Y, Chu K, et al. Safety, tolerability, and immunogenicity of an inactivated SARS-CoV-2 vaccine in healthy adults aged 18–59 years: a randomised, double-blind, placebo-controlled, phase 1/2 clinical trial. Lancet Infect Dis. 2021;21(2):181-92. doi: 10.1016/S1473-3099(20)30843-4.
19. Blanco-Melo D, Nilsson-Payant BE, Liu W-C, Uhl S, Hoagland D, Møller R, et al. Imbalanced Host Response to SARS-CoV-2 Drives Development of COVID-19. Cell. 2020;181(5):1036-1045.e9. doi: 10.1016/j.cell.2020.04.026.
20. Zhang Z, Zheng Y, Niu Z, Zhang B, Wang C, Yao X, et al. SARS-CoV-2 spike protein dictates syncytium-mediated lymphocyte elimination. Cell Death Differ. 2021;28(9):2765-77. doi: 10.1038/s41418-021-00782-3.
21. Ghosh S, Dellibovi-Ragheb TA, Kerviel A, Pak E, Qiu Q, Fisher M, et al. β-Coronaviruses Use Lysosomes for Egress Instead of the Biosynthetic Secretory Pathway. Cell. 2020;183(6):1520-1535.e14. doi: 10.1016/j.cell.2020.10.039.
22. Bastard P, Gervais A, Voyer TL, Rosain J, Philippot Q, Manry J, et al. Autoantibodies neutralizing type I IFNs are present in ~4% of uninfected individuals over 70 years old and account for ~20% of COVID-19 deaths. Sci Immunol [Internet]. American Association for the Advancement of Science; 2021 [citado el 5 septiembre 2021]; doi: 10.1126/sciimmunol.abl4340.
23. Tay MZ, Poh CM, Rénia L, MacAry PA, Ng LFP. The trinity of COVID-19: immunity, inflammation and intervention. Nat Rev Immunol. 2020;1-12. doi: 10.1038/s41577-020-0311-8.
24. Lin G-L, McGinley JP, Drysdale SB, Pollard AJ. Epidemiology and Immune Pathogenesis of Viral Sepsis. Front Immunol. 2018;9. doi: 10.3389/fimmu.2018.02147.
25. McKechnie JL, Blish CA. The Innate Immune System: Fighting on the Front Lines or Fanning the Flames of COVID-19?. Cell Host Microbe. 2020;27(6):863-9. doi: 10.1016/j.chom.2020.05.009.
26. Diehl S, Rincón M. The two faces of IL-6 on Th1/Th2 differentiation. Mol Immunol. 2002;39(9):531-6. doi: 10.1016/S0161-5890(02)00210-9.
27. Mason RJ. Pathogenesis of COVID-19 from a cell biologic perspective. Eur Respir J. 2020; doi: 10.1183/13993003.00607-2020.
28. Ackermann M, Verleden SE, Kuehnel M, Haverich A, Welte T, Laenger F, et al. Pulmonary Vascular Endothelialitis, Thrombosis, and Angiogenesis in Covid-19. N Engl J Med. 2020; doi: 10.1056/NEJMoa2015432.
29. Li H, Liu L, Zhang D, Xu J, Dai H, Tang N, et al. SARS-CoV-2 and viral sepsis: observations and hypotheses. The Lancet. 2020;395(10235):1517-20. doi: 10.1016/S0140-6736(20)30920-X.
30. Varga Z, Flammer AJ, Steiger P, Haberecker M, Andermatt R, Zinkernagel AS, et al. Endothelial cell infection and endotheliitis in COVID-19. The Lancet. 2020;395(10234):1417-8. doi: 10.1016/S0140-6736(20)30937-5.
31. Oronsky B, Larson C, Hammond TC, Oronsky A, Kesari S, Lybeck M, et al. A Review of Persistent Post-COVID Syndrome (PPCS). Clin Rev Allergy Immunol. 2021; doi: 10.1007/s12016-021-08848-3.
32. Lauring AS, Hodcroft EB. Genetic Variants of SARS-CoV-2-What Do They Mean?. JAMA. 2021;325(6):529-31. doi: 10.1001/jama.2020.27124.
33. CDC. Centers for Disease Control and Prevention [Internet]. 2021. SARS-CoV-2 Variant Classifications and Definitions [citado el 15 junio 2021]. Disponible: https://www.cdc.gov/coronavirus/2019-ncov/cases-updates/variant-surveillance/variant-info.html.
34. WHO. [Internet]. 2021. Tracking SARS-CoV-2 variants [citado el 10 junio 2021]. Disponible: https://www.who.int/activities/tracking-SARS-CoV-2-variants.
35. Di Giacomo S, Mercatelli D, Rakhimov A, Giorgi FM. Preliminary report on severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) Spike mutation T478K. J Med Virol. 2021;jmv.27062. doi: 10.1002/jmv.27062.
36. Nelson G, Buzko O, Spilman P, Niazi K, Rabizadeh S, Soon-Shiong P. Molecular dynamic simulation reveals E484K mutation enhances spike RBD-ACE2 affinity and the combination of E484K, K417N and N501Y mutations (501Y.V2 variant) induces conformational change greater than N501Y mutant alone, potentially resulting in an escape mutant. bioRxiv. 2021;2021.01.13.426558. doi: 10.1101/2021.01.13.426558.
37. Jackson CB, Zhang L, Farzan M, Choe H. Functional importance of the D614G mutation in the SARS-CoV-2 spike protein. Biochem Biophys Res Commun. 2021;538:108-15. doi: 10.1016/j.bbrc.2020.11.026.
38. Volz E, Hill V, McCrone JT, Price A, Jorgensen D, O’Toole Á, et al. Evaluating the Effects of SARS-CoV-2 Spike Mutation D614G on Transmissibility and Pathogenicity. Cell. 2021;184(1):64-75.e11. doi: 10.1016/j.cell.2020.11.020.
39. Ali F, Kasry A, Amin M. The new SARS-CoV-2 strain shows a stronger binding affinity to ACE2 due to N501Y mutant. Med Drug Discov. 2021;10:100086. doi: 10.1016/j.medidd.2021.100086.
40. Luan B, Wang H, Huynh T. Molecular Mechanism of the N501Y Mutation for Enhanced Binding between SARS-CoV-2’s Spike Protein and Human ACE2 Receptor. FEBS Lett. 2021;n/a(n/a). doi: 10.1002/1873-3468.14076.
41. Fratev F. The N501Y and K417N mutations in the spike protein of SARS-CoV-2 alter the interactions with both hACE2 and human derived antibody: A Free energy of perturbation study. bioRxiv. 2020;2020.12.23.424283. doi: 10.1101/2020.12.23.424283.
42. Greaney AJ, Loes AN, Crawford KHD, Starr TN, Malone KD, Chu HY, et al. Comprehensive mapping of mutations to the SARS-CoV-2 receptor-binding domain that affect recognition by polyclonal human serum antibodies. bioRxiv. 2021;2020.12.31.425021. doi: 10.1101/2020.12.31.425021.
43. Scheepers C, Everatt J, Amoako DG, Mnguni A, Ismail A, Mahlangu B, et al. The continuous evolution of SARS-CoV-2 in South Africa: a new lineage with rapid accumulation of mutations of concern and global detection. 2021;2021.08.20.21262342. doi: 10.1101/2021.08.20.21262342.
44. Liu Z, VanBlargan LA, Rothlauf PW, Bloyet L-M, Chen RE, Stumpf S, et al. Landscape analysis of escape variants identifies SARS-CoV-2 spike mutations that attenuate monoclonal and serum antibody neutralization. bioRxiv. 2020;2020.11.06.372037. doi: 10.1101/2020.11.06.372037.
45. Tchesnokova V, Kulakesara H, Larson L, Bowers V, Rechkina E, Kisiela D, et al. Acquisition of the L452R mutation in the ACE2-binding interface of Spike protein triggers recent massive expansion of SARS-Cov-2 variants. bioRxiv. Cold Spring Harbor Laboratory; 2021;2021.02.22.432189. doi: 10.1101/2021.02.22.432189.
46. Biswas N, Kumar K, Mallick P, Das S, Kamal IM, Bose S, et al. Structural and Drug Screening Analysis of the Non-structural Proteins of Severe Acute Respiratory Syndrome Coronavirus 2 Virus Extracted From Indian Coronavirus Disease 2019 Patients. Front Genet. Frontiers; 2021;12. doi: 10.3389/fgene.2021.626642.
47. GISAID - NextStrain. [Internet]. 2021. COVID-19 Phylogeny [citado el 18 mayo 2021]. Disponible: https://www.gisaid.org/phylodynamics/global/nextstrain/.
48. Rambaut A, Holmes EC, O’Toole Á, Hill V, McCrone JT, Ruis C, et al. A dynamic nomenclature proposal for SARS-CoV-2 lineages to assist genomic epidemiology. Nat Microbiol. 2020;5(11):1403-7. doi: 10.1038/s41564-020-0770-5.
49. O’Toole Á, Scher E, Underwood A, Jackson B, Hill V, McCrone J, et al. [Internet]. 2021. PANGO lineages [citado el 26 marzo 2021]. Disponible: https://cov-lineages.org/index.html.
50. PHE. GOV.UK [Internet]. 2021. Investigation of SARS-CoV-2 variants of concern: technical briefings [citado el 16 junio 2021]. Disponible: https://www.gov.uk/government/publications/investigation-of-novel-sars-cov-2-variant-variant-of-concern-20201201.
51. Instituto Nacional de Salud (INS). [Internet]. 2021. COVID-19 Secuenciamiento Genético del Virus SARS-COV-2 en Perú [citado el 2 septiembre 2021]. Disponible: http://web.ins.gob.pe/es/covid19/secuenciamiento-sars-cov2.
52. Konings F, Perkins MD, Kuhn JH, Pallen MJ, Alm EJ, Archer BN, et al. SARS-CoV-2 Variants of Interest and Concern naming scheme conducive for global discourse. Nat Microbiol. Nature Publishing Group; 2021;1-3. doi: 10.1038/s41564-021-00932-w.
53. WHO. [Internet]. 2021. Weekly epidemiological update on COVID-19 - 15 June 2021 [citado el 16 junio 2021]. Disponible: https://www.who.int/publications/m/item/weekly-epidemiological-update-on-covid-19---15-june-2021.
54. Davies NG, Abbott S, Barnard RC, Jarvis CI, Kucharski AJ, Munday JD, et al. Estimated transmissibility and impact of SARS-CoV-2 lineage B.1.1.7 in England. Science. American Association for the Advancement of Science. 2021;372(6538). doi: 10.1126/science.abg3055.
55. Tegally H, Wilkinson E, Giovanetti M, Iranzadeh A, Fonseca V, Giandhari J, et al. Emergence and rapid spread of a new severe acute respiratory syndrome-related coronavirus 2 (SARS-CoV-2) lineage with multiple spike mutations in South Africa. Epidemiology. 2020 [citado el 26 marzo 2021]. Disponible: http://medrxiv.org/lookup/doi/10.1101/2020.12.21.20248640.
56. Sabino EC, Buss LF, Carvalho MPS, Prete CA, Crispim MAE, Fraiji NA, et al. Resurgence of COVID-19 in Manaus, Brazil, despite high seroprevalence. The Lancet. 2021;397(10273):452-5. doi: 10.1016/S0140-6736(21)00183-5.
57. Resende PC, Bezerra JF, Vasconcelos RHT de, Arantes I, Appolinario L, Mendonça AC, et al. Virological [Internet]. 2021. Spike E484K mutation in the first SARS-CoV-2 reinfection case confirmed in Brazil, 2020 - SARS-CoV-2 coronavirus / nCoV-2019 Genomic Epidemiology [citado el 26 marzo 2021]. Disponible: https://virological.org/t/spike-e484k-mutation-in-the-first-sars-cov-2-reinfection-case-confirmed-in-brazil-2020/584.
58. Cherian S, Potdar V, Jadhav S, Yadav P, Gupta N, Das M, et al. Convergent evolution of SARS-CoV-2 spike mutations, L452R, E484Q and P681R, in the second wave of COVID-19 in Maharashtra, India. bioRxiv. Cold Spring Harbor Laboratory; 2021;2021.04.22.440932. doi: 10.1101/2021.04.22.440932.
59. Zhang W, Davis BD, Chen SS, Martinez JMS, Plummer JT, Vail E. Emergence of a novel SARS-CoV-2 strain in Southern California, USA. medRxiv. 2021;2021.01.18.21249786. doi: 10.1101/2021.01.18.21249786.
60. WHO. Global Consultation on SARS-CoV-2 Variants of Concern and their Impact on Public Health Interventions. [Internet]. Geneva; 2021 [citado el 10 junio 2021].
61. Casadevall A, Henderson J, Joyner M, Pirofski L. SARS-Cov2 variants and convalescent plasma: reality, fallacies, and opportunities. J Clin Invest. 2021. doi: 10.1172/JCI148832.
62. Khatamzas E, Rehn A, Muenchhoff M, Hellmuth J, Gaitzsch E, Weiglein T, et al. Emergence of multiple SARS-CoV-2 mutations in an immunocompromised host. Infectious Diseases (except HIV/AIDS); 2021 [citado el 26 marzo 2021]. Disponible: http://medrxiv.org/lookup/doi/10.1101/2021.01.10.20248871.
63. Kemp SA, Collier DA, Datir RP, Ferreira IATM, Gayed S, Jahun A, et al. SARS-CoV-2 evolution during treatment of chronic infection. Nature. 2021; doi: 10.1038/s41586-021-03291-y.
64. Harvey RA, Rassen JA, Kabelac CA, Turenne W, Leonard S, Klesh R, et al. Association of SARS-CoV-2 Seropositive Antibody Test With Risk of Future Infection. JAMA Intern Med. 2021; doi: 10.1001/jamainternmed.2021.0366.
65. Andreano E, Piccini G, Licastro D, Casalino L, Johnson NV, Paciello I, et al. SARS-CoV-2 escape in vitro from a highly neutralizing COVID-19 convalescent plasma. bioRxiv. 2020;2020.12.28.424451. doi: 10.1101/2020.12.28.424451.
66. Ritchie H, Mathieu E, Rodés-Guirao L, Appel C, Giattino C, Ortiz-Ospina E, et al. Coronavirus Pandemic (COVID-19). Our World Data [Internet]. 2020 [citado el 4 septiembre 2021]; Disponible: https://ourworldindata.org/covid-vaccinations.
67. Danchin A, Timmis K. SARS-CoV-2 variants: Relevance for symptom granularity, epidemiology, immunity (herd, vaccines), virus origin and containment?. Environ Microbiol. 2020;22(6):2001-6. doi: 10.1111/1462-2920.15053.
68. Zhang L, Jackson CB, Mou H, Ojha A, Peng H, Quinlan BD, et al. SARS-CoV-2 spike-protein D614G mutation increases virion spike density and infectivity. Nat Commun. 2020;11(1):6013. doi: 10.1038/s41467-020-19808-4.
69. Twohig KA, Nyberg T, Zaidi A, Thelwall S, Sinnathamby MA, Aliabadi S, et al. Hospital admission and emergency care attendance risk for SARS-CoV-2 delta (B.1.617.2) compared with alpha (B.1.1.7) variants of concern: a cohort study. Lancet Infect Dis. 2021; doi: 10.1016/S1473-3099(21)00475-8.
70. Guangzhou Center for Disease Control and Prevention. [Internet]. 2021. China’s inactivated vaccines effective against Delta variant: study [citado el 6 septiembre 2021]. Disponible: http://www.news.cn/english/2021-08/23/c_1310144035.htm.
71. Instituto Nacional de Salud (INS). [Internet]. 2021. La variante brasilera tiene una amplia circulación en varios distritos de Lima [citado el 18 mayo 2021]. Disponible: https://web.ins.gob.pe/es/prensa/noticia/ins-variante-brasilera-tiene-una-amplia-circulacion-en-varios-distritos-de-lima/.
72. Romero P, Dávila-Barcklay A, Gonzáles L, Salvatierra G, Cuicapuza D, Solis L, et al. Virological [Internet]. 2021. Novel sublineage within B.1.1.1 currently expanding in Peru and Chile, with a convergent deletion in the ORF1a gene (Δ3675-3677) and a novel deletion in the Spike gene (Δ246-252, G75V, T76I, L452Q, F490S, T859N) - SARS-CoV-2 coronavirus / nCoV-2019 Genomic Epidemiology [citado el 9 mayo 2021]. Disponible: https://virological.org/t/novel-sublineage-within-b-1-1-1-currently-expanding-in-peru-and-chile-with-a-convergent-deletion-in-the-orf1a-gene-3675-3677-and-a-novel-deletion-in-the-spike-gene-246-252-g75v-t76i-l452q-f490s-t859n/685.
73. Johnson & Johnson. Content Lab U.S. [Internet]. 2021. Johnson & Johnson COVID-19 Vaccine Authorized by U.S. FDA For Emergency Use [citado el 18 marzo 2021]. Disponible: https://www.jnj.com/johnson-johnson-covid-19-vaccine-authorized-by-u-s-fda-for-emergency-usefirst-single-shot-vaccine-in-fight-against-global-pandemic.
74. Novavax. A Phase 3, Randomized, Observer-Blinded, Placebo-Controlled Study to Evaluate the Efficacy, Safety, and Immunogenicity of a SARS-CoV-2 Recombinant Spike Protein Nanoparticle Vaccine (SARS-CoV-2 rS) With Matrix-M1TM Adjuvant in Adult Participants ≥ 18 Years [Internet]. clinicaltrials.gov; 2021 [citado el 17 marzo 2021]. Rapport no NCT04611802. Disponible: https://clinicaltrials.gov/ct2/show/NCT04611802.
75. Bernal JL, Andrews N, Gower C, Gallagher E, Simmons R, Thelwall S, et al. Effectiveness of COVID-19 vaccines against the B.1.617.2 variant. medRxiv. 2021;2021.05.22.21257658. doi: 10.1101/2021.05.22.21257658.
76. Altmann DM, Boyton RJ, Beale R. Immunity to SARS-CoV-2 variants of concern. Science. 2021;371(6534):1103-4. doi: 10.1126/science.abg7404.
77. Abdool Karim SS, de Oliveira T. New SARS-CoV-2 Variants — Clinical, Public Health, and Vaccine Implications. N Engl J Med. 2021;0(0):1866-8. doi: 10.1056/NEJMc2100362.
78. FDA. Comirnaty and Pfizer-BioNTech COVID-19 Vaccine. FDA [Internet]. 2021 [citado el 6 septiembre 2021]; Disponible: https://www.fda.gov/emergency-preparedness-and-response/coronavirus-disease-2019-covid-19/comirnaty-and-pfizer-biontech-covid-19-vaccine
79. Connors M, Graham BS, Lane HC, Fauci AS. SARS-CoV-2 Vaccines: Much Accomplished, Much to Learn. Ann Intern Med. 2021;174(5):687-90. doi: 10.7326/M21-0111.
80. Reuters. [Internet]. 2021. Sinopharm, Sinovac COVID-19 vaccine data show efficacy: WHO [citado el 30 mayo 2021]. Disponible: https://mobile.reuters.com/article/amp/idUSKBN2BN1K8?.
Citar como: Bedoya-Sommerkamp M, Medina-Ranilla J, Chau-Rodríguez V, Li-Soldevilla R, Vera Albújar Á, García PJ. Variantes del SARS-CoV-2: epidemiología, fisiopatología y la importancia de las vacunas. Rev Peru Med Exp Salud Publica. 2020;38(3):442-51. doi: https://doi.org/10.17843/rpmesp.2021.383.8734.
Correspondencia: Marcelo Bedoya-Sommerkamp; marcelo.bedoya.s@upch.pe
Recibido: 30/06/2021
Aprobado: 15/09/2021
En línea: 31/09/2021